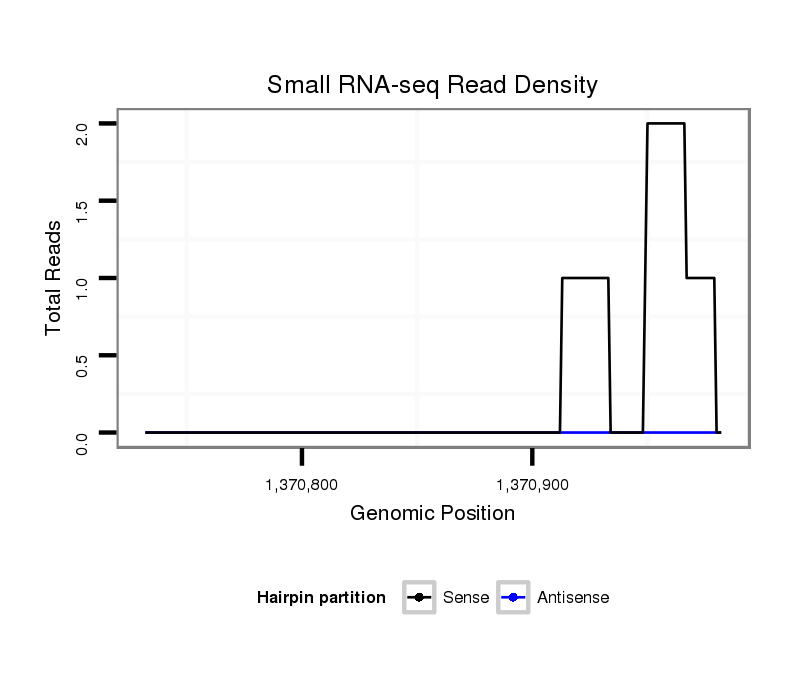
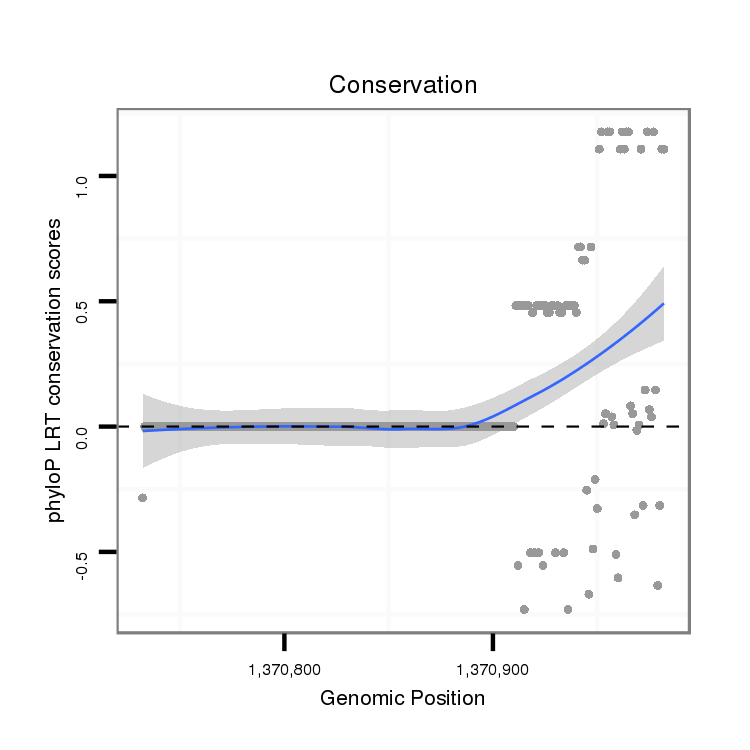
ID:dvi_17807 |
Coordinate:scaffold_13049:1370782-1370932 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_12738:2]; CDS [Dvir\GJ12793-cds]; intron [Dvir\GJ12793-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AAGCTAATTAATCCATTTTCTTGGCATCTCAATGACAAGTACCAGCAGGATTCGTATTCAAAGAAATTGTTATGTCAAATGACACGTTCAATTTGAAATGTTCTCTCGAATCATTCAAAGTGACAACCGAAAGAGAACTCTATGCATATTTTATATTTTACCGCAAATACTTGTGCCTATTAAATAGCTTTTTACGTTCAGGCTTAATCAACGTGTTACCAATAAGACCGACTACATGAGCCAGCTGCGGC **************************************************..(((((((((((((..((((((.....))))))..)))..))))))..(((((((((..(((((...)))))....)))..)))))).....(((((.....(((((......)))))..)))))((((...)))).....)))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060681 Argx9_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR1106718 embryo_6-8h |
V047 embryo |
M027 male body |
SRR060671 9x160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................................................AAATAGCTTTTTACGTTCAGG................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................GTTACCGATAAGATCGACT.................. | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................AACTCTCTGCATATTTTCTAT............................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................TATTAAATACCTTTTTACG....................................................... | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................................................................ACCAATAAGACCGACTAC................ | 18 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................CCAATAAGACCGACTACATGAGCCAGCTGC... | 30 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................CTACCGATAGAGAACTCTAC............................................................................................................ | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................................................................................................AACCGAAAGACACCTCTCT............................................................................................................ | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTCGATTAATTAGGTAAAAGAACCGTAGAGTTACTGTTCATGGTCGTCCTAAGCATAAGTTTCTTTAACAATACAGTTTACTGTGCAAGTTAAACTTTACAAGAGAGCTTAGTAAGTTTCACTGTTGGCTTTCTCTTGAGATACGTATAAAATATAAAATGGCGTTTATGAACACGGATAATTTATCGAAAAATGCAAGTCCGAATTAGTTGCACAATGGTTATTCTGGCTGATGTACTCGGTCGACGCCG
**************************************************..(((((((((((((..((((((.....))))))..)))..))))))..(((((((((..(((((...)))))....)))..)))))).....(((((.....(((((......)))))..)))))((((...)))).....)))).....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060659 Argentina_testes_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
M047 female body |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................................................................................................................................................GCAAGTCCGTATTAGTTGC...................................... | 19 | 1 | 1 | 2.00 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............GGCAAAAGAACCGTAGAGTTA.......................................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................TGGGAACTCTGGCTGATGT............... | 19 | 3 | 18 | 0.17 | 3 | 0 | 0 | 0 | 0 | 1 | 1 | 1 |
| .......................................................................................................................................................................................................................AAGGGTTATTCGGGCTGAT................. | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13049:1370732-1370982 + | dvi_17807 | AAGCTAATTAATCCATTTTCTTGGCATCTCAATGACAAGTACCAGCAGGATTCGTATTCAAAGAAATTGTTATGTCAAATGACACGTTCAATTTGAAATGTTCTCTCGAATCATTCAAAGTGACAACCGAAAGAGAACTCTATGCATATTTTATATTTTACCGCAAATACTTGTGCCTATTAAATAGCTTTTTACGTT----------CAGGCTTAATCAACGTGTTACCAATAAGACCGACTACATGAGCCAGCT--GCGGC |
| droMoj3 | scaffold_6654:2461198-2461280 - | A----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAAACTAACCTCTAACGTTTCTCCATTAATAGGTTGAATCAACGCTTGAACAATAAAACTGACTACATGAGTCAGTT--GAAGC | |
| droGri2 | scaffold_15110:2483401-2483443 + | C--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACGTTTGGCCAAAAAACGTGACTATTCGCGTCAGTT--GAAGC | |
| droSim2 | 3l:15994510-15994544 + | C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAATAAAATGGACTACAACAGCTATTTTGACGGC | |
| droSec2 | scaffold_0:8474683-8474717 + | C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTAAAATGGACTACAACAGCTATTTTGACGGC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 11:31 PM