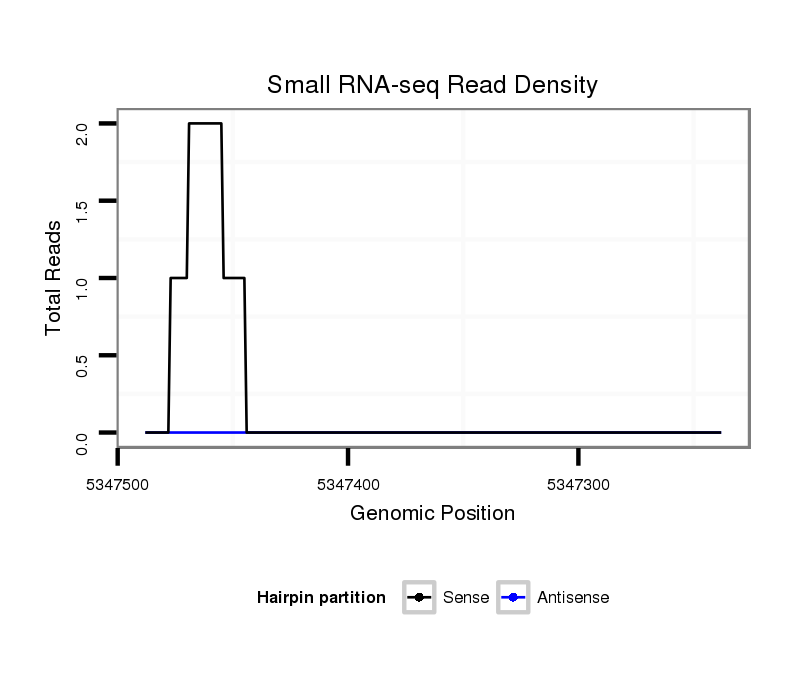
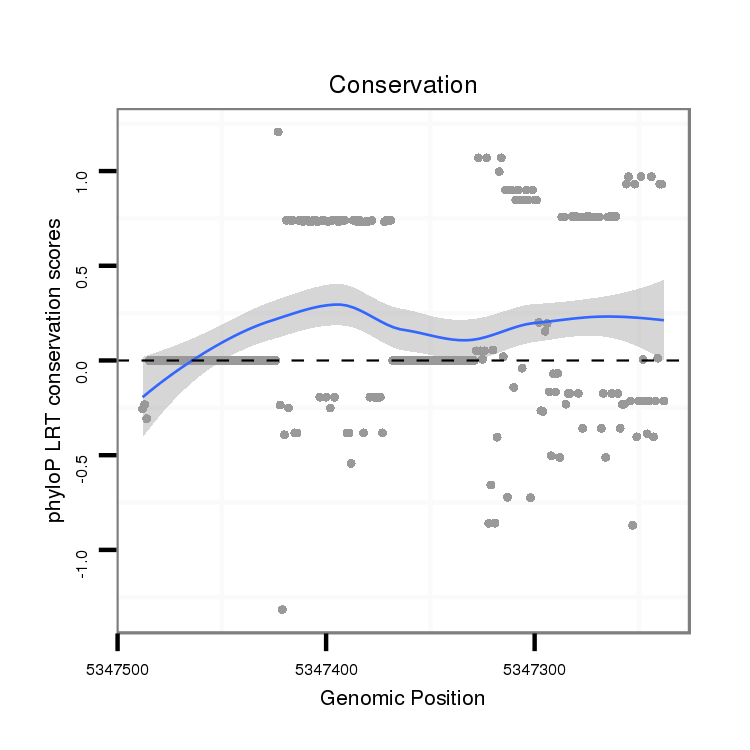
ID:dvi_1757 |
Coordinate:scaffold_12723:5347288-5347438 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17860-cds]; exon [dvir_GLEANR_2435:3]; intron [Dvir\GJ17860-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AACGAACTTAAACTTAACGGCAGCATCGAGCAGGGATCCTGTGCAGCCAGGTAAGCGAATCTGTGTTTAATTTCCAACTTCCAACCTTGCAACTCTTTGGCATGTGTCCAAGAAAGCTTTAAATTAGTCCTTAAATGAAGTTCGTCCTTTTCTCCTAAGTAATCTATCTGCCAAACTTTGTGACTCAAGCTCCCAGCTCGGCCACTATTCCTGATGCGGGATGCTAATCTTAGTCCGAGAACTGTTCACCC **************************************************.((((((......)))))).................((((((...(((((((.(((.........(((.......))).((((...((((........))))...)))).....))).)))))))..))))))....((((...))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060667 160_females_carcasses_total |
V047 embryo |
M028 head |
SRR1106727 larvae |
|---|---|---|---|---|---|---|---|---|---|---|
| ...........ACTTAACGGCAGCATCGAGCAGG......................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...................GCAGCATCGAGCAGGGATCCTGTGC............................................................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................ATCGAGCAGGGATCCTGTTCAGCCAGC........................................................................................................................................................................................................ | 27 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................AGGGCGAGAACTGTTCAC.. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 |
| ................................................................................CCAACCTGGCAACTCT........................................................................................................................................................... | 16 | 1 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 1 |
| ................................................................................................TTGGGATGTGTCCATGGAA........................................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 |
|
TTGCTTGAATTTGAATTGCCGTCGTAGCTCGTCCCTAGGACACGTCGGTCCATTCGCTTAGACACAAATTAAAGGTTGAAGGTTGGAACGTTGAGAAACCGTACACAGGTTCTTTCGAAATTTAATCAGGAATTTACTTCAAGCAGGAAAAGAGGATTCATTAGATAGACGGTTTGAAACACTGAGTTCGAGGGTCGAGCCGGTGATAAGGACTACGCCCTACGATTAGAATCAGGCTCTTGACAAGTGGG
**************************************************.((((((......)))))).................((((((...(((((((.(((.........(((.......))).((((...((((........))))...)))).....))).)))))))..))))))....((((...))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
SRR060674 9x140_ovaries_total |
SRR060677 Argx9_ovaries_total |
V053 head |
SRR060659 Argentina_testes_total |
M027 male body |
SRR060656 9x160_ovaries_total |
SRR060679 140x9_testes_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................................................GGTCGAGCTGGTGATAAGGACTACGT................................. | 26 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................CGAGCTGGTGATAAGGACTACGTCT............................... | 25 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................TCGTCCCTAGGACACGTCGGTCCATA..................................................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................CTTTCGAAATGTAATCATGAAAT..................................................................................................................... | 23 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TTACTTCAAGCTGGAGAGGA.................................................................................................. | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................GGTAGAAAGTTGGAACGTGG.............................................................................................................................................................. | 20 | 3 | 12 | 0.25 | 3 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................GGTAGAAAGTTGGAACGT................................................................................................................................................................ | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................TCGTGCCGATGATAAGGAAT..................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................TAGTCAGGAAGGTACTTCA.............................................................................................................. | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................TGGTAGAAAGTTGGAACGT................................................................................................................................................................ | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ........................................................................................................................................................................................TGTTCGAGCGTCGAGTCGGT............................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................CAGGAAAAGAGGACAGATT......................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:5347238-5347488 - | dvi_1757 | AACGAACTTAAACTTAACGGCAGCATCGAGCAGGGATCCTGTGCAGCCAGGTAAGCGAATCTGTGTTTAATTTCCAACTTCCAACCTTGCAACTCTTTGGCATGTGTCCAAGAAAGCTTTAAATTAGTCCTTAAATGAAGTTCGTCCTTTTCTCCTAAGTAATCTATCTGCCAAACTTTGTGACTCAAGCTCCCAGCTCGGCCACTATTCCTGATGCGGGATGCTAATCTTAGTCCGAG-AACTGTTCACCC |
| droMoj3 | scaffold_6473:14154516-14154532 - | AAC-------------------------------------------------------------------------------------------------------------------------------------------------------------GACCCAAAATTCAA------------------------------------------------------------------------------ | |
| droGri2 | scaffold_15116:973569-973611 - | AGC-------------------------------------------------------------------------------------------------------------------------------------------------------------AATATACCTTTCAGATTTTGTGACTCGAGCTTTCAAATTG---------------------------------------------------- | |
| droWil2 | scf2_1100000004949:1012002-1012039 - | AGA-------------------------------------------------------------------------------------------------------------------------------------------------------------AATCTATATACCAAAATTGGTGGCTCTAGCTCTTA--------------------------------------------------------- | |
| droRho1 | scf7180000763660:1650-1654 - | CAC--------------------------------------------------------------TC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415878:241993-242050 + | GTT--------------------------------------------------------------TCCAAATTAAAACTTCCAACTTTACTATTCTTTTTGATGTGGCCGAAGGGTCTTT------------------------------------------------------------------------------------------------------------------------------------ | |
| droEug1 | scf7180000407353:785-811 + | AAT--------------------------------------------------------------TTTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------GTTCGAAAATTCATTTGCCT | |
| droYak3 | 3L:21556275-21556342 - | GAC--------------------------------------------------------------TCGT-------------------------------------------------------------------------------------------------------------------------CTCCTGCGCTCCCTTCATTTCGGATGCGGGCCCCTGATTGATGTCGGGG-ATCTGTCCGCCC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 09:34 PM