
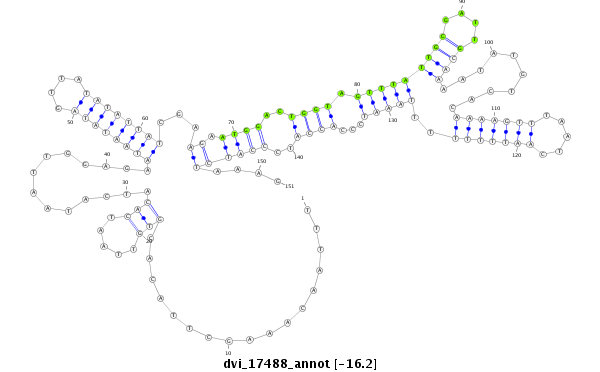
ID:dvi_17488 |
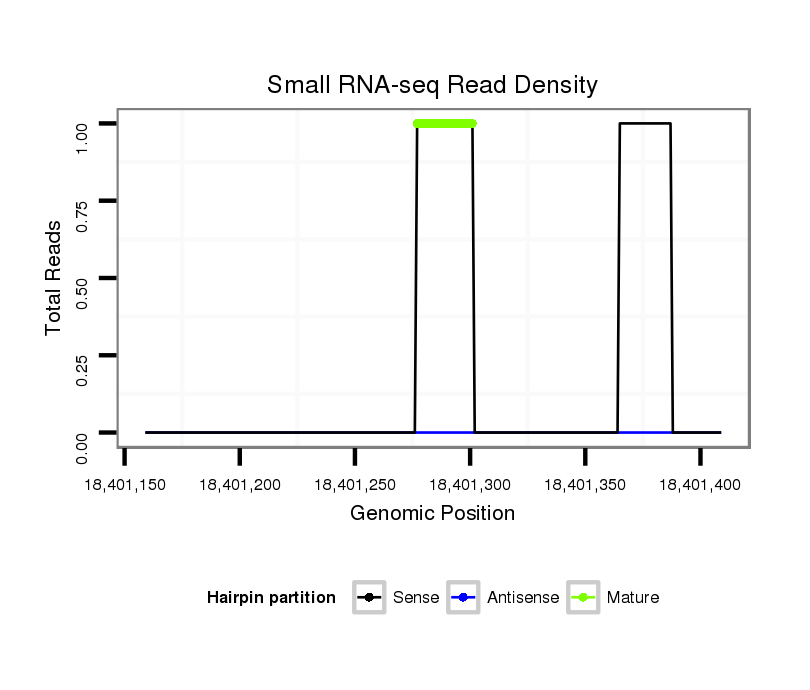
Coordinate:scaffold_13047:18401209-18401359 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -16.2 | -16.0 | -16.0 |
 |
 |
 |
exon [dvir_GLEANR_9905:2]; CDS [Dvir\GJ24640-cds]; intron [Dvir\GJ24640-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTTTCCGTTCATAAGCTTAACTTAATAAAGTTGTAAAAAATGTTGCTAATTTTAACAAAGCTTACACGTGTTAATCACATCATAATTGGAGAATAATATAGTTATATATTATCGAAGAATGGACTGGTAGTTTATTGCGATTGCAAAATATGTCACAAAAGTTTAATCAATTTTTTTTAAATCCCACCATCCCATCTAAAGTGGACAATGCCTCAGAAAATGCAGCCGAGTTACCCTGCGGCAACGTGGAT **************************************************.................(((.....)))..............((((((((....))))))))...(((.(((..((((.(((((((((....))))..........(((((((.....)))))))..)))))...))))..))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
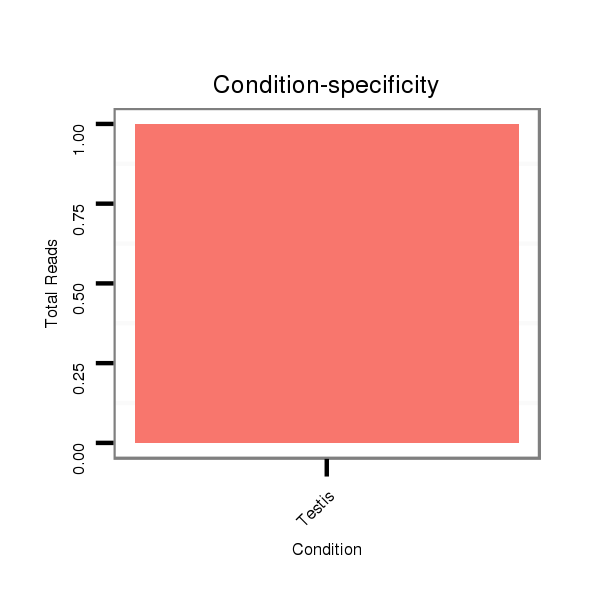
SRR060678 9x140_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060660 Argentina_ovaries_total |
SRR060679 140x9_testes_total |
M028 head |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................................AGTCGAGTGACCCTGCGCC......... | 19 | 3 | 13 | 3.85 | 50 | 9 | 10 | 0 | 10 | 9 | 7 | 3 | 0 | 1 | 1 | 0 | 0 |
| ......................................................................................................................ATGGACTGGTAGTTTATTGCGATTG............................................................................................................ | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................AATGCCTCAGAAAATGCAGCCGA...................... | 23 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................AGTCGAGTGACCCTGCGC.......... | 18 | 3 | 20 | 0.55 | 11 | 0 | 10 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AAGAAAGTGCTGGTAGTTTA..................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................CAGTCGAGTGACCCTGCGC.......... | 19 | 3 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................AGTCGAGTCACCCTGCGCC......... | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................ATATTATCTAAGACTAGACT.............................................................................................................................. | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................................................................ATTATAATTGGAGGATAA........................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .........................................................................................................................................................CACAAACGTTTAATCGATAT.............................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GAAAGGCAAGTATTCGAATTGAATTATTTCAACATTTTTTACAACGATTAAAATTGTTTCGAATGTGCACAATTAGTGTAGTATTAACCTCTTATTATATCAATATATAATAGCTTCTTACCTGACCATCAAATAACGCTAACGTTTTATACAGTGTTTTCAAATTAGTTAAAAAAAATTTAGGGTGGTAGGGTAGATTTCACCTGTTACGGAGTCTTTTACGTCGGCTCAATGGGACGCCGTTGCACCTA
**************************************************.................(((.....)))..............((((((((....))))))))...(((.(((..((((.(((((((((....))))..........(((((((.....)))))))..)))))...))))..))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060686 Argx9_0-2h_embryos_total |
V116 male body |
SRR060665 9_females_carcasses_total |
SRR060677 Argx9_ovaries_total |
SRR060669 160x9_females_carcasses_total |
SRR060663 160_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060673 9_ovaries_total |
SRR060674 9x140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................CAGTGTTTTCAAATCAGGTA................................................................................ | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................TGGGGTGGTAGGGTACAT..................................................... | 18 | 2 | 3 | 0.67 | 2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................TGGGGTGGTAGGGTACATTC................................................... | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................AAAAAAATTTAGGTTGGAAGAGT......................................................... | 23 | 3 | 4 | 0.50 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................ATTGAGGGTGGTGGGGTAG....................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................GGGTGGTAGGGTACATTCA.................................................. | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........GTATTGGAATTGAAGTAT................................................................................................................................................................................................................................ | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................................CAGGGGGGTAGCGTAGATT.................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........AGTATTGGAATTGAAGTA................................................................................................................................................................................................................................. | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................................................ATTTAGGGGGGTTGGGTAT....................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
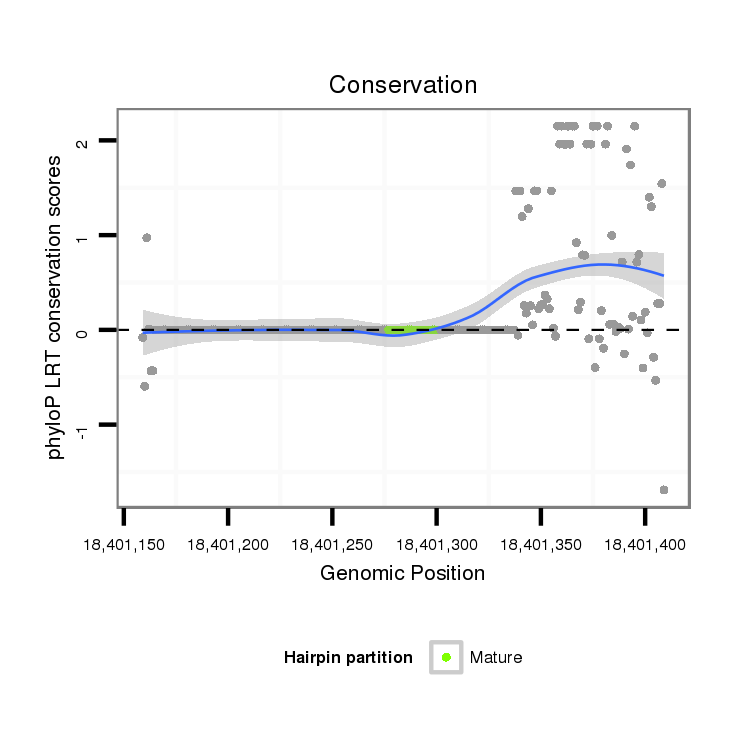
| droVir3 | scaffold_13047:18401159-18401409 + | dvi_17488 | C-------------------------------------TTTCCGTTCATAAGCTTAACTTAATAAAGTTGTAAAAAATGTTGCTAATTTTAACAAAGCTTACACGTGTTAATCACATCATAATTGGAGAATAATATAGTTATATATTATCGAAGAATGGACTGGTAGTTTATTGCGATTGCAAAATATGTCACAAAAGTTTAATCAATTTTTTTTAAATCCCACCATCCCATCTAAAGTGGACAATGCCTCAGAAAATGCA----GCCGAGTTACCCTGCGGCA-----ACGTGGAT |
| droMoj3 | scaffold_6540:13098738-13098804 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAT------ATTTTATTTATAGTGGACAACACCTCGGAAAATGCA----ACTCAGTCAACTTGCGGCA-----ACATAAAC | |
| droGri2 | scaffold_15074:6299991-6300045 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGGACAATTCCTCGGAGAATACA----GCCGAGTTACCTTGTGT-T-GTTGACAAGGAG | |
| droWil2 | scf2_1100000004902:6530057-6530111 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGGACAATGCCACGGAGAAT-CACGTTGCCGCT-----TTGCCTCAGGTACACAAGGAG | |
| droAna3 | scaffold_13340:19890599-19890640 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGGACAATGCCACCGACAGC-CACGTT--------AGCCTGCCATTGGT---------- | |
| droBip1 | scf7180000396413:2501809-2501850 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGGACAATGCCACCGACAGC-CACGTT--------AGCCTGCCACTGGT---------- | |
| droFic1 | scf7180000453800:1815132-1815199 - | T-------------------------------------TTCCC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCTAATGATTCCTCTTACAGTGGACAATGCCACGGAGACC-CACGAT--------AGCCTGCCACTGGT---------- | |
| droEle1 | scf7180000486474:699508-699580 - | A-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCTAATGATTCCTCTTGCAGTGGACAATGTCACAGAGACG-CACGAG--------AGCCTGCCGTTGGTACACTCGGAT | |
| droBia1 | scf7180000302075:5199097-5199163 - | A-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATCTAATGATTCCTCTTGCAGTGGACAATGCCACGGAAACC-CACGAT--------AGCCTACAGCAAGTAC------AC | |
| droTak1 | scf7180000415765:1278688-1278748 + | A-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCTAATGATTCCTCTTGCAGTGGACAATGCAACGGAAAGC-CACGAT--------AGCCTACAGCTG------------ | |
| droEre2 | scaffold_4820:8402871-8402974 + | AAATATACCTCCAAATTGAAGTTAGCCTTGCTTGAAGTGTCAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTCTAATGATTCCTCTTGCAGTGGACAATGTCACGGAGACC-CACGAT--------AGCCTGCCGCTGG----------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:57 AM