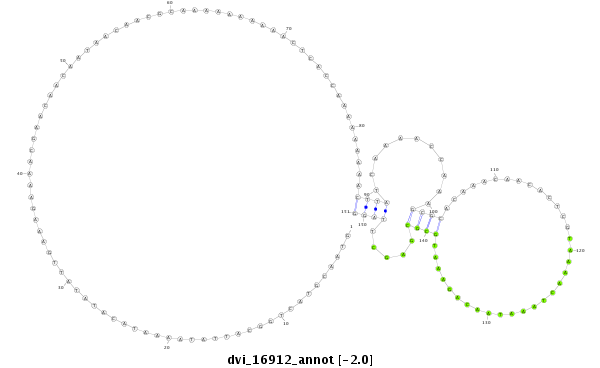


ID:dvi_16912 |
Coordinate:scaffold_13047:14015915-14016065 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
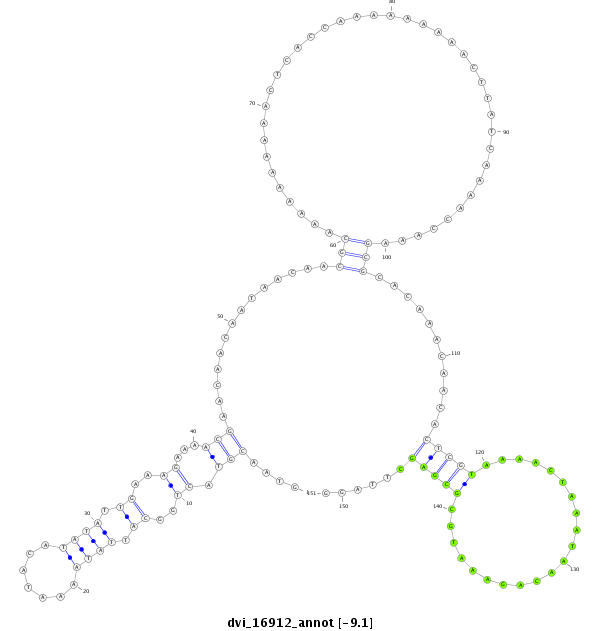
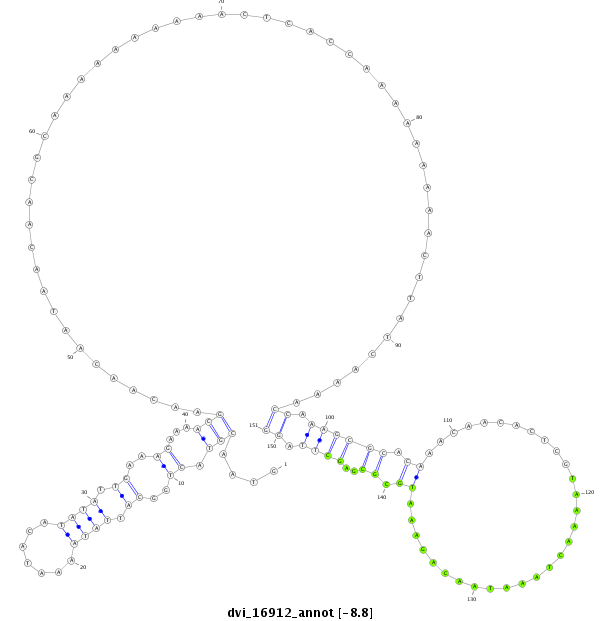
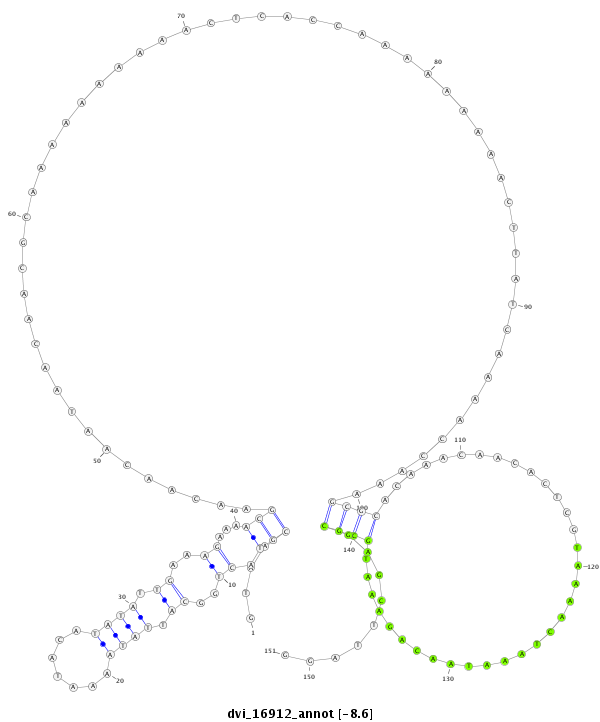
| -9.34 | -9.09 | -8.8 | -8.57 |
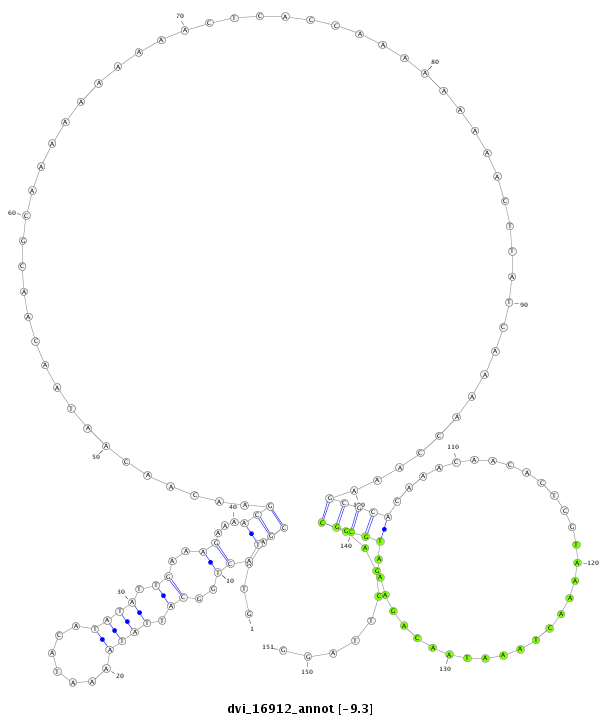 |
 |
 |
 |
CDS [Dvir\GJ24414-cds]; exon [dvir_GLEANR_9692:1]; intron [Dvir\GJ24414-in]
No Repeatable elements found
| ------------------------##########################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GTGAGAGAGGAGTCGATACAAGTGATGTTCAAGTTATTGAAACAAAAAGTGTAACGTACTGGCATTATAAAATACATATATTGAAAGAAAACGAACAACAATAACAACGCAAAAAAAAAACTCACCAAAAAAAAACTTATCAAAACCAAAGCGCACAAACAACACTCGTAAAACTAAATAACAGAAATGCGCGAGCTTAGGGAGCTGCGACAGCAGCGTCAGGCGTAAGTACAATATGTGAGCGGGGTGAA **************************************************.....................................................................................((((...........((((..................................)))).....))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060674 9x140_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060680 9xArg_testes_total |
V116 male body |
SRR060682 9x140_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060665 9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................TAAAACTAAATAACAGAAATGCGCGAGC....................................................... | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................AGTGATGTTCAAGTTATTGAAACA............................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................ATTGAAATGCACGAGCTTAGGGA................................................ | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................AGATATTGAAACAAAATGTGT....................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................AATGAGCGAGCTTAAGGAG............................................... | 19 | 2 | 3 | 0.67 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| .........................................ACAAAAGGCGTAACGTTCTG.............................................................................................................................................................................................. | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................AAAGGAACAGCAATAACAACGC............................................................................................................................................. | 22 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................................AAACTTATCACGACCAAAACG.................................................................................................. | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................AAGTTCTTGAAACAAAAA........................................................................................................................................................................................................... | 18 | 1 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................AAACAACTCACCAATAAGAAA.................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................AGGTGCGACAGCAGGGTT............................... | 18 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CACTCTCTCCTCAGCTATGTTCACTACAAGTTCAATAACTTTGTTTTTCACATTGCATGACCGTAATATTTTATGTATATAACTTTCTTTTGCTTGTTGTTATTGTTGCGTTTTTTTTTTGAGTGGTTTTTTTTTGAATAGTTTTGGTTTCGCGTGTTTGTTGTGAGCATTTTGATTTATTGTCTTTACGCGCTCGAATCCCTCGACGCTGTCGTCGCAGTCCGCATTCATGTTATACACTCGCCCCACTT
**************************************************.....................................................................................((((...........((((..................................)))).....))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060666 160_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060658 140_ovaries_total |
M047 female body |
SRR060670 9_testes_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................AAATTGCATGACCGTAATATATT................................................................................................................................................................................... | 23 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................TCGACCCTGTCGTCGCAATC............................. | 20 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TTGAGTAGTTTTGATTGCGCGT................................................................................................ | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................CGCTATTTTTTTGAGTGGTT........................................................................................................................... | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................ACATTGCCGAACCGTAATAT...................................................................................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................CATTGCCGAACCGTAATATT..................................................................................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................................................................GTTGTTTTGTTGGGTGGTTTTTT....................................................................................................................... | 23 | 3 | 20 | 0.10 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................GTTGGTTGTGACCATTTTGATT.......................................................................... | 22 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................................................................................................GTTGGTTGTGACCATTTTGAT........................................................................... | 21 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................TTAGTTATATAACTTTCCTTT................................................................................................................................................................ | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:14015865-14016115 + | dvi_16912 | GTGAGA--------------GAGGAGT---CGATA--------------------CAAGTGATGTTCAAGTTATTGAAACAAAAAGTGTAACGTACTGGCATTATAAAAT------ACATATATT-----------GAAAGAAA-----ACGAAC-A-----ACAATAA-CAACGCAAAAAAAAAACTCACCAAAAAAAAACTTATCAAAACC------------------------------AAAGCGCAC-----AAACA-ACACTCGTAAA------------------ACTA--------------AATAACAGAAATGCGCGAGCTTAGGGAGCTGCGACAGCAGCGTCAGGCGTAAGTACA---ATATGTG----------------------------AGCGGGGTGAA |
| droMoj3 | scaffold_6540:26071134-26071430 - | GGGAGCGAGTGCAAGACAATG----------------------------------TGGATAACGTGGAAGTTATTGAAACAAAAAGTGTAACGTACTGTCCTTATAATTGATATTAACC--CAAA-----------CAAACAAAAAACAAGAAGCAAAACTTA---C-AGCTACGCAAAAA-----GTCAACAA----ATATTTATCGTAACC------------------------------AA----CAC---------AAACACTCGCAAAACAAAACAAAACAAAAAAA---------ACAACAAA-AACACCGAAATGCGCGAGCTTTGTGAACTGCTACA---GTGCCAGGCGTAAGTAAA---ATATGTGTGAGAGAGAGAGAGAGAGAGTAAAAGAGAGAGGGGTAAA | |
| droGri2 | scaffold_14830:5939203-5939476 - | GAGGGAGGGAGAAAGAGAGAGAGGACAAAACGCAT--------------------CACGTTATGTTCAAGGCATTAAAGGAAAAACTGTAACGTACTGGCGAAATAACTA------ACA--TTTA-----------GAAAATTC-----ACTAAT-A-----ACAAAAACCTACGCAAAAA------CCAACAA----CAATTTATCAAAA------------------------------------CACAC-----AAACAAATACTCGTAA----------------------------A-----AGCAAAAAAAAAAATGCGCGAGCTTAGGGAGCAGCGACA---ACGTCAGGCGTAAGTACTACAATATATATGAGAGAAGGACAGTGTGATAGAAAGAGAGAG-----AA | |
| droWil2 | scf2_1100000004943:12549115-12549345 + | dwi_3452 | TCAAGAGCATC------------------------------------AGGAGACAATGGTTTTCATAAGGCTGCTAAAAAGCAAACTGTAATGTATAGAAATAC------------ACT--TTTGGCGACGAACTACAACTACA-----ACAAAACAACGCTAC----ACATCAGCAACAA--------------------------------CAACAACAACGAAAACAACACCACCACCACCAACAACAA------------------------------------CAACA---------ACAACAACAACAGCAACAATGCGTGAGCTTAGGA------------------AGACGTAAGTACT---ATAGGGG----------------------------AGTAGAGAAAA |
| droAna3 | scaffold_13340:14770901-14771144 + | dan_3099 | GGAAGA--------------GAAGCCACCATCCACCACCATCGCTGCTCAAGTC-CCGGCCATGTTCAAGTTTTTGAAAAGCAAACTGTAACGTATTTAAAAAAAA----------------------AAGGCCTACAAC----AAACAAAAAAAAAACTATACAAAGTTATCCGCAAAAA---AACTAAAAAAAAAAAAA--C-------------------------------ATCAAAACAAAGCGTGACAAAAAAAAAAA----------------------------ACTACGTTTC-----TGCGACAAAAACAATGCGCGAATTTG---------------------AGGCGTAAGTAT--------GC----------------------------GAGTG-----GG |
| droBip1 | scf7180000395971:411418-411609 - | GGAAGA--------------GAAGCCACCA----------CCGCTGCTCAAGTC-CCGGCCATGTTCAAGTTTTTGAAAAGCAAACTGTAACGTATTTTAAAAA------------------------AAGCCCTA--------AAACAAAAAAAAAACTATACAAAGTTATCCGCGAAAA----------------AAAA--C-------------------------------ACCAAAACAAAGCGTGA-----CAGAAAA----------------------------ACTACGTTTC-----TGCGACAAAAACAATGCGCGAATTTGAGG-------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302461:1848762-1848819 - | AGGAACTCG------------------------------------GCGGGCGGC-ATGGTCTTCTTCAAGTTGCTGAAGAGCAAACTGTAACGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491280:4574079-4574260 - | G---------------------------------------------------------GTTTTCTTCAAGTTCTTGAAGAGCAAACTGTAACGTATATATGTAGATATAC------ACA--CATA-----------TAAACACA-----ACAGATCA-----ACGTAAACATACGCGAAAA--------------------------------AGATACGAGAGAAAACAAAAAAGA----------TACAA-----AGTAAGATACTATTCA----------------------------ACCCACGAG-AACAACAATATGCGCGAAATT---------------------CAGGCGTAAGTA--------------------------------------------------- | |
| dm3 | chr3R:3118811-3118860 + | GAGAGCTGGCC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTGGCGCCAGCCG---------------------------GAAAGTTCAGCTAATGAACAGTG-----GC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
Generated: 05/17/2015 at 04:30 AM