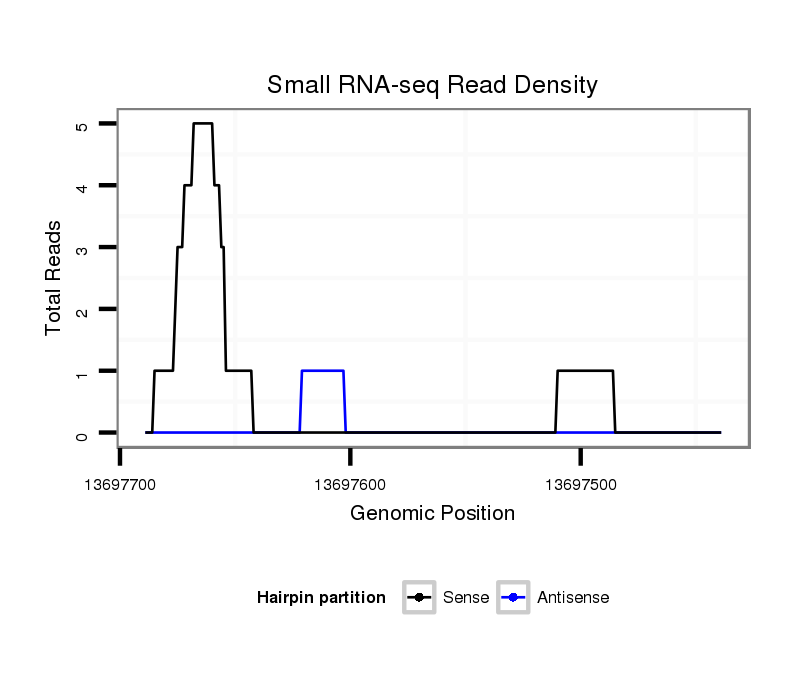
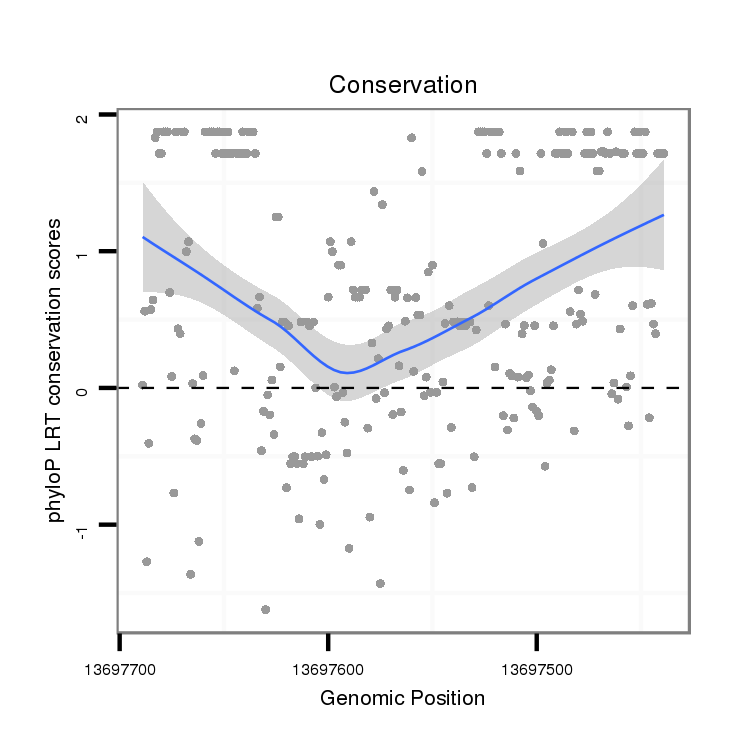
ID:dvi_16848 |
Coordinate:scaffold_13047:13697489-13697639 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ22862-cds]; exon [dvir_GLEANR_8228:1]; intron [Dvir\GJ22862-in]
No Repeatable elements found
| ---------------------------------#################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CATCATAAGCAATTCTAGAAACACAGGCGCAAAATGTTCTCACGACCCAGGTAAGCTAATAAACCGAAACCAACAGAAGTCGAATCCCCCTGGCATCAAGTTGCCAAAGGAATTCGCAATGCTTGCAACTATTTTTGGTACAAACGCGAATTGACGAACTGTTATGCAATTTCAATTGTCAGGCCACGGTTCCGCGTCCCAGCTGTTTTTGTCAGTGACCTAGTCTTTTTCGTGGCAGACGAGTGTGCGCC **************************************************............(((((((.......(((((((((.((..(((((......)))))..)).)))))....))))........)))))))....(((((.......(((((((..((((((....)))).))....))))))))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M047 female body |
SRR060668 160x9_males_carcasses_total |
M061 embryo |
SRR060659 Argentina_testes_total |
SRR060671 9x160_males_carcasses_total |
V053 head |
SRR060667 160_females_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................CACAGGCGCAAAATGTTCTCACGACC............................................................................................................................................................................................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................TGTTCTCACGACCCAGCTT...................................................................................................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....ATAAGCAATTCTAGAAACACAGGCGC............................................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............CTAGAAACACAGGCGCAAA.......................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................CAGGCGCAAAATGTTCTCACGACCCAGCTT...................................................................................................................................................................................................... | 30 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............TCTAGAAACACAGGCGCAAAAT........................................................................................................................................................................................................................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AAATGTTCTCACGACCCAGCT....................................................................................................................................................................................................... | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................CAGGCCACGGTTCCGCGTCCCAGCT............................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................GAAACACAGGCGCAAAAT........................................................................................................................................................................................................................ | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................CGCGTCCCAGCTGATGTTGGC...................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ......................................................................GAACAGACGTAGAATCCCC.................................................................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................TCGAATCCTCATGGCACCA......................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................AATGCGCCTGGCATGAAGTT..................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................................................TTACGCGAAATGACGAACT........................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
GTAGTATTCGTTAAGATCTTTGTGTCCGCGTTTTACAAGAGTGCTGGGTCCATTCGATTATTTGGCTTTGGTTGTCTTCAGCTTAGGGGGACCGTAGTTCAACGGTTTCCTTAAGCGTTACGAACGTTGATAAAAACCATGTTTGCGCTTAACTGCTTGACAATACGTTAAAGTTAACAGTCCGGTGCCAAGGCGCAGGGTCGACAAAAACAGTCACTGGATCAGAAAAAGCACCGTCTGCTCACACGCGG
**************************************************............(((((((.......(((((((((.((..(((((......)))))..)).)))))....))))........)))))))....(((((.......(((((((..((((((....)))).))....))))))))))))....************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
V116 male body |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060655 9x160_testes_total |
SRR060683 160_testes_total |
M027 male body |
SRR060665 9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060658 140_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................TGGTTGTCTTCAGCTTAGG.................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................GCTATGGTTGGCTTCAGATTA...................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................TTCAGCTTAGTGGGACCGTGT.......................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................TTTGGCTTTGGTTGTCTGAA........................................................................................................................................................................... | 20 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................CTATGGTTGGCTTCAGATTA...................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................TGCCAAGACGCCGGGTCTAC.............................................. | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................TCAGCGTTGGGGGACCATA........................................................................................................................................................... | 19 | 3 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................CTTGCCAAGACGTGAAAGTT............................................................................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................TGGCTTTGGTTGTCTGAA........................................................................................................................................................................... | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..................................................................TATGGTTGGCTTCAGATTA...................................................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
| ..............................................................................CAGCTTAGGGGCAACGGAG.......................................................................................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................TACAAGAGTCCTGGTTACA....................................................................................................................................................................................................... | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................TGGGTTGTCTTCAGATTA...................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:13697439-13697689 - | dvi_16848 | CAT-CATAAGCAATTCTAGAAA-----------------CACAGGCGCAAAATGTTCTCACGACCCAGGTAAGCTAATAAAC-CGAAACCAACAGAAGTCGAATCCCCC-------------------------------------TGGCATCAAGTTGCCAAAGGAATTC--G-------------------C--------------A----ATGCTTGC--AACTATTTT-TG------GTACAAACGCGAATTGACGAACTGTTATGCAA---TTTCA-ATTGTCA-------------------GGCCACGGTTC---CGCGTCCCAGCTGTTTTTG-TCAGTGACCTAGTCTT----T---------TTC-GTGGCAGACGAGTGTGCGCC |
| droMoj3 | scaffold_6540:10926057-10926332 - | ATA-AAAAAGCAATTCAAGAAA-CACAAAATTCTAGAAGCAGAAACGCAAAATGTTCTCACGGCCCAGGTAAGCTACGTCAA-CGAAAACTGTTCATATCAA-GAAACCAGGTGGGCAT---------------------------TGGCATCGTATTGCCAAAGAAATTG--G-------------------A--------------A----ATGCTTGT--AGCTATTTT-TG------GTAAATTTGCAAATTGACGAAACGTTATGCAA---TTTCA-ACTGTCA-------------------GCCATTCGGCC---CGCGTCCCAGCTGTTCTTC-TCAGTGACCTAGTCGT----T---------GCC-GTGGTAGACGAGCGTGCGCC | |
| droGri2 | scaffold_14830:6253630-6253842 + | CATCAAAAAGCAATTCTACGAA-----------------CATAAGCTCAAAATGTTCTCACGACCCAGGTAAGCTTTCAA-----------------------TTCAGCTGGCAAGCCT---------------------------TGTC-----CTTGCCAAATTAATTTTTG----------------------------------C----ATACTTGC--AAATATTTT-TG---------------TGT-----------ATTATGCAA---TTTCCGACCGACTGTT------------------CCAC--ATC---CACATCCCAGCTGTTGTTCGTCAGTGT----GTC--------TAA-----TTC-GCCGCAGACGAGTGTGCGCC | |
| droWil2 | scf2_1100000004902:5373922-5374211 + | CAT-TAAAAGCAATTC-ACGAAGAA--GCATACAAAAAGCAGC-AGGCAAAATGTTCTCACGGCCCAGGTAAGTAGCGTTTC-CGA-----------------------AGAAACGCCGAGCCAGCAACAATCGGTAATAAGGGGTTGGGATTGTAT--------AAAAGG--G-------------------A------------------------TAT--GAATTTCAT-CATTGTCAGCATG--TAAGC-----------ATTATGCAACGAATTCTGCATGACAGACATACAGAAATACATACGGACA-TGGTCTGG-----GCCAGCTGTTTG-C-CCAGTGACCTAGTCGCAAATTCGGAGACTCCTCGGGGCCAGACGAGAGGCCGCC | |
| droFic1 | scf7180000453776:152354-152553 + | TAC-GGAAAGCAATAAAACGAA-----------------------TTAAAAATGTTCTCACGACCCAGGTAAGCTTT------------------------------------------------------------------------------------------------------TTGCAAGGACTC-GCGGCCAGGATG--TGAGTGTG----TGT--GAGTGCTT---GGGGG---------CGGGA-----------ATTATGCAAC-ATTTCG-CGATTCG-------------------GGACGAGGAGCTGC-----CCCAGCTGGTAG-C-CCAGTGACCTAGTCAT----TCGGA-----TTC-GGATCAGACGAGTAGCCGCC | |
| droEle1 | scf7180000491280:1023340-1023555 + | CAACGGAAAGCAATTCTACGAA-----------------------GTGAAAATGTTCTCACGACCCAGGTAAGCTTTTTT-------------------------------------------------------------------------------------------------TTTTGCAAGGACTTCGCGGTCAGGATGTGTGAGTGAG----TAC--GAGTGTTTT-CGGGGG---------CGGGA-----------ATTATGCAAC-ATTTCG-CGACTCG-------------------GGACGAGGAACTGCCCAGCCCCAGCTGGTAG-C-CCAGTGACCTAGTCAT----TCCGT-----TTC-GGATCAGACGAATAGCCGCC | |
| droEug1 | scf7180000409770:441529-441734 + | CAGCGGAAAGCAATTATACGAA-----------------------CCAAAAATGTTCTCACGACCCAGGTAAGCTTTT---------------------------------------------------------------------------------------------------TTCTGTAAGGACTTTTCGAGCAGGATGTGTGAGTGTG----AACGTGAATGTTTTTTTGGGG---------CGGGA-----------ATTATGCAAC-ATTTCG-CGATTCG-------------------GGACGAGGAACTTC-----CCCAGCTGGTAG-C-CCAGTGACCTAGTCAT----T-----------C-GGTTCAGACGAGTAGCCGCC | |
| droYak3 | 3R:23516855-23517055 - | --------AGCAATTAAACGAA-----------------------ATAAAAATGTTCTCACGACCCAGGTAAGCTTTTTTTTTCGC--------------------------------------------------------------------------------GACTTTCGGTTTTTTGCAAGGACTC-TCG--AAGGATTTGTTAGTGTG----TGT--GA-----------AGG---------CGCGC-----------ATTATGTAAG-AATTCG-CGATGCG-------------------GGAGGAGCAACTGC-----CCCAGCTGGTAG-C-CCAGTGACCTAGTCAC----T-----------C-GGATCAGACGAGCAGCCGCC | |
| droEre2 | scaffold_4820:5059703-5059904 - | -----GAAAGCAATTATACGAA-----------------------ATAAAAATGTTCTCACGACCCAGGTAAGCTATATTTT-CGA----------------------------------------------------------------------------------------GTTTTTTGCAAGGACTCGCCTCCAAGGATTTGTTAGTGTG----CGT--G--TGT-----GAGGG---------CGCGA-----------ATTATGTAAG-ATTTCG-CGATGCG-------------------GGAGGAGCAACTGC-----CCCAGCTGGTAG-C-CCAGTGACCTAGTCAC----T-----------C-GGATCAGACGAGCAGCCGCC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:29 AM