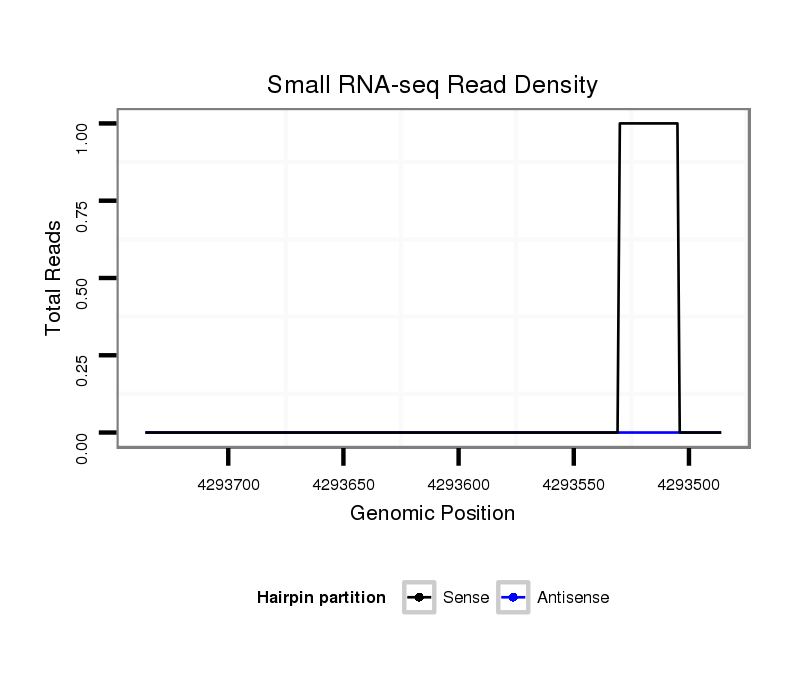
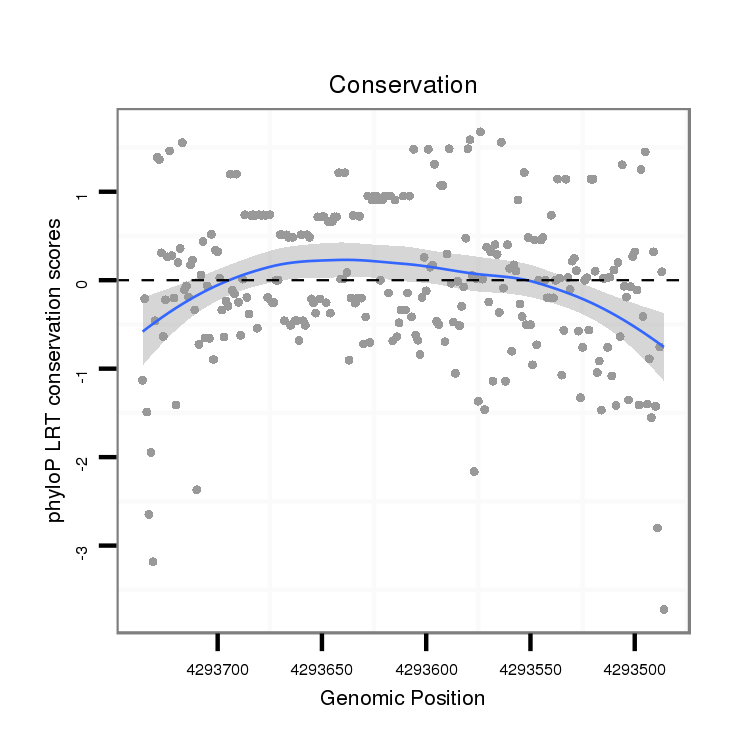
ID:dvi_1650 |
Coordinate:scaffold_12723:4293536-4293686 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17908-cds]; exon [dvir_GLEANR_2479:1]; intron [Dvir\GJ17908-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTCTTCCACGTCCACCCTAAAAGAAGGCAACAAACAGAACTCTGGAGAAAGTGAGGACTCCATTGCATCAGACGACATATCAACTAAAACGGATTCCTTAAATACGCCAGCTTCGGTAGATGACATAGAGAACACCAAAAATGAAGAAAAACCAGAAACTACAAAAGACTCTTCTAAATCGAAAGACCCCACAATCTCGATCGAAGAATCAAACAAAGAGCTTTCGGGGAATTTAAAATTTAAAAAGCAAG **************************************************.((((((.(((.............................))).)))))).....(((......))).........................(((((......................)))))...(((((..((........)))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060659 Argentina_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................ATAGAAGACTCTTCGAAATCGAA.................................................................... | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................AATCAAACAAAGAGCTTTCGGGGAAT................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................GAACTCTGGGGAAAGTGAGGA.................................................................................................................................................................................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................CAACAAACAGAACTCTGGGGAAAGT....................................................................................................................................................................................................... | 25 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................ACTGGAGAAAGTGAGCACTT............................................................................................................................................................................................... | 20 | 3 | 6 | 0.50 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................ACTATAGAAGACTCTTCGAAA......................................................................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................TGACATAGAGAACACCACC................................................................................................................ | 19 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................ACTCTGCGAAGTCGAAAGAC................................................................ | 20 | 3 | 11 | 0.36 | 4 | 0 | 0 | 0 | 0 | 3 | 0 | 1 | 0 |
| .........................................CTGGGGAAAGTGAGGAGT................................................................................................................................................................................................ | 18 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................ACTCTGCTAACTCGAAAGAG................................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................................GAAGGGCTTTCTGGGAATT.................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GAGAAGGTGCAGGTGGGATTTTCTTCCGTTGTTTGTCTTGAGACCTCTTTCACTCCTGAGGTAACGTAGTCTGCTGTATAGTTGATTTTGCCTAAGGAATTTATGCGGTCGAAGCCATCTACTGTATCTCTTGTGGTTTTTACTTCTTTTTGGTCTTTGATGTTTTCTGAGAAGATTTAGCTTTCTGGGGTGTTAGAGCTAGCTTCTTAGTTTGTTTCTCGAAAGCCCCTTAAATTTTAAATTTTTCGTTC
**************************************************.((((((.(((.............................))).)))))).....(((......))).........................(((((......................)))))...(((((..((........)))))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M027 male body |
V116 male body |
M028 head |
M047 female body |
SRR060687 9_0-2h_embryos_total |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR1106730 embryo_16-30h |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................................................................................................TAGCTTCTTAGTTTTT.................................... | 16 | 1 | 20 | 2.95 | 59 | 58 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .......................................................................................................................................................................................................TAGCTTCTTAGTTTT..................................... | 15 | 1 | 20 | 1.25 | 25 | 25 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CTAGCTTCTTAGTTTTT.................................... | 17 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................TCTGCGGTGTTAGAGCTA.................................................. | 18 | 1 | 2 | 1.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| GAGAAGGTGAAGGTCGGAT........................................................................................................................................................................................................................................ | 19 | 2 | 5 | 0.60 | 3 | 0 | 0 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| GAGAAGGTGAAGGTCGGA......................................................................................................................................................................................................................................... | 18 | 2 | 10 | 0.30 | 3 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 |
| ......................................................................................................................................................................................................CTAGCTTCTTAGTTTCTA................................... | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................AGCTTAGTAGTTCGTTTCTCGA............................. | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................TGGTTTTTACTTCTT....................................................................................................... | 15 | 0 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................CTGTCGGAGCCATATACTG............................................................................................................................... | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................CTCTTGTAGTTTCTACTTGTT....................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................TAACGTAGTCTGCGGTG............................................................................................................................................................................. | 17 | 2 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................TAGCTTCTTAGTTTA..................................... | 15 | 1 | 20 | 0.10 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................GTCGGAGCCATATACTGTG............................................................................................................................. | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......GTTTAGGTGGGATTTTATTC................................................................................................................................................................................................................................. | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................AGTCGAAGCCAATTACTGTA............................................................................................................................. | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................TAGTTCTCTGGGGTGTTGG....................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................TGTAGCTTCTTAGTTTGT.................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:4293486-4293736 - | dvi_1650 | CTCTTC-----CACGTCCACCCTAAAAGAAG-----------------------------------------GCAACAAACAGAACTC-------------------------------------------------------------TGGAGAAAGTGAGGACTCCATTGCATCAGACGACATATCAACTAAAACGGAT------------------------------------TCCTTAAATACGCCAGC-----TTCGGTAGATGACATAGAGAACACCAAAAATGAAGAAAAACCAGAAACTACA---------------------AAAGACTCTT---------------CTAAATCGAAAGACCCCACAATCTCGATCGAAGAATCAAACAAAGAGCTTTCGGGG---------------AA-T-------------------------------TTAAAATTTAAAAAGCAAG |
| droMoj3 | scaffold_6500:14375191-14375455 + | CTCATC-----TACGACCACCTTGAAGGAAA-----------------------------------------CTAAGAGTGAGAGCTCTGACGAGGACAGTAAAGAGGATAGAACTGTAGAAA----------------ATGAAAA------------------------------------------AGTTAAAGCAGATAAGTCTA-TAGA-----------------------GTTTTCC------TCAAC-----TTCGGTAAACGCAGTGAAAACTGACGA----------TTCTTCGT--CATCAGTAAGTACAAAGAAACCAGAAGGCGATTCCT---------------CCACATCGGTAAATGCA-CA---------------------------------GAA---------------GATTACGGGATACGCAACAGAAACTCCAGTGGTGTCCGAAGA--AGAAAAATCG | |
| droGri2 | scaffold_15252:3387407-3387591 + | TTCAAG-----TACGTCCACCTTAAGAGAAG-----------------------------------------GTGAGACACAGGAAAATAATCAGTTCTAT-------------------------------------------------AA---------------------ATTAGTCAGAACTTCGATTGAAACAGATAAGCCAGATACAGCCAGAAGTTCCACAAATTCA-----------------------------------------------------------------------------------------------------------------------------------------------------ATTAAACTATTCTACGAAGCTGTTATACCAGAGCCAATAAAACATCA-T-------------------------------TGGCAAGTTAATGAATAAT | |
| droWil2 | scf2_1100000004952:576165-576217 - | TAGAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACTTAAAAGATCAAAAAGTCCAAGAAATAAAA---------------------AAAAATTCTT---------------TCA------------------------------------------------------------------------------------------------------------------------AA | |
| droAna3 | scaffold_12943:3465031-3465133 + | TTTTTTAAATGTACATACATCGGAAAAAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCA-TA-----------------------TATTTTTTG------AG---------------------------AGTTTCGAAAAAAAACCAATACTACAAATTAGA---------------------AAAAACTCTT---------------CTGATTT---------------------------------------------------------------------------------------------------------------TAAGTAC | |
| droBip1 | scf7180000396737:1081434-1081518 - | CTTCAGAA--------GGACATGAAGAA--------------------------------------------------CACAAAGGA-----------------------------------AAAACAAAAGTCATAAACCAAAAAGTCTGAAG----------------------------------------------------------A--GCAAAGCCCGAA--------------------------------------------------------------------------------------------------------------------------------------------A----------------------AAC----------------------------------------------------------------------------------CAAACAA | |
| droKik1 | scf7180000302318:18471-18534 + | ATGGAA------------------------------------------------------------------TAAAAGAAA----------------------------------------------------------CCAAAAAGTATGAAGTA---------------------------------------------------------AACGAAAGTACGAAAGACTTA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATAAAACAAA | |
| droFic1 | scf7180000453832:30830-30895 - | ACCCCA------------------------------------------------------------------CCC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTTAATAGACGATCTTATTGAAAAACTTTCCGAA---------------AA-A-------------------------------GTAATTTTTACCAAGTAAG | |
| droEle1 | scf7180000490579:4883-4944 + | AAAATG------------------------------------------------------------------ACA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAAACCGAAAACGGTA---------------------GCAGGTTTTT---------------AGAAAT--------------------------------------------------------------------A-------------------------------GAAAATGTGACCCTTTAAT | |
| droRho1 | scf7180000774181:1167-1318 - | AAGAAT------------------------------------------------------------------ATAATAAATCG----------------------------------------------------------ATCCATTCTGTAACA---------------------------------------------------------AAAGAAAGCCCCATAAATA--TATTTTTTAGAAGCACAATCTCCAGTTCAGTAGAAGACATAGAAAAAATCGAGAAGA----------------AAGA---------------------AGAAGAA*CTCCAAAACTCTCTTGCCTAACCTGGAA--------------------------------------------------------------------------------------------------------------GACC | |
| droBia1 | scf7180000302267:8939-9056 + | AACGGTGAAACAACAG--------AATC---AACAAGTTCAAAGCAAGACAGTCCAAATACACAGAAAACGATCATT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAAGAACGAA-CGTACAAAAGGGCAACTGGA---------------AATT-------------------------------GAAAATTTT-GAAACAGAA | |
| droTak1 | scf7180000415357:68226-68320 - | TTCCA--------CATCCACTCTAAAAGCTA-----------------------------------------GCAATGAACAG------------CTCTCG-------------------------------------------------AA-TTCAAGGAGCACTCTAAA------------------------------------------GGCGGAGGAACGACACTCCGA-----------------------------------------------------------------------ACCAAA---------------------A----------------------------------------------------------------------------------------------------------------------------------------------CAGATCC | |
| droEug1 | scf7180000409528:319742-319779 + | GAAAAC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAAAAATGCAA--------------------------------------------------------------------AAATCTCGAAAACT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------ATAAAAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/19/2015 at 11:25 AM