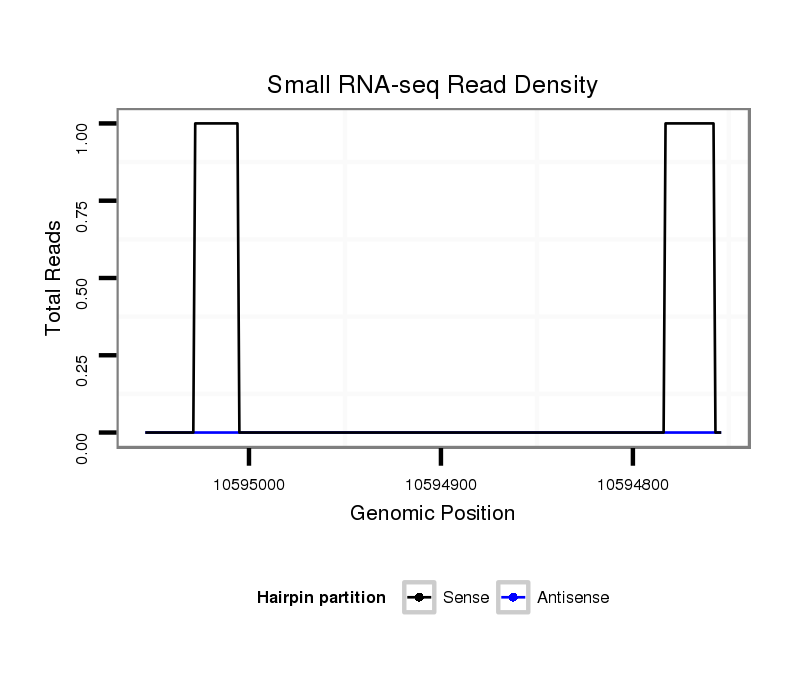
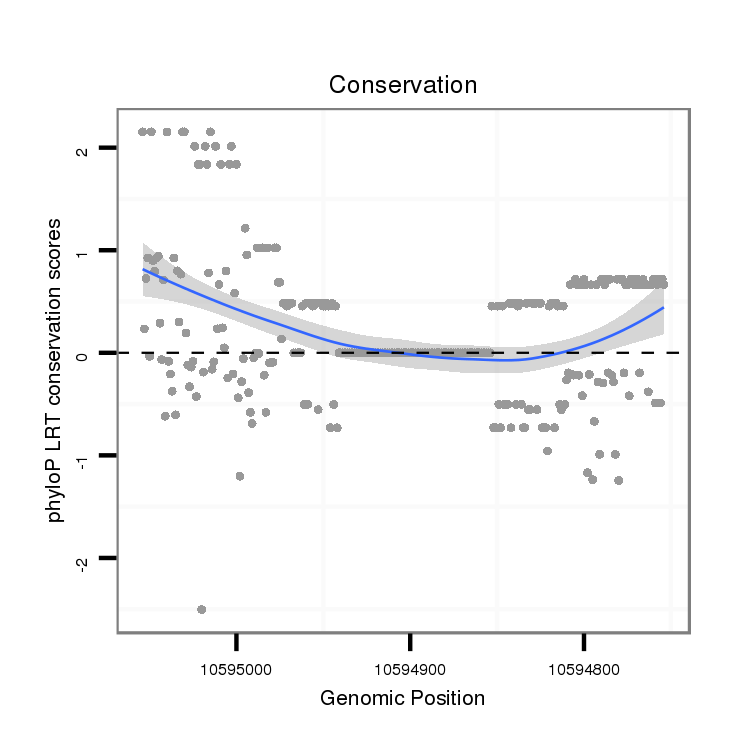
ID:dvi_16382 |
Coordinate:scaffold_13047:10594804-10595004 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ23036-cds]; exon [dvir_GLEANR_8394:1]; CDS [Dvir\GJ23036-cds]; exon [dvir_GLEANR_8394:2]; intron [Dvir\GJ23036-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGGACTACAAAACATTTTTGGACTTCGCTTTCGCACTGGAGAATCGACAGGTGTGAGTTTAGGCACACTAAGTGCGTTTAAATGTATGTTTAGCTTATCTTTAAGCTCGAACCAGAGCGGTCTAGGAAAATTTGCATTTGCTTTCAATCTGATTTTCCTAACATTAGAAGCGTATTACACTGATTTACCAAGTTGTGTCTAGACGTGGATAAGTTCCTCGCTAATATAAAGCACGTTCTCTACCAGCACAGACTTCTTCGGCAATGCACTATATCTTCCGAATGCTGGACGTGCAGCACAC **************************************************.((((.(((((((((((...(((((((((.(((((......((((((....))))))...(((.....))).((((((((((.....(((....)))...)))))))))))))))..)))))))))..(((.........))))))))))))))..(((.....)))..(((.......)))..............)))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
M027 male body |
V053 head |
SRR060670 9_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060665 9_females_carcasses_total |
V116 male body |
SRR1106729 mixed whole adult body |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................................................................................TATCTTCCGAATGCTGGACGTGCAGC.... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................TCTAACATAAAGCACGTTCT.............................................................. | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................GCTTTCGCACTGGAGAATCGACA............................................................................................................................................................................................................................................................ | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................ACCATGGTGTGACTAGACGT............................................................................................... | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................CTTCGCACTGGAGAATTCACA............................................................................................................................................................................................................................................................ | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................GAACCATAGCAGTCTAGCA.............................................................................................................................................................................. | 19 | 3 | 9 | 0.22 | 2 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GATTTACCAAGTGATGTCGA.................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................................................................TATCTTCCGAATGGTGACC........... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GATTTACCAAGTGATGTCG..................................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................CGCACTGGAGGACCGA.............................................................................................................................................................................................................................................................. | 16 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................CACTGGAGGGGCGACAGGT......................................................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................................................................................................................................................................................................................................................................................ATCTTCCGAATGGTGACC........... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
ACCTGATGTTTTGTAAAAACCTGAAGCGAAAGCGTGACCTCTTAGCTGTCCACACTCAAATCCGTGTGATTCACGCAAATTTACATACAAATCGAATAGAAATTCGAGCTTGGTCTCGCCAGATCCTTTTAAACGTAAACGAAAGTTAGACTAAAAGGATTGTAATCTTCGCATAATGTGACTAAATGGTTCAACACAGATCTGCACCTATTCAAGGAGCGATTATATTTCGTGCAAGAGATGGTCGTGTCTGAAGAAGCCGTTACGTGATATAGAAGGCTTACGACCTGCACGTCGTGTG
**************************************************.((((.(((((((((((...(((((((((.(((((......((((((....))))))...(((.....))).((((((((((.....(((....)))...)))))))))))))))..)))))))))..(((.........))))))))))))))..(((.....)))..(((.......)))..............)))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060672 9x160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060689 160x9_testes_total |
M047 female body |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
SRR060665 9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
SRR060657 140_testes_total |
SRR060683 160_testes_total |
SRR060661 160x9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................................................................................................................TAATATAGAAGTCTTAGGACC............. | 21 | 3 | 2 | 1.50 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TTTCGTCCACGAGAGGGTCG...................................................... | 20 | 3 | 7 | 1.14 | 8 | 0 | 0 | 2 | 2 | 1 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TTGGTCATGCCAGACCCTTTT........................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................................ATGGTCGTGTCTAAAGAAGAG........................................ | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................................................TTATGTGTAATAGAAGGCTTA.................. | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................TTCGGGCAAGGGATGGGCG...................................................... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TTCCGTGCAAGAGAGAGTC....................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
| ........................................................................................................................................................................................................................................TGCACGAGATGGTCGACTCT................................................. | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................................................AAGGATTTTAATATTCCCAT............................................................................................................................... | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................AACACAGATCTGGACCACT.......................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................GCCAGATCCTTTTGA......................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................................................................................................................................................................................................TGGTCGTGTCCAAAGAAGA......................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................GCCAGATCCTTTATACCG...................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................AGCTTGGACGCGCCAGTT................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:10594754-10595054 - | dvi_16382 | TGGACTACAAAACATTTTTGGACTTCGCTTTCGCACTGGAGAATCGACAGGTGTGAGTT--TAGGCACACTAAGTGCGTTTAA-----ATG----T------------------------------------ATGTTTAGCTTATCTTTAAGCTCG--------AACCAGAGCGGTCTAGGAAAATTTGCATTTGCTTTCAATCTGATTTTCCTAACATTAGAAGCGTATTACACTGATTTACCAAGTTGTGTCTAGACGTGGATAAGTTCCTCGCTAATATAAAGCACGTTCTCTACCAGCACAGACTTCTTCGGCAATGCACTATATCTTCCGAATGCTGGACGTGCAGCACAC |
| droMoj3 | scaffold_6540:28719665-28719906 - | TGGACTACAAAACGTATCTGGGCTTCGTCCTGGCCCTGGAGAATCGACAGGTGTGAGTT--TA-------------------AGCCGAATGAACTTACGAGCAGGATTCATATTGTTTGGATAGTGTACTAAAT-----GTTCATCTTAAAGCTCTTTGGCTCTAGCA----------------------------------------------------------------------------------------GCAGCTGGCGATTTTCTTTATTTTATTAATACGGTTATCCTCTTGCATAGACATCCTCCTCGTTGCACTACTTATTCCGTATGCTGGACGTGCAGCACAC | |
| droGri2 | scaffold_14624:332526-332612 - | TTCTTTCCTAATCATGCACCACCTTTTC----------GA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACGCAGTCATCGCCATCGCTTCATTTCCTGTTTCGTATGCTAGACGGGCACCAGAC | |
| droWil2 | scf2_1100000004921:2671800-2671858 - | TGGACTACAAAACATTCTTGGACTTTGTCCTCGCTCTGGACAATCGTCAGGTATGGCTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 2:15402981-15403058 + | TGGACTACAAGACCTACCTCGACTTTGATATGGCTATGGAAAATCGTAGAGTAAGGAGG-----CTACACTATCTGTATTTAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_0:5010314-5010391 - | TGGACTACAAGACCTACCTCGACTTTGATATGGCGATGAATAATCGTAGAGTAAGGAGG-----CTACACTATCTGTATTTAA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12911:2815870-2815949 - | TAGATTATAAGGGGTTTTTGGAATTTTTCTTCGCCCTGGAAAACAGAGATGTAAGTTAATCTATGGTTATTAAGTTCGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBip1 | scf7180000396714:294704-294763 + | TGGACTATAAAGGATTTTTGGACTTTTTCTTCGCCTTGGAATATAGAGATGTAAGTTAA-----------------------G--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491008:675127-675182 - | TGGACTACAGAACCTTCCTGGACTTTTACTTTGCAATGGAACATCGAAAAGTAAGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000768079:829-888 + | TGGACTACAAAACCTTTCTGGACTTTTACTTTGCCATGGAATATCGAAAAGTGAGATCT--T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000302402:9058384-9058441 + | TGGATTACAAATCCTTTCTCGACTTCCACTTTGCAATGGAAAATCGAAAAGTGAGGGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415630:57584-57641 + | TGGACTACAAATCCTTTCTTGACTTTTATTTTGCAATGGAAAACCGAAAGGTGAGAAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 10:23 PM