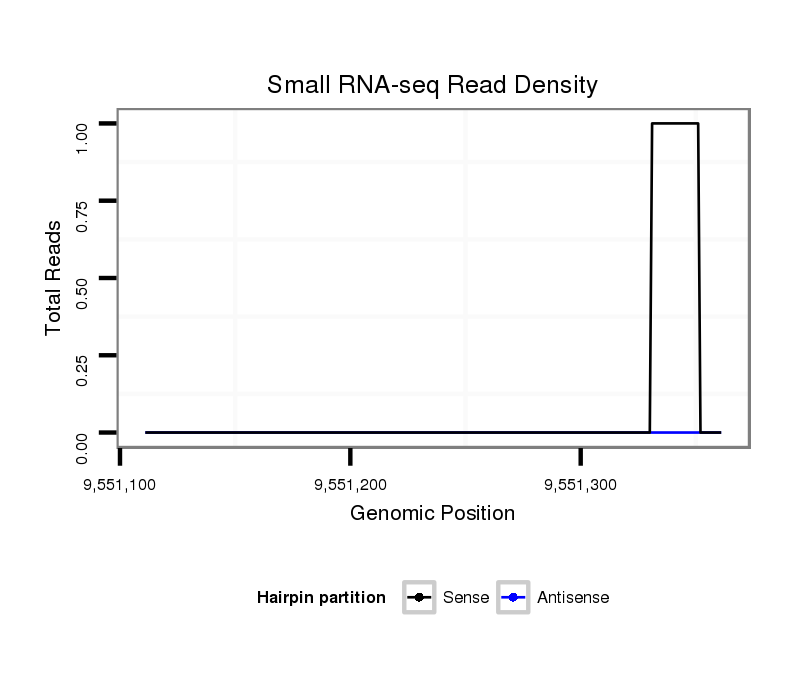
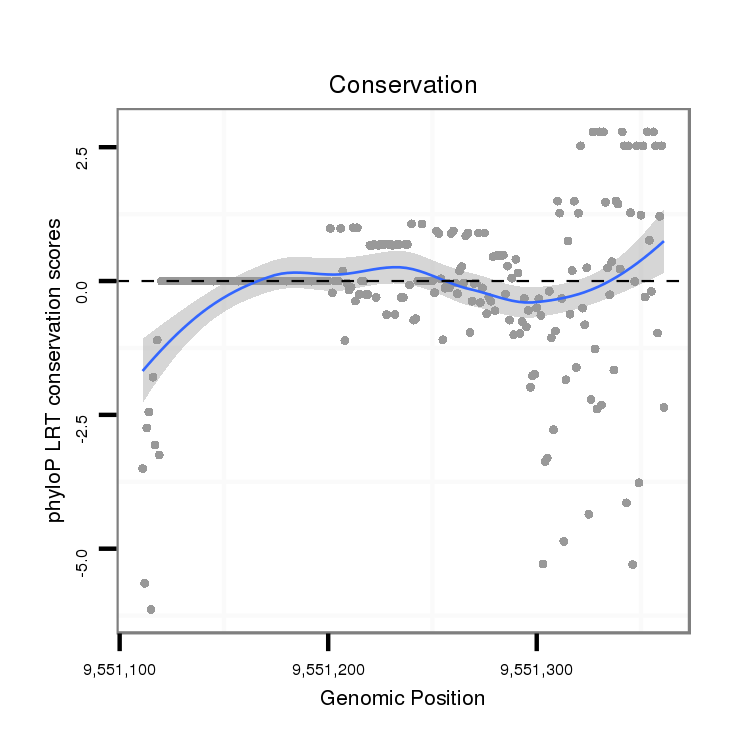
ID:dvi_16172 |
Coordinate:scaffold_13047:9551161-9551311 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ24124-cds]; exon [dvir_GLEANR_9427:4]; intron [Dvir\GJ24124-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GATACGAAACGAATTCGATATCCATCGATTTTAAGCACAAGCTATTTGCTATTATAAGCTTAAGAATATCTAAATTTTGAAACGCCTCCTTGAAAATTGCTTTACAATCGCATGAATGAAGCTATTTACAGGGTATGCTCTAGTCAGACATTCTCGAATAGAGCAAAAGTATTTGTTTTTCATTATCTTTAACTCACGCAGCGCAACTGCCTGCCTTGATTAAGATCTTCTCGGCACGTGCCAATTCCCGA **************************************************.......................((..(((((.((..(((((.((((.((((((...((....)).)))))).)))).)))))..(((((((.(((((...))).)).))))))).........)).)))))..))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060687 9_0-2h_embryos_total |
M027 male body |
SRR060666 160_males_carcasses_total |
M047 female body |
M028 head |
SRR060665 9_females_carcasses_total |
V116 male body |
SRR060669 160x9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
M061 embryo |
SRR060657 140_testes_total |
SRR060663 160_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060664 9_males_carcasses_total |
V053 head |
SRR060660 Argentina_ovaries_total |
SRR060675 140x9_ovaries_total |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....GAAACGGATTCGATACCCGT.................................................................................................................................................................................................................................. | 20 | 3 | 4 | 6.50 | 26 | 2 | 0 | 2 | 0 | 0 | 3 | 0 | 4 | 3 | 1 | 3 | 0 | 0 | 0 | 1 | 0 | 0 | 2 | 0 | 2 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....CGAAACGGATTCGATACCCGT.................................................................................................................................................................................................................................. | 21 | 3 | 2 | 4.50 | 9 | 3 | 0 | 1 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....CGAAACGGATTCGATACCC.................................................................................................................................................................................................................................... | 19 | 2 | 2 | 3.00 | 6 | 1 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....GAAACGGATTCGATACCC.................................................................................................................................................................................................................................... | 18 | 2 | 4 | 2.50 | 10 | 1 | 4 | 0 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................TAAGATCTTCTCGGCACGTGC.......... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......AAACGGATTCGATACCCGT.................................................................................................................................................................................................................................. | 19 | 3 | 13 | 0.92 | 12 | 1 | 0 | 0 | 1 | 0 | 1 | 1 | 0 | 0 | 1 | 1 | 0 | 2 | 1 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................AATCTCCAGTAGAGCAAAAG.................................................................................. | 20 | 3 | 6 | 0.67 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...GCGAAACGGATTCGATACCC.................................................................................................................................................................................................................................... | 20 | 3 | 7 | 0.57 | 4 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................ATTTTGAAACGCCTGCTTGGA............................................................................................................................................................. | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....CGAAACGGATTCGATACCCGTC................................................................................................................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....CGAAACGGATTCGATACC..................................................................................................................................................................................................................................... | 18 | 2 | 8 | 0.38 | 3 | 0 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................ATAGGCTTAAAAATATCTAAA................................................................................................................................................................................. | 21 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...GCGAAACGGATTCGATAACC.................................................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................GCCGGCCTTGATTATGAACT....................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................AAATTCTTGAATAGGGCAAAA................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................................................................................................ATCTCCAGTAGAGCAAAAG.................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
CTATGCTTTGCTTAAGCTATAGGTAGCTAAAATTCGTGTTCGATAAACGATAATATTCGAATTCTTATAGATTTAAAACTTTGCGGAGGAACTTTTAACGAAATGTTAGCGTACTTACTTCGATAAATGTCCCATACGAGATCAGTCTGTAAGAGCTTATCTCGTTTTCATAAACAAAAAGTAATAGAAATTGAGTGCGTCGCGTTGACGGACGGAACTAATTCTAGAAGAGCCGTGCACGGTTAAGGGCT
**************************************************.......................((..(((((.((..(((((.((((.((((((...((....)).)))))).)))).)))))..(((((((.(((((...))).)).))))))).........)).)))))..))...............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060684 140x9_0-2h_embryos_total |
GSM1528803 follicle cells |
SRR060657 140_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................CGAAATCTTATAGATTGAAA.............................................................................................................................................................................. | 20 | 2 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................CTATAGGTCGCTAAA............................................................................................................................................................................................................................ | 15 | 1 | 15 | 0.07 | 1 | 0 | 0 | 1 | 0 | 0 |
| .................TAGAGGTAGGTAAAATTC........................................................................................................................................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| .......................................................TCCGAATTTTTATAGATTT................................................................................................................................................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................................................GAAAAGTAATAGAAACTG.......................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................TCGCATTGAGGGACGGAT.................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:9551111-9551361 + | dvi_16172 | GATACGAAACGAATTCGATATCCATCGATTTTAAGCACAAGCTATTTGCTATTATAAGCTTAAGAATATCTAAATTTTGAAACGCCTCCTTGAAAATTGCTTTACAATCGCATGAATGAAGCTATTTACAGGGTATGCTCTAGTCAGACATTCT----------------------CGAATAGAGCAAAAGTATTTGTTTTTCAT----TATC-------------------TTTAACTCACGCAGCGCAACTGCCTGCCTTGATTAAGATCTTCTCGGCACGTGCCAATTCCCGA |
| droMoj3 | scaffold_6540:12121987-12122105 + | GTATATGCT------------------------------------------------------------------------------------------------------------------------------------AGTGCAGCAATCT----------------------CGACAAGAACACGTGAATTTATGTCTAAT----CCAT-------------------TTAATCTCTTGCAGACCAACTGTCCGCCTTGATTAAGATCTTCTCAGCACGCGCCAACTCCCGT | |
| droGri2 | scaffold_14906:2441507-2441583 + | TAATTGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TA----ATTT-------------------CATTTCATTTACAGAACAATTACCCGTCTTGGTTAAGATCTTCTCGGCACGCGCCAATTCCCGA | |
| droWil2 | scf2_1100000004902:4979887-4980029 + | CCTCTATGA---------------------------------------------------------------------------------------TTACTTTAT--CTGCAAGAATCAAGGTAAATACATA--A---------CTAA--AACCC---------GCTTTAGCAACATGAGTTAAAAAAAA------------------------------------------GAATTAATTTTTAGCACAACTGCCTCCAATTGTGAAGATTTTCTCAGCACGTGCAAATTCAAGG | |
| dp5 | 2:2901967-2902048 - | CAAAATGAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTACC----TCGT-------------------ATTTTGCTTTTCAGGTAGTGTACCTGCGATTGTGAAGATTTGCTCGGCCCGGTCAAGTTCCCGT | |
| droPer2 | scaffold_1196:2244-2346 - | CGGAGAGAA---------------------------------------------------------------------------------------CGGAATGA----------------------------------------------------TTTGCAAG---------AT---------------------GA-TTTACC----TCGT-------------------CTTTTGCTTTTCAGGTAGTGTACCTGCGATTGTGAAGATTTTCTCGGCACGGTCAAGTTCCCGT | |
| droAna3 | scaffold_13340:23544040-23544111 + | TCAGTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TG-------------------TATTATTTGCTTAGCCAAACTGCCCACCATTTTGAAGGTTTTCTCCGCTCGTGCCAACTCCCGT | |
| droBip1 | scf7180000396413:2274674-2274759 - | AAATATTTC-----------------------------------------------------------------------------------------------------------------------------------------------------------TT-------------------------------T--GT--CATG-ACTA-------------------TCTGTAACTTTCAGATGAGCTTCCTGCTGTTGTAAAAATATTTTCGGCCCGAGCCAATTCCCGA | |
| droKik1 | scf7180000302696:420266-420373 - | AGGGTAGGA---------------------------------------------------------------------------------------TC------------------------------------------CAGGGAGACATT--CACTGAGTA----------AA---------------------A------CC----TTGG----------------GAAATCTTCCTTCGCAGAGTCACTGCCTCCGGTGGTCAGAGCCTTCTCGGCCAGGGCCAACTCTCGG | |
| droFic1 | scf7180000454096:350792-350897 + | ATTTTCGAC---------------------------------------------------------------------------------TA---AT----------------------------------------------GAAGC--TCCTTGCTTATTG-----------------------------------------T------TC-----------CTTTTAATTTTTCCTATATTAGAGAAACTGCCTGCCCTGATTAAGATTTTCTCCGTACGTGCCAATTCCCGA | |
| droEle1 | scf7180000491261:151876-151974 + | GAATAACAC---------------------------------------------------------------------------------------CT----------------------------------------------------------------------------------------------------------AAATTT-TTGATTTCCGAAACTAAT---AATATCTTTTACAGATCAGCTTCCCCTGATAGTCAAGGTGTTTTCGGCCAGGGCCAACTCGCGT | |
| droRho1 | scf7180000779872:323830-323941 - | CAGGGTAAG---------------------------------------------------------------------------------------TA------------------------------AACC--A---------AAGA--TCATAGTTTAAAGG---------AA---------------------AT-GTAATA----ACAA-------------------TTTGTAACCTTCAGACCAGCTTCCTGCCATTGTGAAGGTATTTTCAGCCAGGGCCAATTCTCGA | |
| droBia1 | scf7180000302155:440996-441067 + | ATTATT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TT-------------------TATTATACTCGTAGAAAAACTGCCTGCTCTCCTGAAGGCTTTCTCCGCTCGTGCCAATTCCCGA | |
| droTak1 | scf7180000415417:299772-299858 - | AAAAATCCC-----------------------------------------------------------------------------------------------------------------------------------------------------------CCTTT-------------------------------CTAAA----TTTC-------------------ATTTAAATTTGTAGAAAATCTGCCTGCCATTCTGAAGGTTTTCTCTGCACGTGCCAATTCCCGC | |
| droEug1 | scf7180000409230:853328-853410 - | AAATGTGTT---------------------------------------------------------------------------------TG---AT----------------------------------------------------------------------------------------------------------TA----TTAT-------------------CTTCTATCTTGTAGGTAAGCTACCTTCTTTTGTAAAGGCCTTCTCAGCCCGTGCCACTTCACGT | |
| dm3 | chr3R:19423903-19423970 - | ATTTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTTGCACAACTCAACAAACACCTGCCTTGATAAAGATTATATCGGCTAGAGCGAATTCCCGA | |
| droSim2 | 2r:20795903-20795965 - | CGCT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCTTTCAGATCAACTTCCAGCGATGGTCAAGGTATTCTCGGCCAGGGCCAATTCCCGG | |
| droSec2 | scaffold_38:60367-60437 - | TTTTAATAC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTATATTTGTAGAAAAACTGCCTGCTGTTCTGAAAGTTTTCTCTGCACGTGCTAACTCACGA | |
| droYak3 | 2R:7949644-7949726 - | TATGCATAT---------------------------------------------------------------------------------------TT----------------------------------------------------------------------------------------------------------CACGGT-TT-------------------TAGTTCATCCTCAGATCAGCTTCCCAGCATTGTGAAGATCTTCTCGGCTCGGGCCAATTCCCGA | |
| droEre2 | scaffold_4770:4591228-4591295 - | TCTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAATGTACTTGTAGAAGAACTGCCTGCTATTCTGAAGGTTTTCTCTGCACGTGCTAATTCACGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 07:52 PM