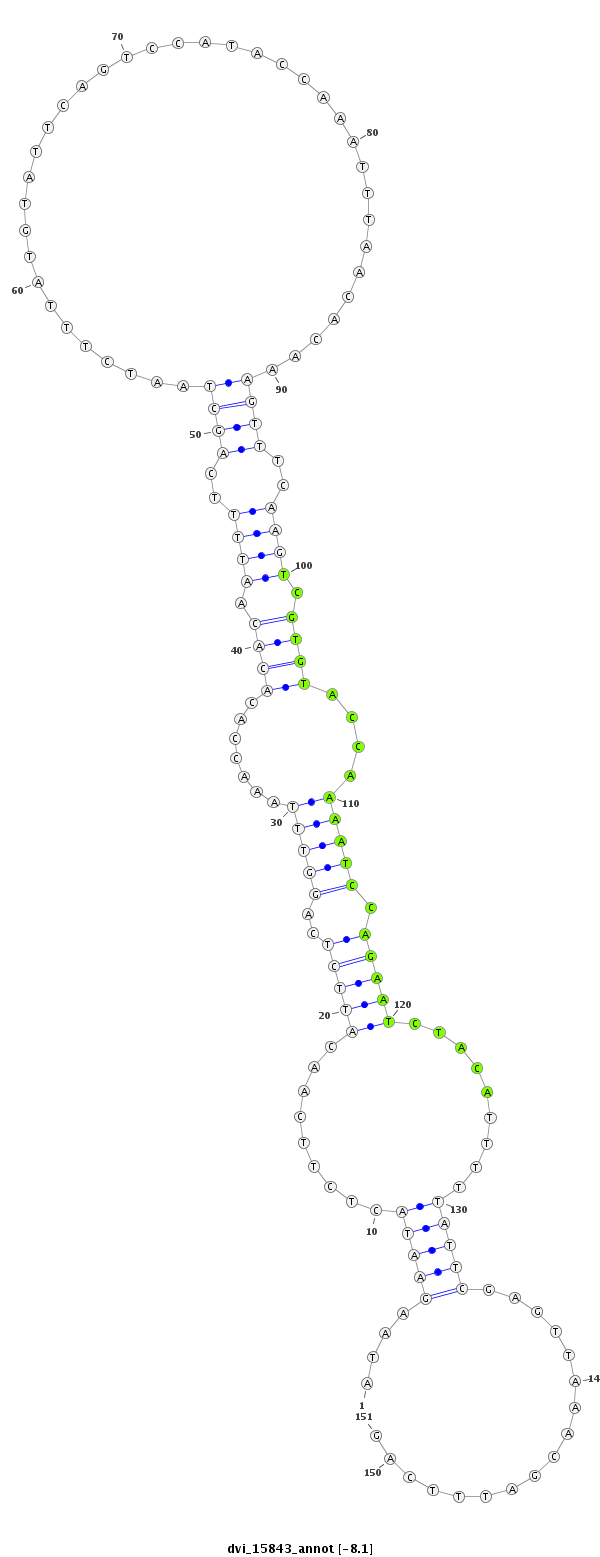
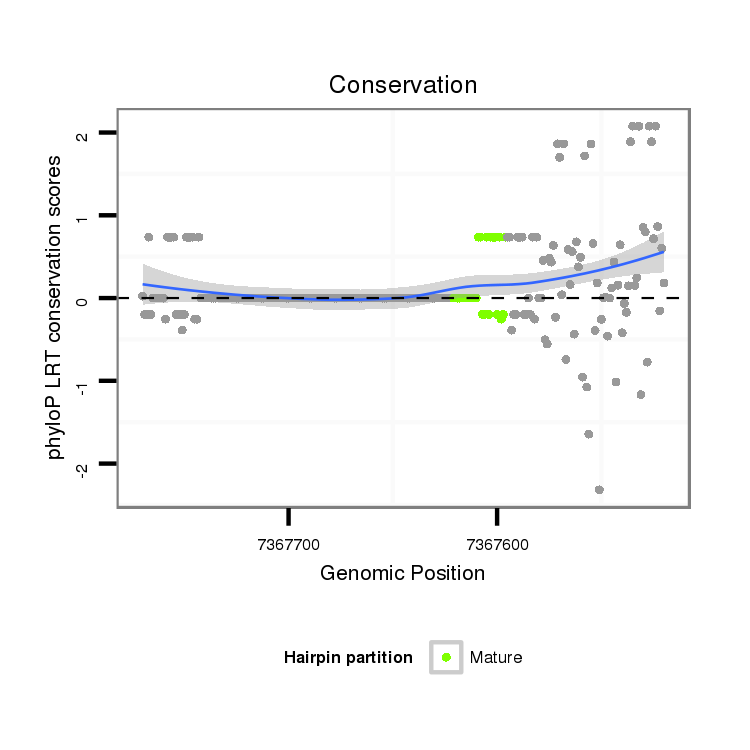
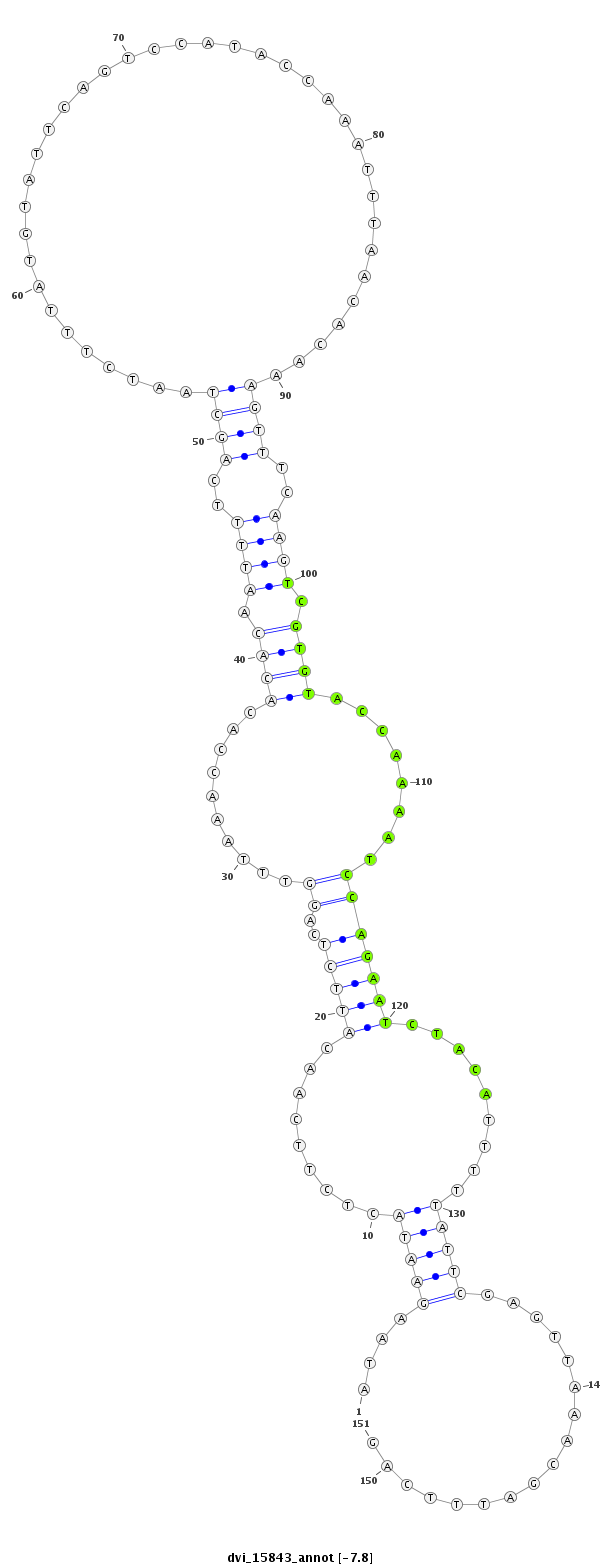
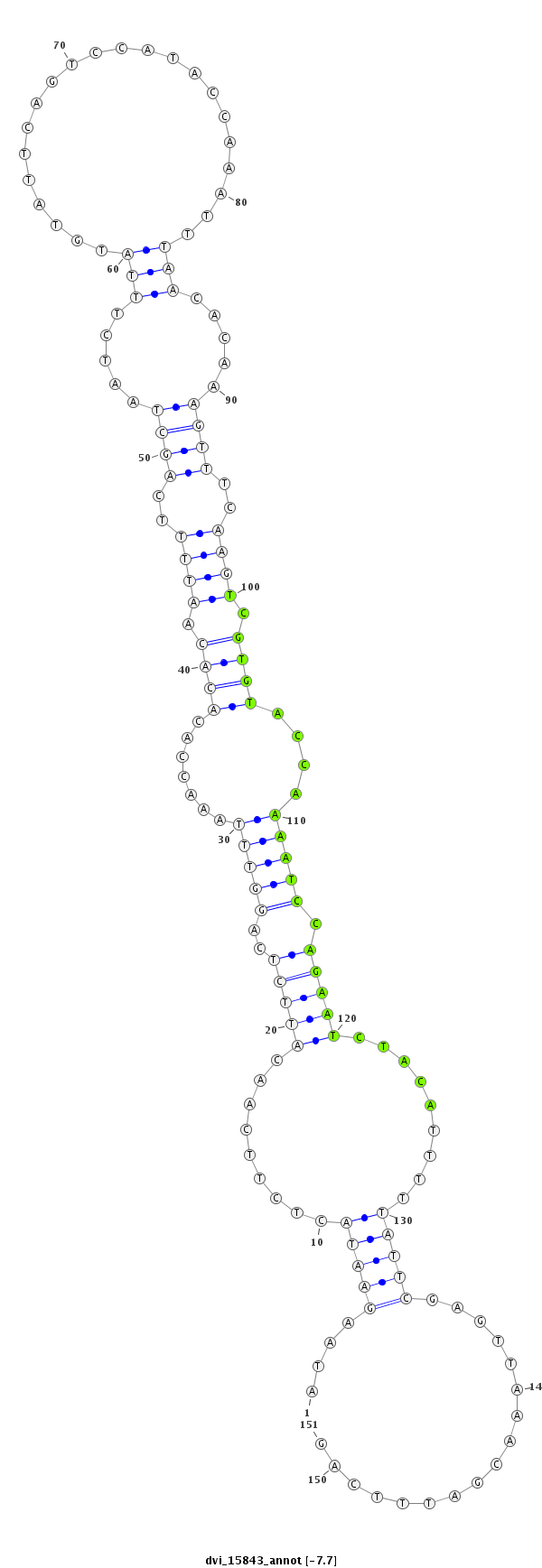
ID:dvi_15843 |
Coordinate:scaffold_13047:7367570-7367720 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -7.93 | -7.75 | -7.7 |
 |
 |
 |
exon [dvir_GLEANR_8593:2]; CDS [Dvir\Obp85a-cds]; intron [Dvir\Obp85a-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AAAATGATTTTAACAAGTTTGAGAAAATGTACCATTCAGATTTGAAGAACATAAGAATACTCTTCAACATTCTCAGGTTTAAACCACACACAATTTTCAGCTAATCTTTATGTATTCAGTCCATACCAAATTTAACACAAAGTTTCAAGTCGTGTACCAAAATCCAGAATCTACATTTTTATTCGAGTTAAACGATTTCAGCTATTGCTCTTTATTTTTGCTAATGCCGAGCCCGAGCTGAGCTGCAAGGA **************************************************....(((((.........(((((..(((((.......((((.((((..((((......................................))))..)))).))))....))))).))))).........))))).................************************************************** |
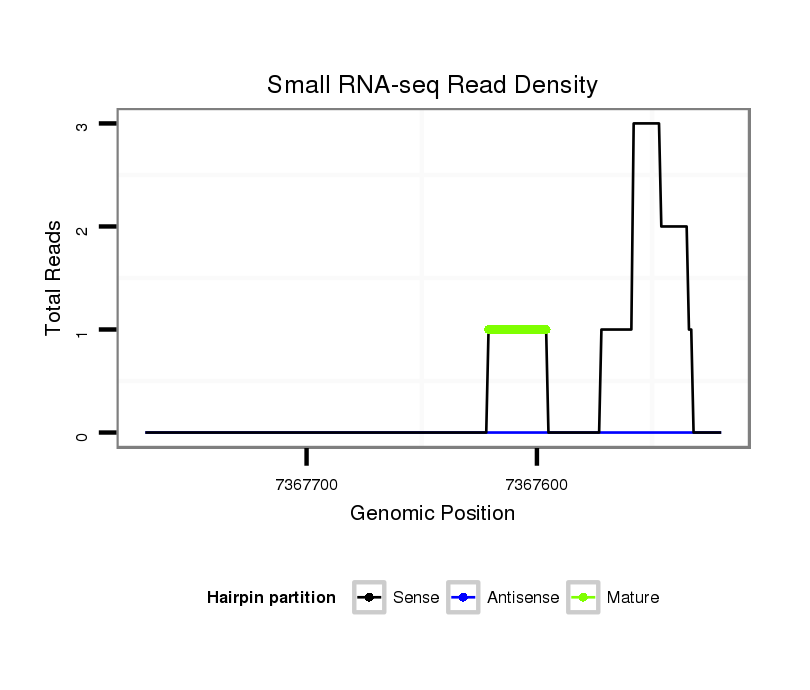
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060675 140x9_ovaries_total |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................TATTTTTGCTAATGCCGAGCCCGAGC............. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CAGCTATTGCTCTTTATTTTTGCTAA........................... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................TATTTTTGCTAATGCCGAGCCCGA............... | 24 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................TCGTGTACCAAAATCCAGAATCTACA............................................................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 |
| ...........................................GAAGACCGTAAGAACACTCTT........................................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................................................................TTACTTTTCGCTAATGCCG...................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
|
TTTTACTAAAATTGTTCAAACTCTTTTACATGGTAAGTCTAAACTTCTTGTATTCTTATGAGAAGTTGTAAGAGTCCAAATTTGGTGTGTGTTAAAAGTCGATTAGAAATACATAAGTCAGGTATGGTTTAAATTGTGTTTCAAAGTTCAGCACATGGTTTTAGGTCTTAGATGTAAAAATAAGCTCAATTTGCTAAAGTCGATAACGAGAAATAAAAACGATTACGGCTCGGGCTCGACTCGACGTTCCT
**************************************************....(((((.........(((((..(((((.......((((.((((..((((......................................))))..)))).))))....))))).))))).........))))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060659 Argentina_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060681 Argx9_testes_total |
V116 male body |
SRR060678 9x140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060680 9xArg_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
V047 embryo |
SRR060660 Argentina_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................................................TGTAAAGCTCCAAATTTGG...................................................................................................................................................................... | 19 | 3 | 20 | 1.70 | 34 | 0 | 6 | 0 | 0 | 5 | 5 | 0 | 4 | 0 | 3 | 3 | 3 | 1 | 0 | 2 | 0 | 0 | 0 | 1 | 1 | 0 |
| ............................................TTCTAGTTTTCTTATGAGACGTT........................................................................................................................................................................................ | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................ATGAGTGCAAATGTGGTG.................................................................................................................................................................... | 18 | 3 | 20 | 0.70 | 14 | 0 | 5 | 0 | 6 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................TGTAAGGCTCCAAATTTGGC..................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................GAGTGCAAATGTGGTGTAT................................................................................................................................................................. | 19 | 3 | 17 | 0.18 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................ATGAGTGCAAATGTGGTGT................................................................................................................................................................... | 19 | 3 | 19 | 0.16 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................TGTAAAGCTCCAAATTTGGT..................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................TTAGAAGTCGGTTAGGAATA............................................................................................................................................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................TCTTATGAGAAGTTCACAGA.................................................................................................................................................................................. | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TACAGAAGCGAGGTATGGT........................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................TCATGTATGGTTTAAATTT................................................................................................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:7367520-7367770 - | dvi_15843 | AAAATGATTTTAACAAGTTTGAGAAAATGTACCATTCAGATTTGAAGAACATAAGAATACTCTTCAACATTCTCAGGTTTAAACCACACACAATTTTCAGCTAATCTTTATGTATTCAGTCCATACCAAATTTAACACAAAGTTTCAAGTCGTGTACCAAAATCCAGAATCTACATTTTTATTCGAGTTAAACGATTTCAGCTATTGCTCT-TTATTTTTGCTAATGCCGAGCCCGAGCTGAGCTGCAAGGA |
| droMoj3 | scaffold_6540:3287375-3287428 - | A-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATTTTTAGCTGTCTCT---GTGTTTTTAT----GCCAAAGCCGAGCTGAGCTGCAGAGA | |
| droGri2 | scaffold_14830:3210423-3210475 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCAGTTGTCGCACT---TTTTGTTTGTT-GTTGATGGCGAGCTAAGTTGCAAGGA | |
| dp5 | 2:23203878-23203933 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTAGCTGCTGGTCTGCTTCTCTTCGCTG-ATGAGTGGGGAGATCAACTGCAAGGC | |
| droPer2 | scaffold_3:5976229-5976284 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTAGCTGCTGGTCTGCTTTTCATCGCTG-ATGAATGGGGAGCTCAACTGCAAGGC | |
| droAna3 | scaffold_13340:4157600-4157704 + | TGGAC------TACAAACCGAAGAATTT-------------------------------------------------------------------------------------------------------------------------------------ATTTAAAATTTTTATTGCCATTTA-ATAA------TGCAGGTTCTGTTGTGGTTCTTGAGCCTC-ATAAACTCTGAAATAAACTGCAAAGC | |
| droBip1 | scf7180000396570:353992-354010 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGATATAAACTGTAAAGC | |
| droKik1 | scf7180000302639:2367696-2367713 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGAAATCAACTGCAAATC | |
| droTak1 | scf7180000415750:167504-167559 - | T---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCAGCTGCTGATCAGTTTTTTTTCCCCG-ATGAACTGCGAAATCGATTGCAAGTT | |
| dm3 | chr3R:4246185-4246239 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGCTACTGCTCAGGTATTTTTACCTG-TTCAACTGCGAAATTAATTGCAAATT | |
| droSim2 | 3r:16730076-16730130 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGCTACTGCTCAGGTACTTTTACCTG-TTCAACTGCGAAATTAACTGCAAATT | |
| droSec2 | scaffold_0:17592731-17592785 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGCTACTGCTCAGGTACTTTTACCTG-TTCAACTGCGAAATTAAATGCAAATT | |
| droYak3 | 3R:8316153-8316207 - | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGCTACTGCTCAGGTATTTTAACCTG-ATCAACTGCGAAATAAACTGCAAATT | |
| droEre2 | scaffold_4770:17330722-17330776 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGCTGCTGCTCAGGTGCTTTTGCCTG-ATCAACTGCGAAATTAATTGCAAATT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:39 PM