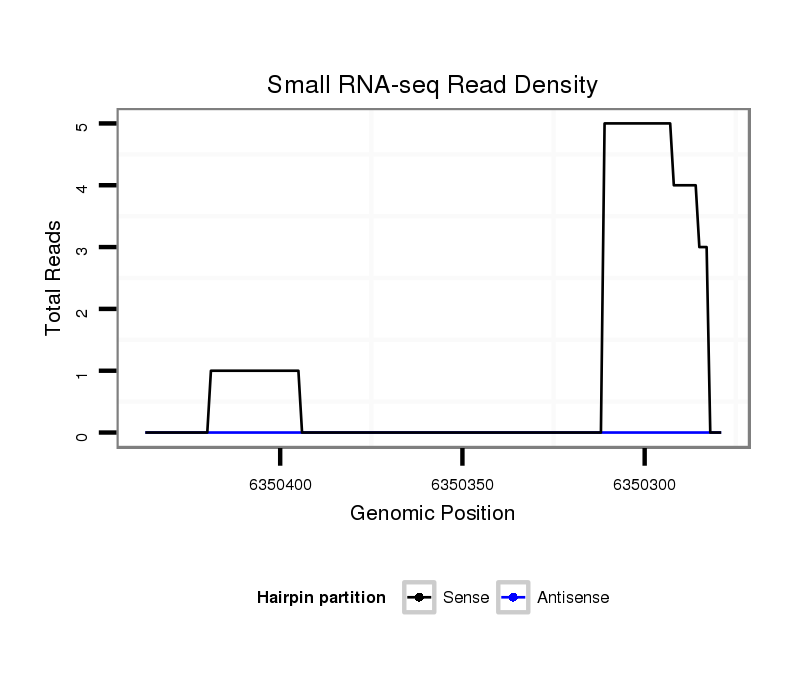
ID:dvi_15706 |
Coordinate:scaffold_13047:6350329-6350387 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ23294-cds]; exon [dvir_GLEANR_8645:9]; exon [dvir_GLEANR_8645:8]; CDS [Dvir\GJ23294-cds]; intron [Dvir\GJ23294-in]
No Repeatable elements found
| ##################################################-----------------------------------------------------------################################################## CACACGGCGGGACGCTAAACTGGAACATCATCGTAACTTTTGCCGCGATCGTAAGTTACAGTCGAATTGTGGGAAGAACTGTTTTTAATTCACGTTTGAATGATTGCAGCTTTGCCGAATAGCTCTGACGACGAGGAGACATTCTCGTCACCCGCGGAA **************************************************........((.(((((.((((((((((....)))))...)))))))))).)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M047 female body |
SRR060662 9x160_0-2h_embryos_total |
M061 embryo |
SRR060661 160x9_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
V116 male body |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................GACGACGAGGAGACATTCTCGTCACCCGC.... | 29 | 0 | 1 | 3.00 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................GATCCTTTGCCGAATAGCTCTGACGAC........................... | 27 | 3 | 1 | 3.00 | 3 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................GAACATCATCGTAACTTTTGTCGC................................................................................................................. | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................GACGACGAGGAGACATTCT.............. | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................GACGACGAGGAGACATTCTCGTCACC....... | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................ACGAGGAGACATTGTAGTC.......... | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................GTAACTTTTGTCGCGATC............................................................................................................. | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................ACTGGAACATCATCGTAACTTTTGC.................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................ACGAGGAGACATTGTAGT........... | 18 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................ACTATGGCCGAGATCGTAAG........................................................................................................ | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................................................................................CAACGAGGAGACATTGTAGTC.......... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................CAACGAGGAGACATTGTAGT........... | 20 | 3 | 12 | 0.17 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................GTCAGATTCTCGTCACCCG..... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTGTGCCGCCCTGCGATTTGACCTTGTAGTAGCATTGAAAACGGCGCTAGCATTCAATGTCAGCTTAACACCCTTCTTGACAAAAATTAAGTGCAAACTTACTAACGTCGAAACGGCTTATCGAGACTGCTGCTCCTCTGTAAGAGCAGTGGGCGCCTT
**************************************************........((.(((((.((((((((((....)))))...)))))))))).)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060675 140x9_ovaries_total |
SRR060662 9x160_0-2h_embryos_total |
V116 male body |
SRR060655 9x160_testes_total |
SRR1106729 mixed whole adult body |
SRR060682 9x140_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................ACGTCGAAACGGCCTATCGAGACTG.............................. | 25 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................AAACTTACTATCGACGAA............................................... | 18 | 2 | 17 | 0.29 | 5 | 0 | 4 | 0 | 0 | 0 | 1 | 0 |
| ..................TGACCTTATAGTAGCATC........................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................ATAACGTGGAAATGGCTTA....................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 3 | 0 | 0 | 0 |
| ....................................................................CACCCTTCAGGACAAAA.......................................................................... | 17 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................GTGACCTTATAGTAGCATC........................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:6350279-6350437 - | dvi_15706 | CACACGGCGGGACGCTAAACTGG-----AACAT-CATCGTAACTTTTGCCGCGATCGTAAGTTACAG---TCGAATTGTGGG-----AAG--AA--------------CTGT---TTTT-----------AATTCACGTTTGAATG-------ATTGCAGC-TTTGCC-------------------------------GAATAGCTCTGACGACGAGGAGAC---ATTCTCGTCACCCGCGGAA |
| droMoj3 | scaffold_6540:1728784-1728944 + | AAGGCGCCGAGACGCCAAACTGG-----AGCAA-CATCGTAACTTCTGTCGGGATCGTAAGTGTTGA---TCAAAGCATTTG-----AGAATGC--------------AGAA---GCCT-----------AAGCCACTAGTTAATC-------CCTGCAGC-TTTACC-------------------------------GAATACATCGGACGATGAGGAGAC---TTTCTCATCGCCTGCAGAG | |
| droGri2 | scaffold_14906:11669686-11669854 - | AATTCACCGTGACGCTGAACAGG------------AGCGCAACTTTTGTTGGGATCGTAAGTGACCACAATTATTTTGCATA-----GAG-------TCATCAAATTAGTGTGCTTATCCAACAT-----------CTCCCAA---CATT---TTTATAGC-ATTGCC-------------------------------AAATAGTTCCATCGATAAGATGAC---CTTCTCGTCGCCGGCGGAA | |
| droWil2 | scf2_1100000004902:8519851-8520012 + | CACACACAGAGACGCCAAACTGGAGTCACATCAGCAGCGTAACTACTGTTGGGATCGTAAGTAGTAA---TAATATCAAAAG-----ATTGTGC--------------ATAGAT---TTTAACAT-----------CGATCGATTT---CTTATCATTAGC-ATTACC-------------------------------CAACAATAATGAGGATAATATTATTGT------------TGAGAAA | |
| dp5 | 2:23784161-23784334 - | CACCATACGGACGGCCAAGGATA-----CACCA-CAACGGAACTACTGCGTGGATCGTGAGTGAAGA---TTTTATTGCCGG-----AGAATAA--------------ATGTATTTTCT-----------AAGCCACTTCCCATACCATTCCATTCACAGCCATTGC--------------------------------CAAGGCCGTTGACAATGAGGTGATTGTGGCCCCAACGCCGGCAGAG | |
| droPer2 | scaffold_3:6557825-6558000 - | CACCATACGGACGGCCAAGGATA-----CACCA-CAACGGAACTACTGCGTGGATCGTGAGTGAAGA---TTTTATTGCCAA-----AAAATAT---------AT---ATGTATTTTCT-----------AAGCCACTTCCCATTCCATTCCATTCACAGCCATTGC--------------------------------CAAGGCCGTTGACAATGAGGTGATTGTGGCCCCAACGCCGGCAGAG | |
| droAna3 | scaffold_13340:5716736-5716912 + | AACGA-----------------G-----AACCTGACCCGCAACTACTGCAAAGATCGTAAGAGCATTCGAT---GTCCCTTTTTGGTGAAATAA--------------ATAT--------TATTTTATTTAAAC--------------TTCTATCCACAGCCATTACCAAATACACTGACGACGACCCGAAACCAAAGC---CTGCCCTGCC--GGAGAAGGTTCTGG--ACACCGACAACGGAA | |
| droBia1 | scf7180000302402:6173124-6173267 - | CCG-------------------G-----AACAATAACCGAGACTACTGCTCCGAACGTAAGCTATAT---TCTTATAATTTG-----AGGTTTAAAATAATAAAAATG---------------------------GTTCGTTGTGC-------ACTGTAGC-TCTGGA-------------------------------AAAGGCCATGGAAAATGATATGCC---GGCCACGACGCCCGTGAAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
Generated: 05/16/2015 at 09:02 PM