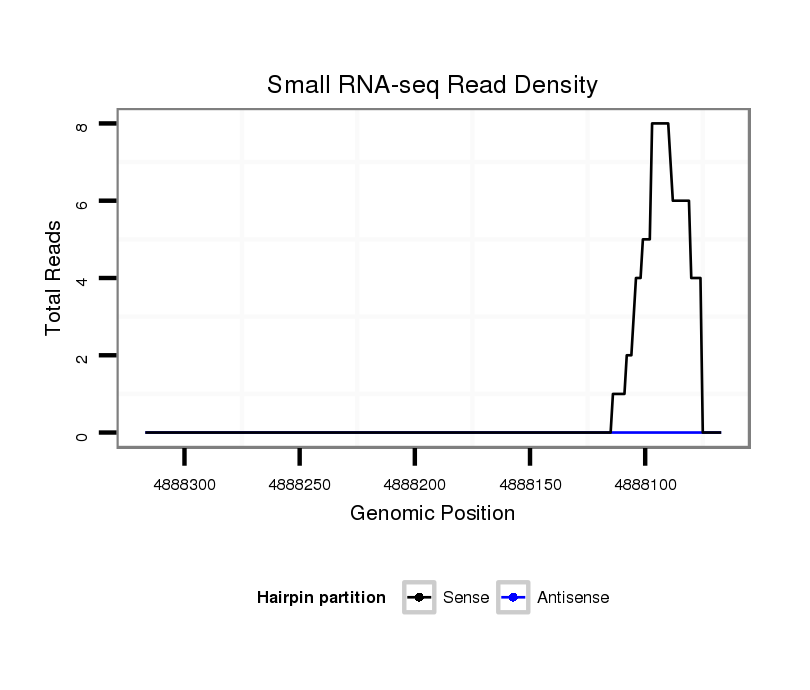
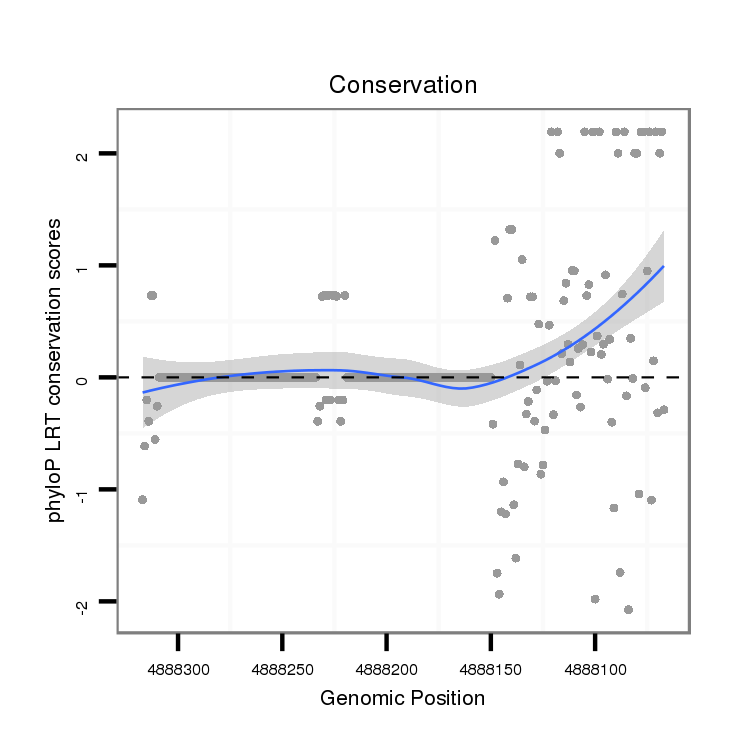
ID:dvi_15594 |
Coordinate:scaffold_13047:4888117-4888267 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ23341-cds]; exon [dvir_GLEANR_8698:2]; intron [Dvir\GJ23341-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TAGGTACACATTAATACACTGTTGATAATTGTACATACATTTATACCCTCTTTTTTCGCTCGTATTGGGTATTGGATATGTCAAGACTCAGTTCGCAAGAATATACCATATATACATAAACTACTCCAAATATATATATTTGGCATAAAGCTCTCGATGCTATTTTCTAAACTTACTTTCGTTTCTAAACTTCTCTTCCAGGCGATATAGACAGCCTATTGAAAAAGTCAGAACAGCCAATTATACAACAA **************************************************....(((...((.(((((((.((((.....)))).))))))).))...)))........................(((((((....))))))).....(((.......))).......((((........)))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060674 9x140_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060656 9x160_ovaries_total |
M061 embryo |
SRR060655 9x160_testes_total |
SRR060689 160x9_testes_total |
SRR060663 160_0-2h_embryos_total |
M047 female body |
SRR060664 9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
V053 head |
SRR060667 160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
M027 male body |
SRR060673 9_ovaries_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................................................................................GCCTGTTGAAAAAGTCGGA................... | 19 | 2 | 2 | 8.00 | 16 | 0 | 7 | 0 | 0 | 1 | 4 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TCAGAACAGCCAATAATGC..... | 19 | 2 | 1 | 7.00 | 7 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................GAAAAAGTCAGAACAGCCAATT......... | 22 | 0 | 1 | 3.00 | 3 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................AATCAGAACAGCCAATAATGC..... | 21 | 3 | 5 | 2.80 | 14 | 4 | 0 | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................CGCCTGTTGAAAAAGTCGGA................... | 20 | 3 | 9 | 1.56 | 14 | 0 | 2 | 0 | 0 | 0 | 3 | 6 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................................................CAGAACAGCCAATAATGC..... | 18 | 2 | 9 | 1.44 | 13 | 6 | 0 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................GATATAGACAGCCTATTGAAAAAGTC...................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................AGCCTATTGAAAAAGTCAGAACAGC.............. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................GCCTATTGAAAAAGTCAGAACAGC.............. | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................GACAGCCTATTGAAAAAGT....................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................TATTGAAAAAGTCAGAACAGCCAATT......... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TCAGAACAGCCAATAATGCCA... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................TCAGAACAGCCAATAATGCC.... | 20 | 3 | 8 | 0.38 | 3 | 1 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................AAATCAGAACAGCCAATAATGC..... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................AAATCAGAACAGCCAATAAT....... | 20 | 2 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................GCATTGGATACGTCCAGACT................................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................CCTGTTGAAAAAGTCGGA................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................CTTAATTTCGTTCCTAGACT............................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................AAGAACATACCATATAGAGAT...................................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................................................................................................................................................AGCCTGTTGAAAAAGTCG..................... | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................................................................ATTTATTGGGCATAAAGC.................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ATCCATGTGTAATTATGTGACAACTATTAACATGTATGTAAATATGGGAGAAAAAAGCGAGCATAACCCATAACCTATACAGTTCTGAGTCAAGCGTTCTTATATGGTATATATGTATTTGATGAGGTTTATATATATAAACCGTATTTCGAGAGCTACGATAAAAGATTTGAATGAAAGCAAAGATTTGAAGAGAAGGTCCGCTATATCTGTCGGATAACTTTTTCAGTCTTGTCGGTTAATATGTTGTT
**************************************************....(((...((.(((((((.((((.....)))).))))))).))...)))........................(((((((....))))))).....(((.......))).......((((........)))).................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060689 160x9_testes_total |
SRR060659 Argentina_testes_total |
SRR060670 9_testes_total |
V116 male body |
SRR060668 160x9_males_carcasses_total |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................GTAGAGAAGGTCCGCTAT............................................ | 18 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................AAGAGAAGGCCCGCGATA........................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................CGAGCATAGGCCATAATCT............................................................................................................................................................................... | 19 | 3 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................AGAAAACAGCGAGTATAGCC....................................................................................................................................................................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................................................................................................................................................GAAGAGAAGGTCTTCTATC........................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................ATATGGGTGAAAAGAGGGAG.............................................................................................................................................................................................. | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:4888067-4888317 - | dvi_15594 | TAGGTACACATTAATACACTGTTGATAATTGTACATACATTTATACCCTCTTTTTTCGCTCGTATTGGGTATTGGATATGTCAAGACTCAGTTCGCAAGAATATACCATATATACATAAACTACTCCAAATATATATATTTGGCATAAAGCTCTCGATGCTATTTTCTAAACTTACTTTCG----TTTCTAA-A----CTTCTCTTCCAGGCGATATAGACAGCCTATTGAAAAAGTCAGAACAGCCAATTATACAACAA |
| droMoj3 | scaffold_6540:21520924-21521003 + | AG----------------------------------------------------------------------------------------------------------------------------------------------------------------------AACTAAT-TTCACT-------------TATCTCACTTTCCAGGCGATTTAGACAGCCTCCTCAAAAAGTCGGAAAAGCCGATAATACAACAG | |
| droGri2 | scaffold_14906:10320959-10321046 - | TC----------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGGCAT-TTAGCTAAAC--T---TTTTTCCTGACTTTCCAGGCGATATTGGCAACCTGTTAAAAACCTCCGAGGCGCCCATTATCCAACAA | |
| droWil2 | scf2_1100000004943:7722664-7722719 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCAGCCAGTACCGATAATCTATTGGAGACTTCTGAAAAGCCAATTTTGAATCAG | |
| dp5 | 2:28495164-28495219 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCAGGCAACATAGCCAACCTCTTGACCAGCTCTAAGCAACCGATTATTAAGCAA | |
| droPer2 | scaffold_6:3825638-3825693 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCAGGCAACATAGCCAACCTATTGACCAGCTCTAAGCAACCGATTATTAAGCAA | |
| droAna3 | scaffold_13340:12156164-12156225 - | C------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCTATTTTAGGCAGTATGTCCAACCTGTTGGAATCGTCAAAGGAACCGATAATTAAACAG | |
| droKik1 | scf7180000302650:415164-415248 + | AT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTACTTTAT----TTATCAA-ATTTATAACAATTCCAGGAAGAATGAGCAACTTGCTGGAGAGTTCCAAGCAGCCGATAATGAAGCAG | |
| droEle1 | scf7180000491008:2726427-2726508 - | CT----------------------------------------------------------------------------------------------------------------------------------------------------------------------TATCCTA---------TTTGC---GTTTCCCTACAACTTTAGGAAGAATGGCCAACTTATTGGAATCATCAAAAAAGCCTATTATCGAACAG | |
| droRho1 | scf7180000760575:18775-18883 - | ATATTAGT----------------------------------------------------------------------------TTCTTAATTCAAGA-----------------------------------------------------------------------AAATAC-TTACCTTGTTTAC---TTTTTCTTAAAATTGAAGGAAGAATGGCAAACTTATTGGAATCATCAAAAAGACCTATAATCGAACAA | |
| droEug1 | scf7180000409797:600889-600944 + | T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCAGAAAGAATGGCCAACCTATTGGAATCATCCAAGGAGCCAATAATCAAGCAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/16/2015 at 08:16 PM