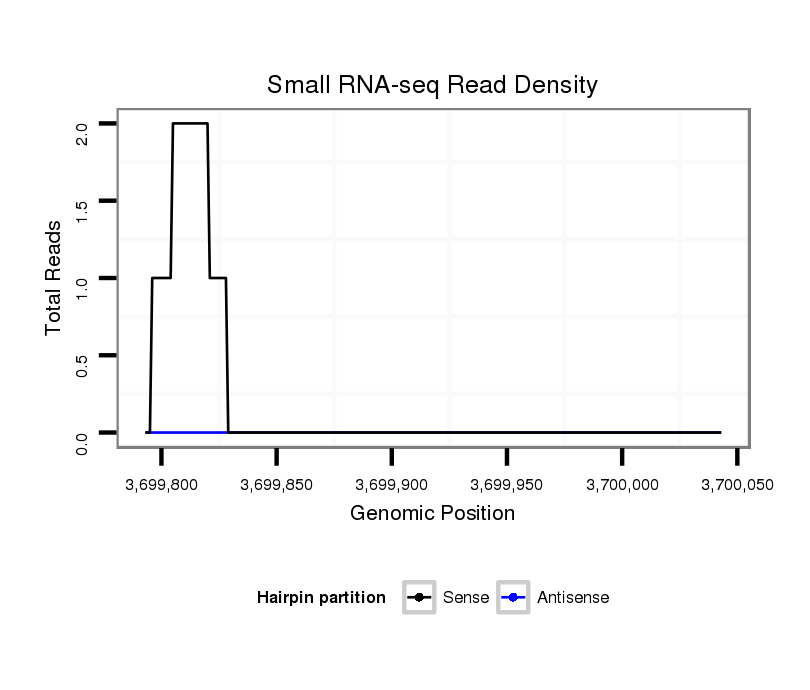
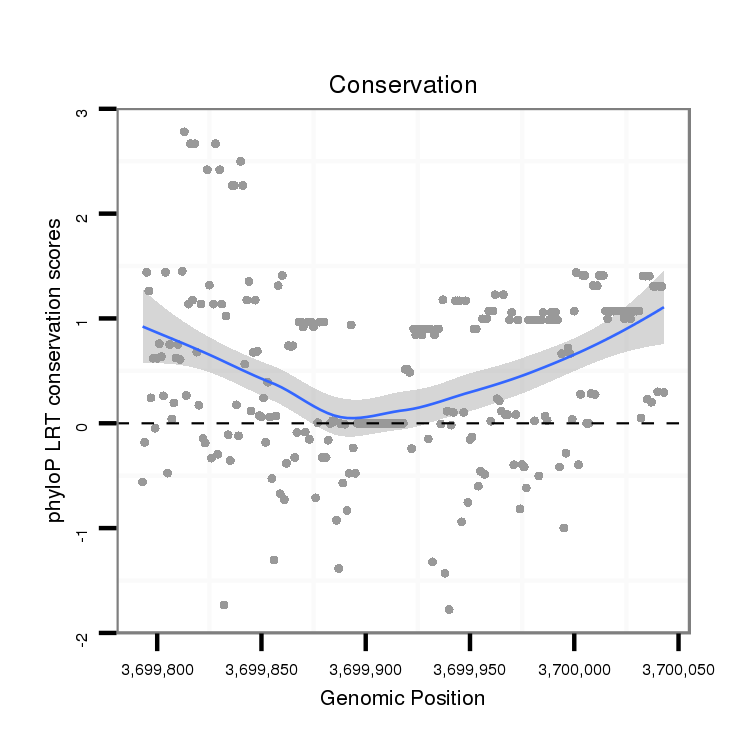
ID:dvi_1552 |
Coordinate:scaffold_12723:3699843-3699993 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_2846:21]; CDS [Dvir\GJ18290-cds]; intron [Dvir\GJ18290-in]
| Name | Class | Family | Strand |
| (TTG)n | Simple_repeat | Simple_repeat | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATTGGCGCAGCTCAAGGAGTTTGTCAACGTGCAGCTGCCTCAGGGTCTGGGTAAGTGCAGTTCCCCGTACACATATTGCAATGCTTTTTGTTCCCCGAAATCACCATTTGTTATACACTGTTTACATTGTTGTTGTTGTTGTTGTTGTGTAAAGAACCCAACACTGAATGTCAGTTCTGTGTATTGCGGCTCACTGACTCTGCTAGCTGTGTTGGTGCCTTATGAAAAAATCATGTTTATTTTTGGCAGCA **************************************************...(((.((((...((((((((((((.((((((..(((((.....)))))....)).)))).))).((((...(((((..(((((..(((.((........)).))).)))))...)))))))))..)))))...))))...)))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060681 Argx9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
SRR060689 160x9_testes_total |
SRR060658 140_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
M047 female body |
GSM1528803 follicle cells |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...GGCGCAGCTCAAGGAGTTTGTCAAC............................................................................................................................................................................................................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............CAAGGAGTTTGTCAACGTGCAGCT....................................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................AGTTCGTGAACGTGCAGCT....................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................GTTCGTGAACGTGCAGCT....................................................................................................................................................................................................................... | 18 | 2 | 6 | 0.50 | 3 | 0 | 0 | 0 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................CTATTTGTTCCCCGAATTTAC................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................GTTGGTGTTGTTGCCGTTGTGTAAA.................................................................................................. | 25 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................TTAGATGTGTTGGTTCCTTA............................. | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................................................................CTAACACTCAAGGTCAGTT........................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................CTGGGTAAGTGAAGTTTC........................................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................TTCTTGTTCCCCGAA........................................................................................................................................................ | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TAACCGCGTCGAGTTCCTCAAACAGTTGCACGTCGACGGAGTCCCAGACCCATTCACGTCAAGGGGCATGTGTATAACGTTACGAAAAACAAGGGGCTTTAGTGGTAAACAATATGTGACAAATGTAACAACAACAACAACAACAACACATTTCTTGGGTTGTGACTTACAGTCAAGACACATAACGCCGAGTGACTGAGACGATCGACACAACCACGGAATACTTTTTTAGTACAAATAAAAACCGTCGT
**************************************************...(((.((((...((((((((((((.((((((..(((((.....)))))....)).)))).))).((((...(((((..(((((..(((.((........)).))).)))))...)))))))))..)))))...))))...)))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
M028 head |
SRR060666 160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
V053 head |
V116 male body |
SRR060672 9x160_females_carcasses_total |
SRR1106721 embryo_10-12h |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
M061 embryo |
SRR060685 9xArg_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................................................................CGGAATACTTCTTTGGTAC................ | 19 | 2 | 2 | 15.00 | 30 | 19 | 5 | 2 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CGGAATACTTCTTTGGTA................. | 18 | 2 | 6 | 3.00 | 18 | 11 | 2 | 1 | 0 | 0 | 1 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CGGAATACTTCTTTTGTAC................ | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....CCGTTGAGTTCCTCAAACACT................................................................................................................................................................................................................................. | 21 | 3 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................AAAATCAAGGGGATTTACTGG.................................................................................................................................................. | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................AAATCAAGGGGATTTACTGG.................................................................................................................................................. | 20 | 3 | 7 | 0.29 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................................ACATAACGCCGTGTAATTGAGA.................................................. | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................CAACATTACATTTCCTGGGT........................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................................AGGGGCTTTGGTGGGAAACG............................................................................................................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................AAAAACAAGGAGATTTACTGGT................................................................................................................................................. | 22 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................TTAGTGGTCAACAGTATTTG..................................................................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................................................................................AACAACAACAACAACAACAACACAT.................................................................................................... | 25 | 0 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................................................................................CGGAATACTTCTTGGGTAC................ | 19 | 3 | 17 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................AGACGATCGAGCCAACGAC.................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CGGAATACTTCTTTGCTAC................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:3699793-3700043 + | dvi_1552 | ATTGGCGCAGCTCAAGGAGTTTGTCAACGTGCAGCTGCCTCAGGGTCTGGGTAAGTGCAGTT------------CC--CC-GTA--------------------CACATATTGCA---------------ATGCTTTTTG---------------------------------------------------------------TT--CCCCGAAATCACCATTTGTTATACACTGTTTACATTGTTGTTGTTGTTGTTGTTGTGTAAAGAACCCAA-CACT--G--AAT----------------------------------------------------GTCAGTTCTGTGTATTGCGGCTCACTGACTCTGC-------------------------------------TAGCT--------------------GTGTTGGTGCCTTATGAAAAAATCATGTTTATTTTTGGCAGCA |
| droMoj3 | scaffold_6500:15091943-15092187 - | ATTGGCGCAGCTCAAGGAGTTTGTCAACGTGCAGCTGCCGCAGGGCTTGGGTAAGTGCGCTACTTTTCACAATTTGCTCCATTTTTTATAT------------------------------------------------------------------------------------------GTTGTTGTTGTTGCCAACAATGCCAACAA---------------------------------------------------------------------CA--GCCACT----------------------------------------------------GTCAGTTCTATGCGCTGCGTCACACTGACTCCGACTCCGACTCCGACTTCAACTCCAACTCCGACTCCGCTCAGCT--------------------GAGTT--GGCCTTATGAAAAAATCATGTTTACTTTTGGCAGCA | |
| droGri2 | scaffold_15252:3934394-3934683 - | ATTGGCACAGCTCAGGGAGTTTGTTAGAGATCAGCTGCCACTTGGTCTGGGTAAGTGCACAA------------TTCACCCTTA------------------------CATTGCAGGCGCCTTTTTGTTTGTGTTTCTCAATTTCCCAAAGTTTTTTGTTGTTCTTGTTTTTGTTATTTCAA-----------------------------------------------------------TTGCTGTTGTTATTGTTGTTGCGTAAACAAACCAAGAGCT--G--ACT----------------------------------------------------GTCAGTTCTGTGCACCGCGGCTCACTGACTCCGT-------------------------------------CAGTTTGCTATCTAGCTTCATATGCGAGTT--TGCCTTATGAAAAAATCATGTTTATTTTTGGCAGCA | |
| droWil2 | scf2_1100000004945:205524-205752 - | dwi_3553 | GTTGGCGCAACTCAAGGAGTTTGTCAATGTGCATCTGCCGCAGGGTTTGGGTAAGCAAAAAC------------GTTATCTCTCCTCG---------------------------------------------C-----------------------------------------------TCTTCT-----------------------------------------------------------------------CTCATGTAAATAACAAAA-A-CCATGCTACTGACAGACTGACTGACGGACAGACTGACTGACGGACCGACAGAGCGACAGTTAGTTGG----------------------------------------------AATGGAGGACTGACTCT------CT--------------------GTGTT--TGCCTTATGAAAAAATCATGTTTATTTTTGGCAGCA |
| dp5 | 4_group4:6390411-6390507 - | ATTGTTGTTGTTG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGTTGTTGTTGTTGTTGCCCAAACGAAA---------------------------------------------TCAATTCGACAAAGTTAAATAGAGACGGAACTAAGGGTGGCGGCCCATGGACTCT----------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_22:97710-97796 - | TTCCACCCACACACGTACGCTTTTCATTTCCCCAATACA----------AACGCACAC--------------------------------------------------------------------------------------------------------ACACACACATGTATATTTTG--------------------------------------------------------------TTGTTGTTGTCGTTGTCGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12916:14004916-14004977 - | GCTGGCGCAGCTGAAGGAGTTCGTCAACGGCCACCTTCCTCTTGGCCTGGGTGAGTTTGGTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396535:963293-963355 - | GCTGGCGCAGCTGAAGGAGTTCGTCAACGGCCACCTACCTCTCGGCCTGGGTGAGTTTGGTT------------C--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302408:963608-963669 + | GCTGGCCCAGCTGAAGGAGTTCGTCAACGTGCACCTGCCGCTCGGTCTGGGTAAGTGTGATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453858:1411228-1411297 - | TTTGTTGTTGTTGTTCTTTTT----------------------------------------------------------------------------------------------------------------------------------------------------------------GTTGTTGTTGTTGCTGTTGTTGCTGTTTGTTGTTTTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------G-------------------------------TTGTTGCGGCT | |
| droEle1 | scf7180000491186:3247354-3247415 - | GCTGGCGCAGCTGAAGGAGTTTGTCAACGGGCACCTTCCCCTCGGTTTGGGTGAGTGCGATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779234:2510-2573 + | GATGGCGCAGCTGAAGGAGTTTGTCAACGGGCATCTTCCTGGAGGTTTGGGTGAGTGCGATT------------TC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000301468:300778-300839 - | GCTGGCGCAGCTCAAGGAGTTCGTCAACGGGCACCTCCCCCTCGGTTTGGGTGAGTGCGATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415667:305866-306005 + | GTTGGCGCAGCTGAAGGAGTTTGTCAACGGGCACCTTCCTCACGGTCTGGGTGAGTGCGATT------------TTCTTCTTTTTCTTGGCGTTTGGATTAAACAACATCTTGAA---------------GTGGCTTTTG---------------------------------------------------------------CTGTCCTTGGGATCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAATT--TGTCTTA----------------------------- | |
| droEug1 | scf7180000409554:4742909-4742970 + | GTTGGCTCAGCTGAGAGATTTTGTCAACGGGCATCTTCCTCTCGGTTTGGGTGAGTGCGATT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr2L:10183444-10183499 - | ATTGGCGCAGCTGAAGGAGTTTGTCAACGGCCACCTTCCACTCGGTTTGGGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 2l:9861141-9861196 - | GTTGGCGCAGCTGAAGGAGTTTGTCAACGGGCACCTTCCACTCGGTTTGGGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_3:5608744-5608799 - | GTTGGCGCAGCTGAAGGAGTTTGTCAACGGGCACCTTCCACTCGGTTTGGGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2L:12856112-12856167 - | GTTGGCGCAGCTCAAGGAGTTTGTCAACGGGCACCTTCCACTCGGTTTGGGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4929:10788649-10788704 - | ATTGGCGCAGCTAAAGGAGTTTGTCAACGGGCACCTTCCACTCGGCTTGCGTACGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 07:47 PM