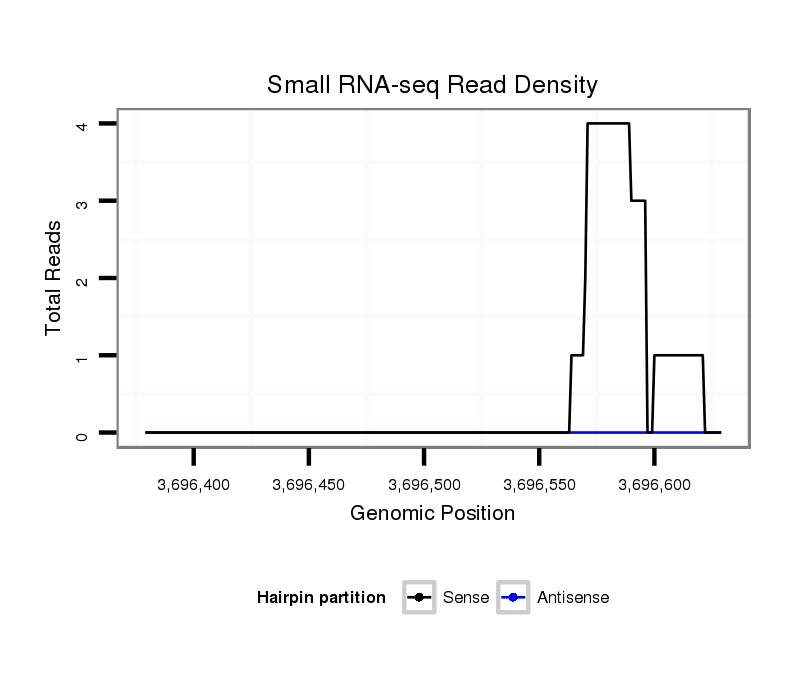
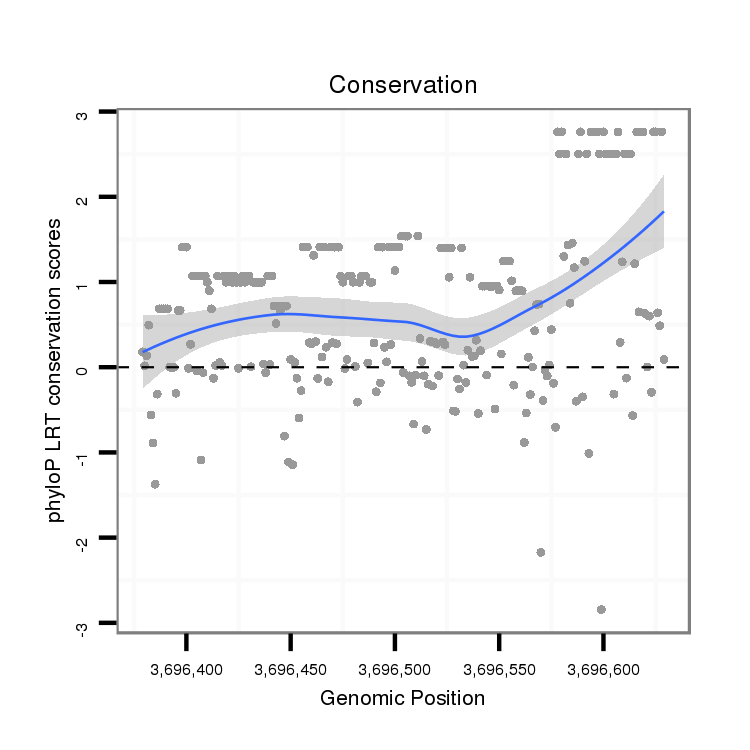
ID:dvi_1550 |
Coordinate:scaffold_12723:3696429-3696579 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_2846:20]; CDS [Dvir\GJ18290-cds]; intron [Dvir\GJ18290-in]
| Name | Class | Family | Strand |
| (TTTTTG)n | Simple_repeat | Simple_repeat | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TTTTTTTTTTTTTTTTAGCTTTCATTTTTGAGAACATTTTCAACTGACAGTTGCGGGCACAAAATACTGTGTCTGCGAAAAGGTCACAATTCTCATGGTTTGCTGATTTGGTTTTAAATTTAATATAAGCGTTTGTTTACATTTATTCATACCATCCGTTTGTTTGTTTTTGCTTTTGTTTTTGCTCTTCTTCTGGTGCAGACCAAAGCGTTCCAATTTCCTGGCCACATCGCGGCGACATCAGCTTCGAG **************************************************(((((((((((......)))))))))))..(((((((((.((....)).))).))))))............((((.(((((.......(((...................))).......))))).)))).((((.((......)).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060683 160_testes_total |
SRR060657 140_testes_total |
SRR060678 9x140_testes_total |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
SRR060689 160x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
V047 embryo |
SRR060668 160x9_males_carcasses_total |
M061 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................................................CTGGTGCAGACCAAAGCGTTCCAATT................................. | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................TCTGGTGCAGACCAAAGCGTTCCAATT................................. | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................TGGCCACATCGCGGCGACATCA........ | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................TCTTCTTCTGGTGCAGACCAAAGCGT........................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................TCTTCCTCTGGTGCAGACCAAAGCGT........................................ | 26 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................TCCAACTGACAGTTGCGGGCACAAAA........................................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................ATGGTTTGGTGATTTGGT........................................................................................................................................... | 18 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................ATGGGTTGCTGATTTGTTTT......................................................................................................................................... | 20 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................CACCATCCGTTTGATTGTT................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................................................TTGATTTTGATTTTGCTCTTC............................................................. | 21 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................TGAGATCATTTTGATCTGAC........................................................................................................................................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ......................................................................................................................................................................................................CACACCAAAGGGATCCAAT.................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
AAAAAAAAAAAAAAAATCGAAAGTAAAAACTCTTGTAAAAGTTGACTGTCAACGCCCGTGTTTTATGACACAGACGCTTTTCCAGTGTTAAGAGTACCAAACGACTAAACCAAAATTTAAATTATATTCGCAAACAAATGTAAATAAGTATGGTAGGCAAACAAACAAAAACGAAAACAAAAACGAGAAGAAGACCACGTCTGGTTTCGCAAGGTTAAAGGACCGGTGTAGCGCCGCTGTAGTCGAAGCTC
**************************************************(((((((((((......)))))))))))..(((((((((.((....)).))).))))))............((((.(((((.......(((...................))).......))))).)))).((((.((......)).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
V116 male body |
SRR060665 9_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060663 160_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
SRR060671 9x160_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060664 9_males_carcasses_total |
M061 embryo |
SRR060669 160x9_females_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
V053 head |
SRR060661 160x9_0-2h_embryos_total |
M028 head |
SRR060675 140x9_ovaries_total |
M027 male body |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
V047 embryo |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................CCCGTGTTTTGTGGTACAGAC................................................................................................................................................................................ | 21 | 3 | 2 | 45.00 | 90 | 12 | 9 | 4 | 8 | 4 | 6 | 8 | 4 | 5 | 2 | 3 | 5 | 3 | 3 | 3 | 3 | 1 | 1 | 0 | 2 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 |
| ......................................................CCCGTGTTTTGTGGTACAGA................................................................................................................................................................................. | 20 | 3 | 5 | 25.40 | 127 | 15 | 2 | 24 | 14 | 7 | 4 | 1 | 5 | 3 | 11 | 3 | 3 | 5 | 5 | 4 | 4 | 3 | 3 | 4 | 0 | 2 | 1 | 0 | 0 | 0 | 2 | 1 | 1 |
| .......................................................CCGTGTTTTGTGGTACAGAC................................................................................................................................................................................ | 20 | 3 | 9 | 13.22 | 119 | 18 | 25 | 2 | 5 | 16 | 9 | 0 | 6 | 4 | 3 | 9 | 3 | 2 | 1 | 2 | 3 | 2 | 2 | 2 | 2 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 |
| .......................................................CCGTGTTTTGTGGCACAGA................................................................................................................................................................................. | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................CCGTGTTTTGTGGTACAGA................................................................................................................................................................................. | 19 | 3 | 20 | 1.00 | 20 | 0 | 5 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 8 | 0 | 4 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| AAAAAAAAAAAAAAAATCGAAA..................................................................................................................................................................................................................................... | 22 | 0 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................GCCCGTGTTTTGTGGTACAGAC................................................................................................................................................................................ | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................CCGTGTTTTGTGGCACAG.................................................................................................................................................................................. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....AAAAAAAAAAATCGGAAGT................................................................................................................................................................................................................................... | 19 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................AAAACGTAAAAATAAACGAGAAGA............................................................ | 24 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................AAATGTAAATGAGTAAGGT................................................................................................. | 19 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................ATCGCAAGGTTAAAGTATCGG......................... | 21 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................CAATGTTGAGAGTACCACA...................................................................................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .AAAAAAAAAAAAAAATCGAA...................................................................................................................................................................................................................................... | 20 | 0 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................ACAAAAACGAAAACAAAA..................................................................... | 18 | 0 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:3696379-3696629 + | dvi_1550 | TTTTTTTTTTTTTTTTAGCTTT-----------------CATTTTTGAG------AACATT-TTC-AACTGACAGTT-GCGGGCACAAAATA-------CTGT----GT--------------------------------------------------------CTGCGAAAAGGTCA--CAATTCTCATGGTTTGCTGATTTGGTTTT-AAAT-TT---AATA------------TAAGCGTTTGTTTACATTTATT-CATA-------CCATC---C----G--------TTTGTTTGTTTTTG------CTTTTGT----------------------TTTTGCTCTTCTTCTGGTGCAGACCAAAGCGTTCCAATTTCCTGGCCACATCGCGGCGACATCAGCTTCGAG |
| droMoj3 | scaffold_6500:15095571-15095904 - | TTTTTGGC-----------TTT-----------------CATT-TCGAGTTTTCAA--ATA-TTC-AACTGACAGTT-GCGGGCGCAAAA-A-------CTCT----CTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTATTTAGTTGAGCAAAAAGGTCACACAATTTTCATGCTTTGTCGATTCGGGCTCTAAAT-TT---AATA------------TAAGCGTTTGTTTGCAGTTATT-CAGT-------GCATT---C----GCTTCGCTCTTTGTTTGTTTATGTGTTGTCTTTT--ATTTTATATTTATTTCTTTTCTTTTTCTTTTTTT-TCTCGTCAGACCAAAGCGTTCCGATTTCCTGGCCACAGCGAGGTTACATCAGCTTCGAG | |
| droGri2 | scaffold_15252:3936876-3937110 - | TTTCTGGC-----------TTT-----------------TATTTTGGAG--------CGTTGCTC-AACTGCCAGTT-TCGGGCGCAAAATACCAAATACTCTCTATAT--------------------------------------------------------ATGGGAAAGGGTCA--CAAATTTCATGCTTTGTCGATTTGGTTTTAAAAT-TTAATAATA------------AAAGCATTTGTCCACATTTATTTCAT--------CTAATTTTCCTTTG--------TTTAT------------------------------------------------------TTGTTTGGGTAGACCAAACCGTTCCAATTTCGTGGCCACAACGCGGCGACATCAGTTTCCAA | |
| droWil2 | scf2_1100000004945:209208-209472 - | TTCATATTTTTTT---TGCTTTGCTTTTTTGGTTTTGTTTGTTCTTCAG----C-AAAATT-TTCAAACTGACAGTTTGCGGGCGGACA-----------------TAA--------------------------------------------------------ATTTTAAAAGGTTA--CAATTTTCATGCATTGTTGATTTGGTATTAAA-TTATT---ATATACATACATACTAAAGAATTTACACAGATATAAT--AATGGATGATCCATC---C---------------------------------CTTTTGT----------------------TTTTCCAT-ATACGACATGAAGACCGGAGTGTACCGATTTCCTGGCCACAACGCGGAGACATCAGCTTCGAA | |
| dp5 | 4_group3:24081-24153 + | TTTTTAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGC------------------------------------------------------TTGCTTTTGTAGACCAGAGCGTTCCCATTTCTTGGCCGCAAAGAGGCGACATCAGCTTCCAA | |
| droPer2 | scaffold_1:1507374-1507446 + | TTTTTAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGC------------------------------------------------------TTGCTTTTGTAGACCAGAGCGTTCCCATTTCTTGGCCGCAAAGAGGCGACATCAGCTTCCAA | |
| droAna3 | scaffold_12916:14006070-14006121 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGACCAAAGTGTACCAATTTCGTGGCCTCAGAGAGGCGACATCAATTTTGAT | |
| droBip1 | scf7180000396535:964450-964514 - | TTTTCTTT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTTAGACCAAAGTGTACCAATTTCTTGGCCTCAGAGGGGCGACATCAATTTTGAT | |
| droKik1 | scf7180000302408:962638-962704 + | CTCTACAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCTTTCAGACCAAAGTGTTCCGATTTCTTGGCCACAAAGGGGAGACATCAGCTTCGAT | |
| droFic1 | scf7180000453838:402554-402605 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATCAAAGTGTTCCGATTTCATGGCCGCAAAGAGGAGATATCCACTTCGAT | |
| droEle1 | scf7180000491186:3248310-3248458 - | AGTTCAAC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------A------------TAACTTGTTAAGTATGTCTGTT-CATA-------TAA-C---A----G--------AC--TTTTTTTATGTGTTTTTTTTTGGAATTTAAAACTACTTT-----------ATCCTTTATACTTTCAGATCAAAGCGTACCCATTTCTTGGCCGCAAAGAGGAGATATCAATTTCGAT | |
| droRho1 | scf7180000779234:1557-1629 + | TTTTATTC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CA------------------------------------------------------TTTCTACTTCCAGATCAAAGCGTACCCATTTCCTGGCCGCAAAGAGGAGACATAAACTTCGAT | |
| droBia1 | scf7180000301468:301722-301776 - | T-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGATCAAAGCGTTCCCATTTCCTGGCCTCAAAGAGGGGATATCCATTTCGAT | |
| droTak1 | scf7180000415667:304830-304955 + | GGTTTTAT-----------TTT-----------------TA------------------------------------------------------------------------------------------------------------------------------ATGAAAAAGAAA--GACATTTTA----------------TTTATACCT-TT---AATA------------TAAACGCTG-----------------------ATT-----------------------CT------------------------------------------------------TTTATACTTTTAGATCAAAGCGTACCCATTTCGTGGCCGCAAAGAGGAGATATCCACTTTGAT | |
| droEug1 | scf7180000409554:4741950-4742015 + | TATTTTTC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACTTTCAGATCAAGGAGTTCCCATTTCTTGGCCGCAAAGAGGTGATATTT-TTTCGAC | |
| droSim2 | 2l:9861978-9862030 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATCAAAGTGTGCCAATTTCGTGGCCACAAAGAGGTGATATCAATTTCGAT | |
| droSec2 | scaffold_3:5609579-5609631 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATCAAAGTGTGCCAATTTCGTGGCCACAAAGAGGTGATATCAATTTCGAT | |
| droYak3 | 2L:12857009-12857077 - | TACTGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTCCACTTTTAGATCAAAGTGTGCCAATTTCGTGGCCACAAAGAGGTGATATCCATTTCGAT | |
| droEre2 | scaffold_4929:10789507-10789559 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGATCAAAGTGTGGCAATTTCGTGGCCACAAAGAGGTGATATCAATTTCGAT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 11:13 AM