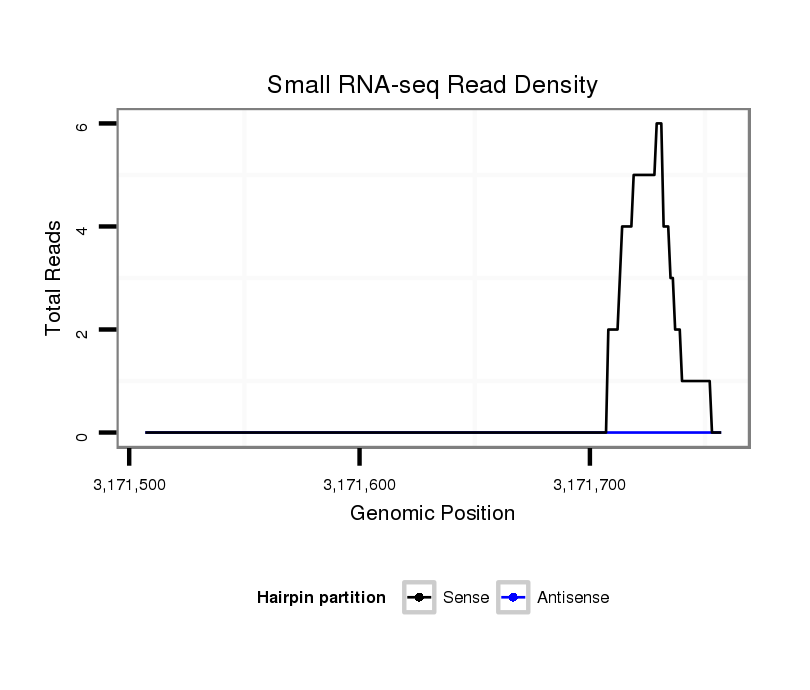
ID:dvi_15349 |
Coordinate:scaffold_13047:3171557-3171707 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\osk-cds]; exon [dvir_GLEANR_9119:3]; intron [Dvir\osk-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGAAAGATACTATTGTTTTTCTATATAAAAAAAGAAAATTTCCAAATGTGGAGAAAACTCTACCTTTGAAAAGGTTGCCAGCGGATAAAACTTTGGCGTTGCCACCTTTTATACAGCACAACCGTTGCCGACACAGGAGAACTTAAGGAAATAGAATTTTTATTGATTTGATCAAACCATTTTTGTGCACTTCATTTGCAGCAACAATCAGTATCAGCTCCCTCAGCTCAATGCTGCCAAGAATAACATCC **************************************************..(((((.((((((((..(((((((.((..((.((.......)).))...)).)))))))...((((......))))((......)).......))))...)))).)))))..(((..((..((((.....))))..))..))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060680 9xArg_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
V053 head |
SRR1106729 mixed whole adult body |
SRR060684 140x9_0-2h_embryos_total |
M027 male body |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................................................................................CAACAATCAGTATCAGCTCCCTCA.......................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................CAACAATCAGTATCAGCTCCCTCAGCT....................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................ATCAGTATCAGCTCCCTCA.......................... | 19 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................TTTTATTGCGTTGATCAAACC......................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................TCAGTATCAGCTCCCTCAGCTCA..................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................TCAGCTCAATGCTGCCAAGAATAA..... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................CAGGAGAACGTAAAGAAATAGAATTTT........................................................................................... | 27 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................ATCAGCTCCCTCAGCTCAATG.................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................CCGACACAGGTGGACT............................................................................................................ | 16 | 2 | 20 | 0.45 | 9 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 9 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................AGGAGAAATTACGGGAATAGA................................................................................................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GAAACAATCAGGATTAGCTC............................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................CGGATGAAACTTTGGGGGT....................................................................................................................................................... | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................TTTGAAACGGTTGCC............................................................................................................................................................................ | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................AAAAATTTGGCGGTACCAC.................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................................................................................................GATTTGAGCAAACCATTTAC................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
ACTTTCTATGATAACAAAAAGATATATTTTTTTCTTTTAAAGGTTTACACCTCTTTTGAGATGGAAACTTTTCCAACGGTCGCCTATTTTGAAACCGCAACGGTGGAAAATATGTCGTGTTGGCAACGGCTGTGTCCTCTTGAATTCCTTTATCTTAAAAATAACTAAACTAGTTTGGTAAAAACACGTGAAGTAAACGTCGTTGTTAGTCATAGTCGAGGGAGTCGAGTTACGACGGTTCTTATTGTAGG
**************************************************..(((((.((((((((..(((((((.((..((.((.......)).))...)).)))))))...((((......))))((......)).......))))...)))).)))))..(((..((..((((.....))))..))..))).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060672 9x160_females_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060660 Argentina_ovaries_total |
V116 male body |
SRR060669 160x9_females_carcasses_total |
M027 male body |
SRR060665 9_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................TCTTCTAAAGGTTTACATCTCTGT................................................................................................................................................................................................... | 24 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................ACGACGGTTCTTATTATAGG | 20 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................AAAAGATATATTTTGTTC......................................................................................................................................................................................................................... | 18 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................AAATTGCAACGGTGGAAAA............................................................................................................................................. | 19 | 2 | 15 | 0.13 | 2 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................CCTCAACGGTGGAAGAGAT.......................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ............................................................................................................................................................................................................................GGAGACGAGTTACAAGGGTT........... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................TACGAGCTTGGTAAAAACAC................................................................ | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................AGTCAGCGTCGTTGTTAG.......................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:3171507-3171757 + | dvi_15349 | TGAAAGATACTATTGTTTTTCTATATAAAAAAAGAAAATTTCCAAATGTGGAGAAAACTCTACCTTTGAAAAGGTTGCCAGCGGATAAAACTTTGGCGTTGCCACCTTTTATACAGCACAACCGTTGCCGACACAGGAGAACTTAAGGAAATAGAATTTTTATTGATTTGATCAAACCATTTTTGTGCACTTCATTTGCAGCAACAATCAGTATCAGCTCCCTCAGCTCAATGCTGCC------------------------------------------AAG------------AATAACATCC |
| droMoj3 | scaffold_6540:20213726-20213798 + | T----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCATTTGCAGCAACAATCAGTATCAGCTACCGCAGCCCAATGCTCTC------------------------------------------AAGAACAGCAACAGCAGCAACATCT | |
| droGri2 | scaffold_15074:7545240-7545338 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTACAGCAACAATCAATATCAGCTACCGCAGCTGGCAGCTCTGAGCAACCACAACAACAACACTAACAACAACAACAACAGCATCAGA------------AACAACATCT | |
| droAna3 | scaffold_13340:3512458-3512513 + | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATTTACGTCGATACTCCGGATCGGCAGACCCAGGTTACAGATCCA------------------------------------------G--------------AGCAACAT-T | |
| droBip1 | scf7180000396712:841143-841193 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATTTACATCGATACTCCGGATCAGCCCACCCAGGTGACAGATCCA------------------------------------------G--------------------AGCT | |
| droRho1 | scf7180000779210:40675-40728 - | ------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATCTACAGCGATCCTCCGGAGGAGCGGACTCAGTTCGCAGTTCCC------------------------------------------GAG-----------------CTTCC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
Generated: 05/16/2015 at 10:42 PM