
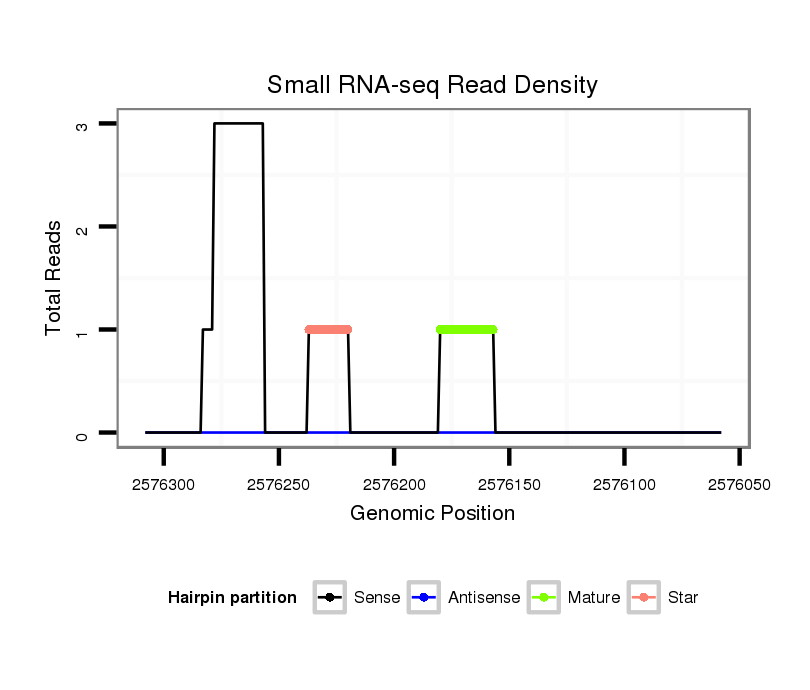
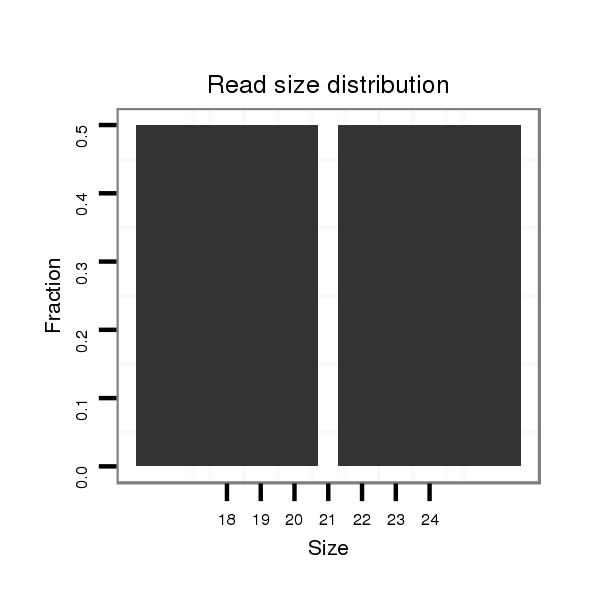
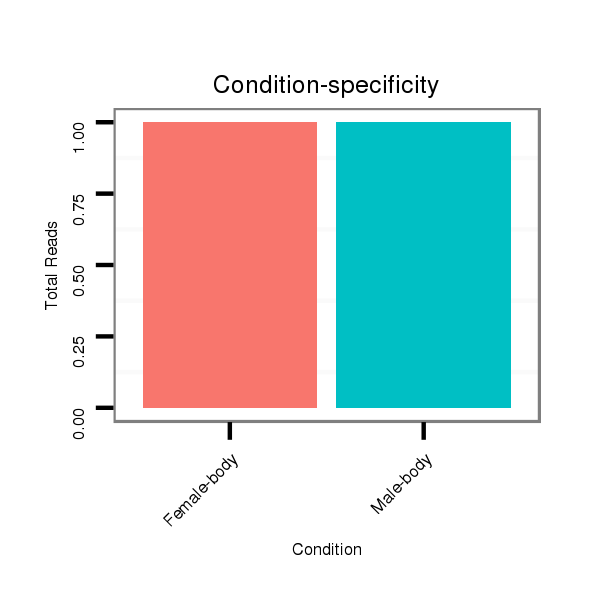

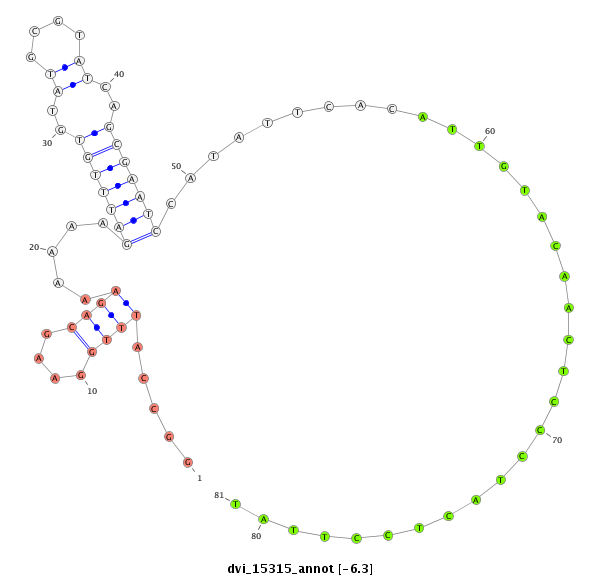
ID:dvi_15315 |
Coordinate:scaffold_13047:2576108-2576258 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
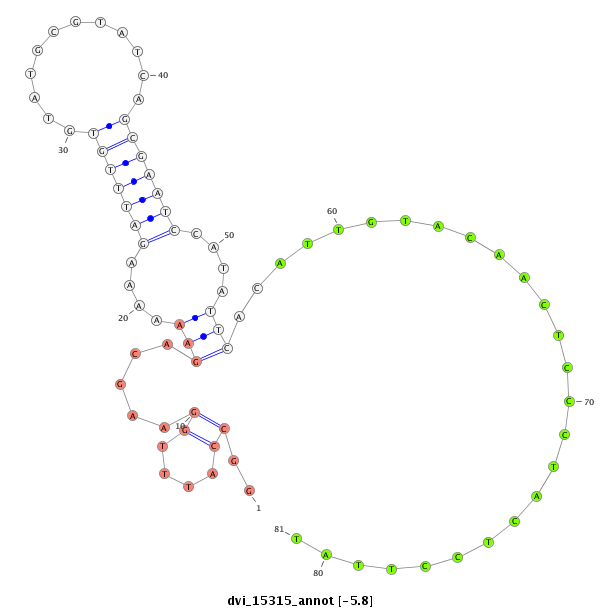
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -6.4 | -6.3 | -5.9 | -5.8 |
 |
 |
 |
 |
exon [dvir_GLEANR_8797:12]; CDS [Dvir\GJ23448-cds]; intron [Dvir\GJ23448-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CACGCTGAGCGCTAGTCTAAATGCCCAAAACAATGCTGTAGCAGCGTCAGGTAATAAACAGGGAGTAAAAAGGCCATTTGGAAGCAGAAAAAAGATTTGTGTATGCGTATCAGCGAATCCATATTCACATTGTACAACTCCCTACTCCTTATTCCTTATTTCATGCCACTCTCTAATTCCCAATTTGTCAGCAATCAAAAAGAGGTTTCTTTGTGAAATTCTGTTTAAAAGTGCCCCAAATAAGTTTATTT ***********************************************************************....((.........((.........(((((((((((....)))....))))..)))).........))..........))*************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | GSM1528803 follicle cells |
M047 female body |
SRR060678 9x140_testes_total |
SRR060687 9_0-2h_embryos_total |
M061 embryo |
SRR060671 9x160_males_carcasses_total |
SRR060680 9xArg_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060679 140x9_testes_total |
V053 head |
V116 male body |
SRR060667 160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................CAAAACAATGCTGTAGCAGCA............................................................................................................................................................................................................. | 21 | 1 | 1 | 4.00 | 4 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................CAATGCTGTAGCAGCGTCAGGT....................................................................................................................................................................................................... | 22 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AATGCTGTAGCAGCGTCAGGTATGCA.................................................................................................................................................................................................. | 26 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................ATTGTACAACTCCCTACTCCTTAT................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................TCTTGCAACTCTCCAATTCCCAATT.................................................................. | 25 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................GGCCATTTGGAAGCAGAA.................................................................................................................................................................. | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................CAAAGAGAGGTTTCTCTGCG.................................... | 20 | 3 | 3 | 1.00 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................TCTTGCAACTCTCCAATTCCCAAT................................................................... | 24 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................CAAAACAATGCTGTAGCAGCGTCAGGT....................................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................TTCTTGCAACTCTCCAATTCCCAAT................................................................... | 25 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................AATCAAGAAGAGGTGTCTT........................................ | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................ATAAACCGGGATTAAAAAAGC................................................................................................................................................................................. | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................CAAAGAGAGGTTTCTCTGC..................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 |
| ....................................................................................................................................................................................................AAAGAGAGGTTTCTCTGCG.................................... | 19 | 3 | 14 | 0.14 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................ATTTGGAAGCGGAGAAAGGA............................................................................................................................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................CAAGTTCTCAGCATTCAAAAA.................................................. | 21 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..................................................................................................................................................................................................CCAAAGAGAGGTTTCTCTG...................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
GTGCGACTCGCGATCAGATTTACGGGTTTTGTTACGACATCGTCGCAGTCCATTATTTGTCCCTCATTTTTCCGGTAAACCTTCGTCTTTTTTCTAAACACATACGCATAGTCGCTTAGGTATAAGTGTAACATGTTGAGGGATGAGGAATAAGGAATAAAGTACGGTGAGAGATTAAGGGTTAAACAGTCGTTAGTTTTTCTCCAAAGAAACACTTTAAGACAAATTTTCACGGGGTTTATTCAAATAAA
***************************************************************************************************....((.........((.........(((((((((((....)))....))))..)))).........))..........))*********************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060656 9x160_ovaries_total |
V116 male body |
SRR060671 9x160_males_carcasses_total |
GSM1528803 follicle cells |
V053 head |
SRR060658 140_ovaries_total |
SRR060674 9x140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................ACGGGTCTTGTGAAGACATCG................................................................................................................................................................................................................. | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................ATCTTGACGGATGAGGACTA................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................ACGGGTCTTGTGAAGACAT................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.20 | 4 | 3 | 0 | 0 | 0 | 0 | 0 | 1 |
| .........................................................................................................................................GAGGGAAGAGGATAAAGGAAT............................................................................................. | 21 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................AGGGATAACGAATAAAGTAG...................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................GCAGTCCATGAGTTGTCCA............................................................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................GGGTTTTGTTGCGACTTAGT................................................................................................................................................................................................................ | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................TAGTCCCTCATTGTTCCTG................................................................................................................................................................................ | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................TGCTTAGGTATAAGTCCAA........................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13047:2576058-2576308 - | dvi_15315 | CACGCTGAGCGCTAGT------------CTAAATGCCCAAAACAATGCTGT---AGC---AGCGTCAGGTAATAAACAGGGAGTAAAAAGGCCATTTGGAAGCAGAAAAAAGATTTGTGTATGCGTATCAGCGAATCCATATTCACATTGTACAACTCCCTACTCCTTATTCCTTATTTCATGCCACTCTCTAATTCCCAATTTGTCAGCAATCAAAAAGAGGTTTCTTTGTGAAATTCTGTTTAA--AAGT-----------GCCCCAAATAAGTTTATTT |
| droMoj3 | scaffold_6540:19818046-19818151 - | CACGCTGAGCGCCAAT------------CTGAACAGCCAAACAAACGTTGG---AGG---GTCGGCAGGTAATTGACGAAATGCAA------CACCTGAGCACTGAGAGAGTGCATGTAAAAGTATATCA-------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15074:7142365-7142413 - | GACACTGAGCGCCAATC------------AGACTGCCCAAACTGCAGCAAC------------GTCAGGTAAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004902:10718223-10718291 - | CATGTTGAGTATTGGTAGTGGTGCCAG---------CCAAACAACTAACAATGCAAATAATCATGCAGGTAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATAAA------- | |
| dp5 | 2:27560359-27560416 - | CACGCTGAGCGCCAACAGT---------------GCACACACCACAAACGCGACACCGAATGCGGCAGGTAAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_6:2879960-2880017 - | CACGCTGAGCGCCAACAGT---------------GCACACACCACAAACGCGACACCGAATGCGGCAGGTAAT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13340:2660751-2660789 + | TCTAAGTAGCGGTGGC------------CTAAGTGCCCAGGCAACCAATGC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396119:2108-2169 + | TTTTTAAATCATTGAA---G---AAAGATTTATTTTGTAATTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGAGTAATTAAGA-----------GCATTAAATAAG------- | |
| droKik1 | scf7180000302650:45413-45447 + | CACACTGAGCGCCAGTAGCA---CTGGACTTAAC------------A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATG------- | |
| droFic1 | scf7180000453213:19132-19206 + | CACGTTGAGTGCCAGCAGCA---CCGGCTTGAATGTGCAGTCCACCAATGC------------GGCAGGTAATTAAAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCGGTTTAAAGG------- | |
| droEle1 | scf7180000490732:29713-29749 - | G--------CGCCAGT------------CTGCAGGCCGACAAGC---------------ATGCTGTAG-C----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGC------- | |
| droRho1 | scf7180000779643:10015-10095 + | TCTTATT----------------------CAACTATCCAATATGCAATTGC------------TAAACCTAATCTAA--------------------------------------------------------------------------------------------------------------------------------------------AAAAAGTTTCT--------------------AGAAAAGTTAAATAAATATTCTAAAAA------- | |
| droBia1 | scf7180000302402:1913429-1913494 + | CACTTTGAGTGCCAGTAGCA---CCGGCTTGAATGTACAGGCCACCAATGC------------GACAGGTAATTAGCA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAG------- | |
| droTak1 | scf7180000415013:57140-57203 + | CACATTGAGTGCCAGTAGCG---GCGGCTTGAATGTGCAGGCCACCAATGC------------AGCAGGTAATTAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAG------- | |
| droEug1 | scf7180000409804:2444115-2444167 + | CACTTTGAGTGCCAGTAGCG---TCGGCTTGAATGTGCAGGCCACCAATGC------------GGCAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr3R:16126082-16126097 + | CACATTGAGTGCCAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 3r:5179897-5179912 - | CACATTGAGTGCCAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_27:613132-613147 - | CACATTGAGTGCCAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 3R:4901081-4901096 + | CACATTGAGTGCCAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4770:5555750-5555765 - | CACATTGAGTGCCAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:03 PM