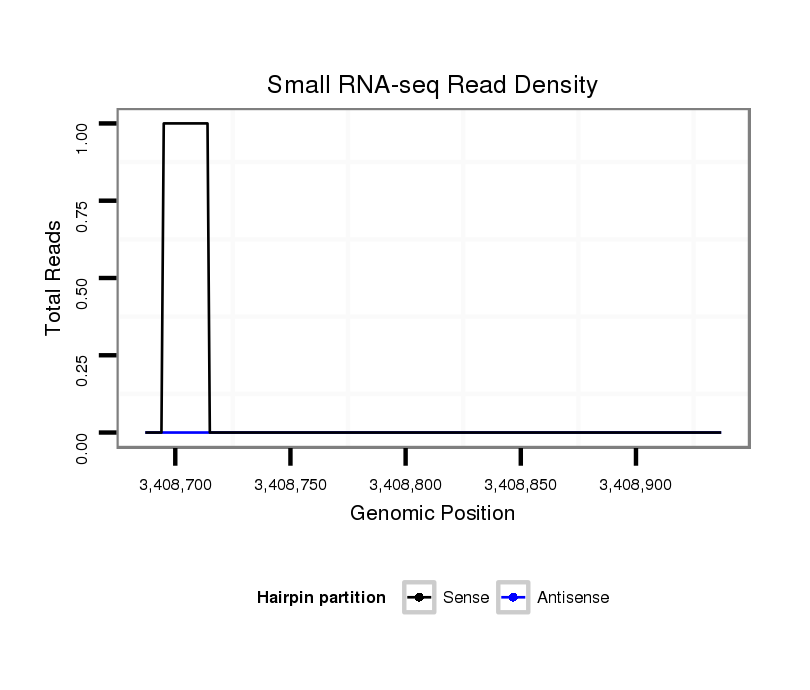
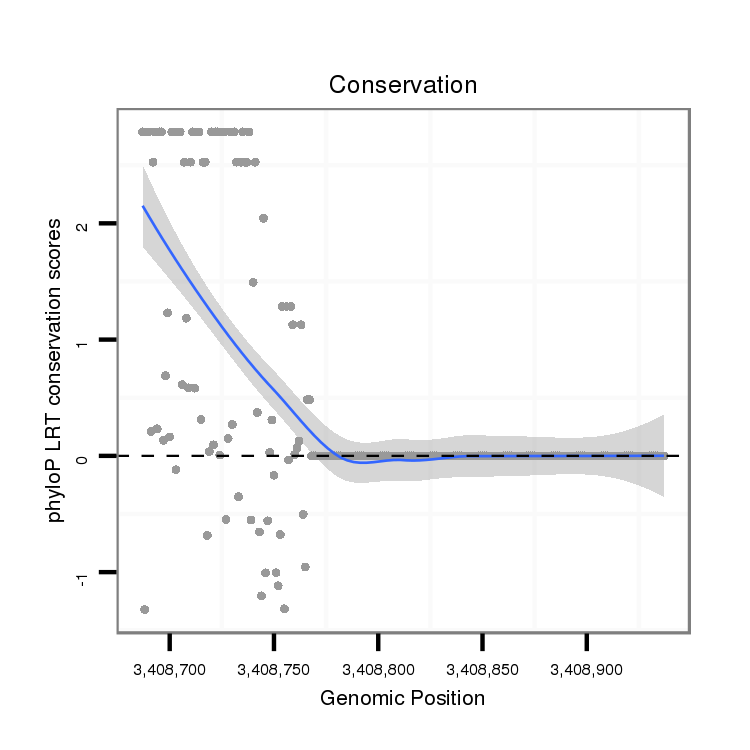
ID:dvi_1494 |
Coordinate:scaffold_12723:3408737-3408887 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_2824:1]; CDS [Dvir\GJ18267-cds]; intron [Dvir\GJ18267-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TGATTGTAATTCTCTTCTTTGCTGTCTTTGGCCTCATCATACTGACCGAGGTAAGTTCAAGAGTTGGTCTGAAGAAAAGATAGAACTACTCTCTTCGGAGTGCCGAAGATTTAAAATTCTTGTTTTATACAGGAAAAAAGTGAAAAAAAGTGTTTTATATGACAATTTTCACAGCCTAGGACTGAGAGAAGAAGACTTAGAGTTTCTTTACGAAATACGCAGAAGACAATATTTTTAAGTCGGGTCTTTGA **************************************************.((((((..((((((.((((.......))))..)))).)).(((((((....))))))).......(((((((.....)))))))....(((((((....((((......)))).))))))).........((......))..))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060683 160_testes_total |
V116 male body |
SRR060667 160_females_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ........ATTCTCTTCTTTGCTGTCTT............................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................GTTTAACAACCTAGGACTGA.................................................................. | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................AGGAAAAAAGTGAAAAAAAA..................................................................................................... | 20 | 1 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................................................CTTCTTTGCGAAATACCCAGA............................ | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................................................................................AAACAGGAAAGAAGTGAAAA......................................................................................................... | 20 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................CTTCTTTGCGAAATACCCAG............................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 |
|
ACTAACATTAAGAGAAGAAACGACAGAAACCGGAGTAGTATGACTGGCTCCATTCAAGTTCTCAACCAGACTTCTTTTCTATCTTGATGAGAGAAGCCTCACGGCTTCTAAATTTTAAGAACAAAATATGTCCTTTTTTCACTTTTTTTCACAAAATATACTGTTAAAAGTGTCGGATCCTGACTCTCTTCTTCTGAATCTCAAAGAAATGCTTTATGCGTCTTCTGTTATAAAAATTCAGCCCAGAAACT
**************************************************.((((((..((((((.((((.......))))..)))).)).(((((((....))))))).......(((((((.....)))))))....(((((((....((((......)))).))))))).........((......))..))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060687 9_0-2h_embryos_total |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
M047 female body |
SRR060660 Argentina_ovaries_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060656 9x160_ovaries_total |
SRR060659 Argentina_testes_total |
V116 male body |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...................................TATTATGAATGGCTCCGTTC.................................................................................................................................................................................................... | 20 | 3 | 6 | 6.00 | 36 | 11 | 0 | 4 | 3 | 2 | 2 | 1 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................TACCGACTCTCTTTTTCTGAAT.................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGTGGCTCCATGCATGTTC.............................................................................................................................................................................................. | 19 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................ACATAAACCGGAGTAATAGGA................................................................................................................................................................................................................ | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................ACGACAGAAAGAGGAGTAG..................................................................................................................................................................................................................... | 19 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................AAAGAAATGCTTTGTGCCTTT............................ | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .............................................................TCGACCATTCTTCTTTTCTAT......................................................................................................................................................................... | 21 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................................................................................AAGAAATGCTTTGTGCCTTT............................ | 20 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| .................AAGCGACAGAAACAGGAG........................................................................................................................................................................................................................ | 18 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................ACGACAGAAACGGGAGAACT.................................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................CGACAAGTCTTCTTTTCTAT......................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:3408687-3408937 + | dvi_1494 | TGATTGTAATTCTCTTCTTTGCTGTCTTTGGCCTCATCATACTGACCGAGGTAAGTTCAA----GA-------GTTGGTCTGAAGAAAAGATAGAACTACTCTCTTCGGAGTGCCGAAGATTTAAAATTCTTGTTTTATACAGGAAAAAAGTGAAAAAAAGTGTTTTATATGACAATTTTCACAGCCTAGGACTGAGAGAAGAAGACTTAGAGTTTCTTTACGAAATACGCAGAAGACAATATTTTTAAGTCGGGTCTTTGA |
| droMoj3 | scaffold_6500:6371796-6371876 - | TCATTGTCATTCTCTTTTTTGGTGTCTTTGGTCTCATCATATTGACCGAGGTAAGTCTAT----GA-------ATTTCTATGAAATAAGCAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_14978:40566-40642 + | TGATTGTCATCCTCTTTTTTGCTGTCTTTGGCCTCATCATTTTAACAGAGGTAAGTTTAA----GG-------GACTCTTTAAAAAAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droWil2 | scf2_1100000004521:5432373-5432429 + | TAATAGTTATCGCATTTTTTGCCGTCTTTGGATTGATCATATTGACAGAGGTAAGTT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 4_group1:2235912-2235967 + | TCATCGTCATTACCTTCTTTGCCGTCTTCGGGCTGATCATCTTGACCGAGGTGAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_5:3013688-3013743 - | TCATCGTCATTACCTTCTTTGCCGTCTTCGGGCTGATCATCTTGACCGAGGTGAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12943:2004830-2004885 - | TGATAGTCATCACCTTCTTCGCCGTGTTTGGTTTGATTATCCTGACCGAGGTGAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396728:1243482-1243537 + | TGATAGTCATCACCTTCTTTGCCGTGTTCGGGTTGATTATCCTGACCGAGGTAAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302382:593904-593967 + | TTATCGTCATCACCTTCTTCGCAGTCTTTGGATTGATTATCCTGACCGAGGTTAGTAGAA----TA-------GT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000453556:1477-1560 - | TCATCGTCATCACGTTCTTCGCCGTCTTTGGATTGATCATTTTAACCGAGGTAAGAAAAT----GGCTTTTACGAGTATATAAAAATA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droEle1 | scf7180000491338:962605-962680 - | TCATCGTCATTACATTCTTCGCCGTCTTTGGATTAATCATTCTGACAGAGGTAAGACAATTAAACA-------GTCGATTTAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000780065:5299-5354 - | TCATCGTCATCACATTCTTCGCCGTCTTTGGATTAATCATTCTGACAGAGGTTAGT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302408:2999957-3000018 + | TCATCGTCATCACCTTCTTCGCCGTCTTCGGCTTGATCATACTGACCGAGGTGAGTGCAG----AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415287:336194-336255 - | TCATCGTTATCACCTTTTTCGCCGTTTTTGGATTGATCATACTGACCGAGGTAAGTCCAG----AA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409554:39702-39760 + | TTATCGTCATCACCTTTTTCGCCGTCTTTGGATTAATAATACTGACCGAGGTTAGTCTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chr2L:13354093-13354151 - | TTATCGTCATCACCTTCTTCGCCGTCTTTGGACTAATCATTCTAACTGAGGTATGTCCA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 2l:12971630-12971688 - | TCATCGTCATCACCTTCTTCGCCGTCTTTGGACTAATTATTCTAACCGAGGTAAGTCCA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_16:1512533-1512591 - | TCATCGTCATCACCTTCTTCGCCGTCTTTGGACTAATCATTCTAACCGAGGTAAGTCCA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2L:9792517-9792577 - | TCATCGTCATCACCTTCTTCGCCGTCTTTGGATTAATCATTCTAACGGAGGTAAGTCCAA----G----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4929:12087025-12087085 + | TCATCGTCATCACCTTCTTCGCCGTCTTTGGACTAATCATTCTAACGGAGGTAAGACCAA----G----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:53 PM