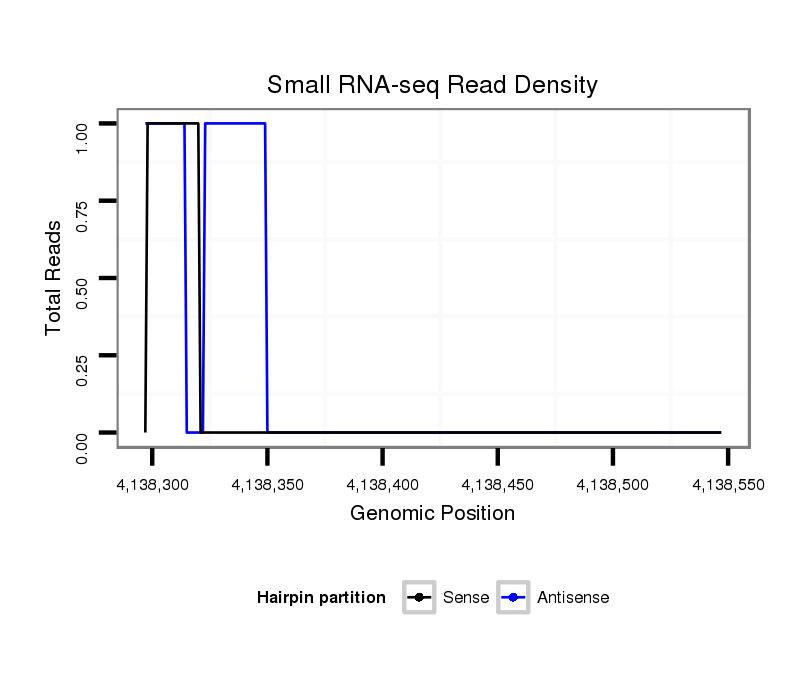
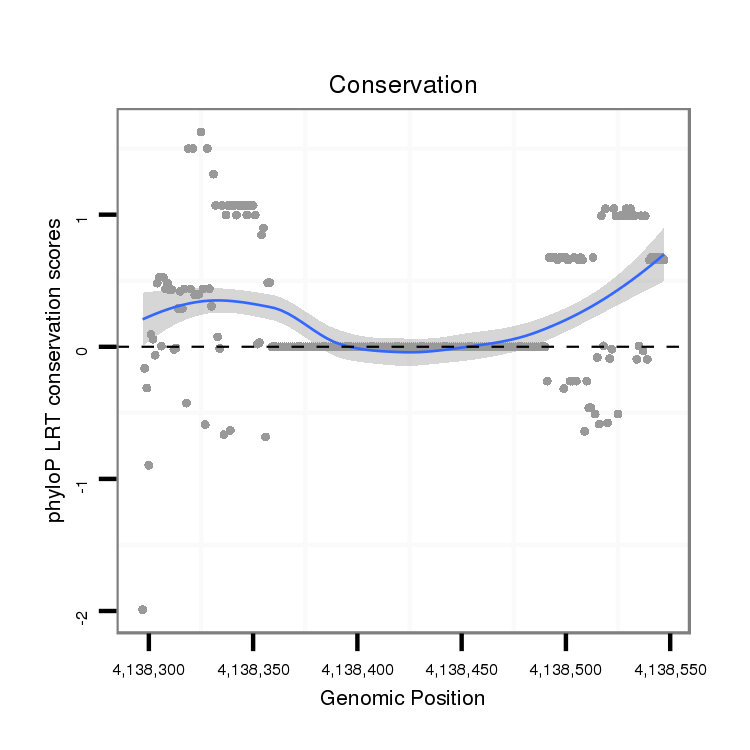
ID:dvi_14685 |
Coordinate:scaffold_13042:4138347-4138497 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ15993-cds]; exon [dvir_GLEANR_16417:2]; intron [Dvir\GJ15993-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GTTTCATCGCCATTTCTCGCAGGTCTCCAAAGATGAGAAGCTTATCAATTGTAAGTACTCGCTAACGTGAAAAGTCATGATTTGACTCAGAAACATTCAATGATAAGTTTAAGCTGAGTAAATCCCAACATAACTGTACTTTTCTCTCCCTTTGTAGACGTTAGGCTGAGCCATTTAAACAGACTTGATCCCAGCATAACATTACTTTTCTCGCAATACACTTAATTCTCCCTCTCCCTCTCCCTTTGTTG **************************************************...........((((((((((((((((.((((((.(((((((((............))))...)))))))))))..........)).))))))).............)))))))((((.......................))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060659 Argentina_testes_total |
M047 female body |
SRR060657 140_testes_total |
GSM1528803 follicle cells |
SRR060664 9_males_carcasses_total |
SRR060665 9_females_carcasses_total |
V047 embryo |
SRR060672 9x160_females_carcasses_total |
SRR060674 9x140_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060666 160_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................CGCAGGTGTCCTAAGATG........................................................................................................................................................................................................................ | 18 | 2 | 2 | 3.00 | 6 | 2 | 3 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................CGCAGGTGTCCTAAGATGAG...................................................................................................................................................................................................................... | 20 | 2 | 1 | 3.00 | 3 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................ACGCAGGTGTCCTAAGATGA....................................................................................................................................................................................................................... | 20 | 3 | 2 | 1.00 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 |
| ............TTTCTCGCAGGTCTCCAAT............................................................................................................................................................................................................................ | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .TTTCATCGCCATTTCTCGCAGGT................................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............GTCGCAGGTCTCCAAAGACG........................................................................................................................................................................................................................ | 20 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................ACGCAGGTGTCCTAAGATGAG...................................................................................................................................................................................................................... | 21 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................CGCAGGTGTCCTAAGATGA....................................................................................................................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................GCAGGTGTCCTAAGATGAG...................................................................................................................................................................................................................... | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................CACCAAAGATGAGAAGACTAT.............................................................................................................................................................................................................. | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................ACGCAGGTGTCCTAAGATG........................................................................................................................................................................................................................ | 19 | 3 | 8 | 0.25 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CAGGTGTCCTAAGATGAG...................................................................................................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................CAGGTGTCCTAAGATGAGC..................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.15 | 3 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................GTGGAAAGTCCTGATTTCA...................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
CAAAGTAGCGGTAAAGAGCGTCCAGAGGTTTCTACTCTTCGAATAGTTAACATTCATGAGCGATTGCACTTTTCAGTACTAAACTGAGTCTTTGTAAGTTACTATTCAAATTCGACTCATTTAGGGTTGTATTGACATGAAAAGAGAGGGAAACATCTGCAATCCGACTCGGTAAATTTGTCTGAACTAGGGTCGTATTGTAATGAAAAGAGCGTTATGTGAATTAAGAGGGAGAGGGAGAGGGAAACAAC
**************************************************...........((((((((((((((((.((((((.(((((((((............))))...)))))))))))..........)).))))))).............)))))))((((.......................))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
V047 embryo |
V053 head |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ..........................GGTTTCTACTCTTCGAATAGTTAACAT...................................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| CAAAGTAGCGGTAAAGAG......................................................................................................................................................................................................................................... | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| ....................................CTTCGAATACATCACATTCAT.................................................................................................................................................................................................. | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...........TTAGGAGCGTTCAGAGGTTT............................................................................................................................................................................................................................ | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................ACTCGGAAAATTGGTATGA.................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:4138297-4138547 + | dvi_14685 | GTTTCATCGCCATTTCTCGCAGGTCTCCAAAGATGAGAAGCTTATCAATTGTAAGTACTCGCTAACGTGAAAAGTCATGATTTGACTCAGAAACATTCAATGATAAGTTTAAGCTGAGTAAATCCCAACATAACTGTACTTTTCTCTCCCTTTGTAGACGTTAGGCTGAGCCATTTAAACAGACTTGATCCCAGCATAACA------TTACTTTTCTCGCAATACACTTAATTCTCCCTCTCCCTCTCCCTTTGTTG |
| droMoj3 | scaffold_6359:2283782-2283838 - | GTTCCATCGCCATTTTTCGCAAGTCTCCAAAGACGAGAAGCTCATCAATTGTAAGTA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_14853:6395825-6395886 - | ATTCCGTCGCCACTTCTCGCAGGTCTCCAACGATGAGCACCTCATCAATTGTAAGCACTAGC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004515:3904100-3904158 - | GTTTCATCGACATTTCACACAGGTCTCCAAGGATGAAAAACTAATCAATTGTAAGTCCT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | 2:11935854-11935855 - | GT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_4:7096620-7096687 + | CCCAG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TATAACAATTGGCTAACCCTCCTCCTCCTCCTCCTCCTTCGCCCTCTCCTCCTCTCTTTGTTG | |
| droAna3 | scaffold_13266:5895854-5895888 - | GCAGCACAGCTGTTTTTCGGGAGCCTCTAGAGTTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000774903:2298-2330 + | CCGCCACCGCCATCCCAGGCATGTCGTCAAGGA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4690:1032476-1032512 - | TTTCTCGCAAC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCCTTCCTCTCCCTCTCCCCCACC-------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:46 PM