
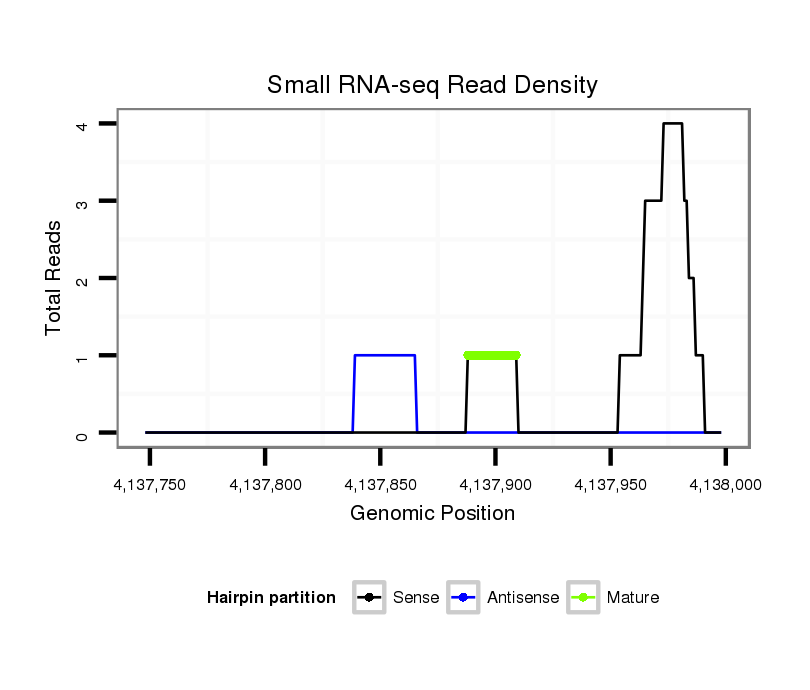
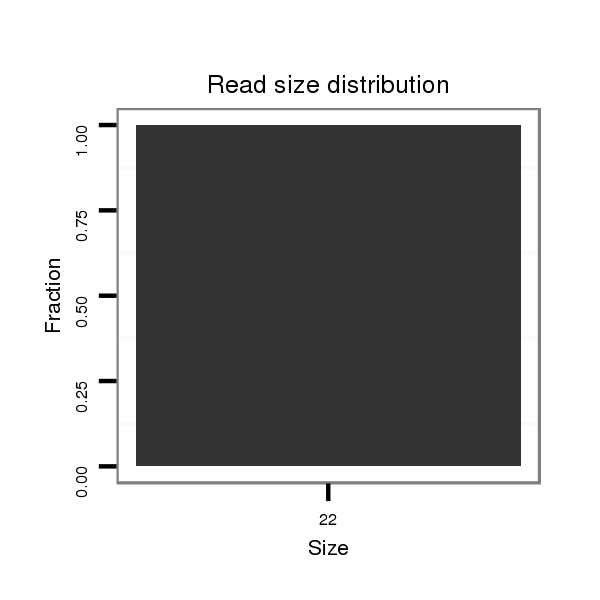
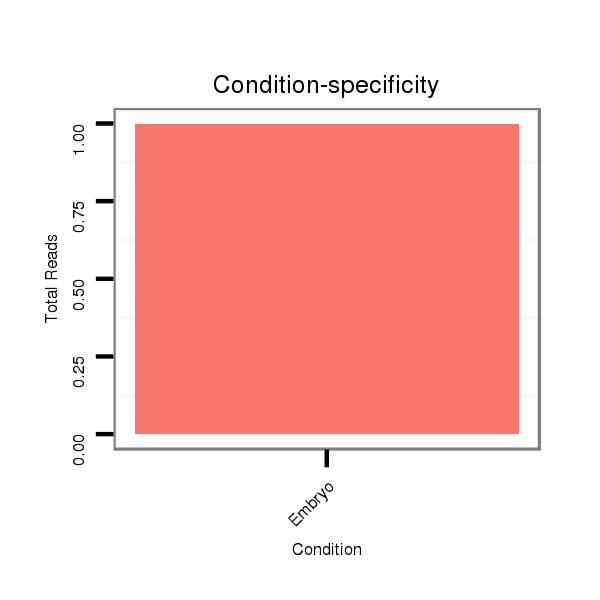
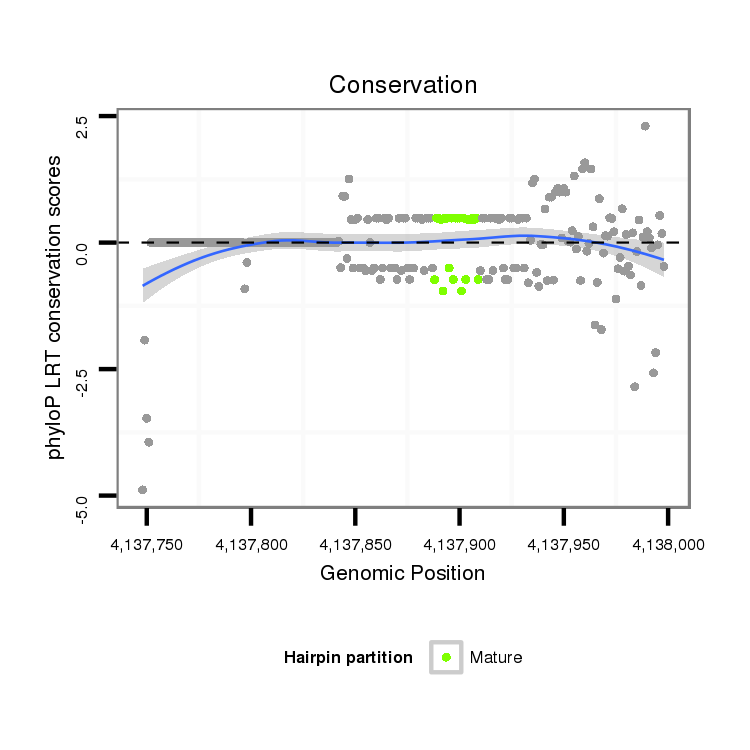
ID:dvi_14684 |
Coordinate:scaffold_13042:4137798-4137948 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.4 | -20.4 | -20.2 | -20.2 |
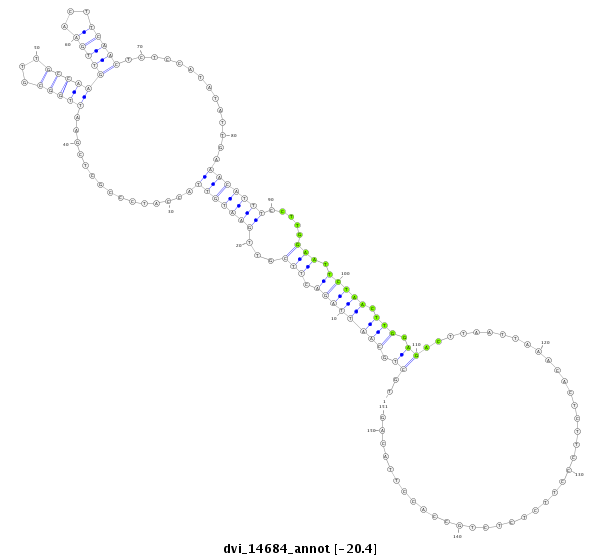 |
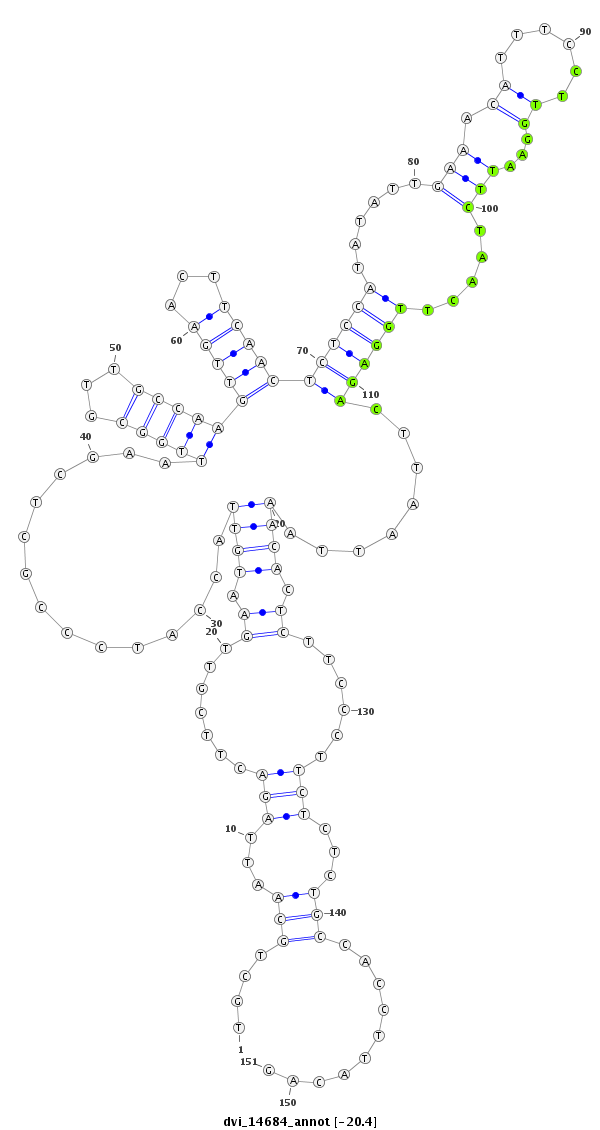 |
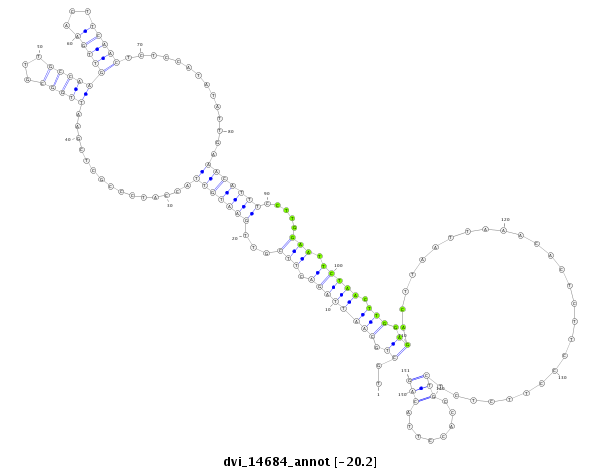 |
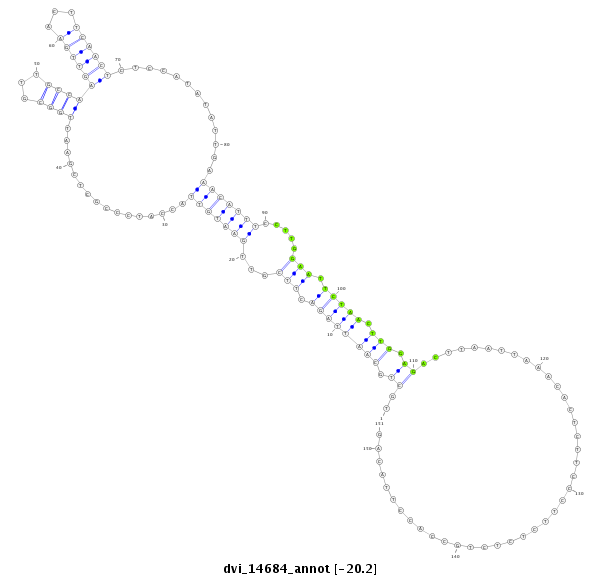 |
exon [dvir_GLEANR_16417:2]; CDS [Dvir\GJ15993-cds]; intron [Dvir\GJ15993-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TTTAAATCAAATGTTGCCATCTGGGGATAAATGCACATATGGCAACGCCATGCTGCAATTAGACTTCGTTGAATGTTACCATCCCGCTCGAATTGGCGTTGCCAAGTTGAACTTCAACTCTCCATATATTGAAACATTTCCTTGGAATTCTAACTTGGAGACTTAATTAAACACTCTTCCCTTCTCTCTGCCACCTTACAGTTCCCTGCCCCAGAGCTACAGCAGCTCCGTCGGACCGCTGCAGTCGAGCG **************************************************..((((....(((.......((.((((.........(((.((((((.(((.(((((......(((((...........))))).......)))))))).)))).)).))).........)))).))......)))....))........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060663 160_0-2h_embryos_total |
V116 male body |
M061 embryo |
SRR060683 160_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
M028 head |
SRR060671 9x160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...AAATCCAACGTTGCCATC...................................................................................................................................................................................................................................... | 18 | 2 | 10 | 6.80 | 68 | 28 | 1 | 3 | 4 | 0 | 0 | 5 | 3 | 5 | 3 | 4 | 3 | 2 | 2 | 0 | 2 | 2 | 0 | 1 | 0 |
| .........................................................................................................................................................................................................................TACAGCAGCTCCGTCGGACCGC............ | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................TGCCCCAGAGCTACAGCAGCTCCGTCGG................. | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................CTTGGAATTCTAACTTGGAGAC......................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................CTACAGCAGCTCCGTCGGAC............... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................CACAGTTCCCTGCCCCAGAGC.................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................CTCCGTCGGACCGCTGCA........ | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AAATCTAACGTTGCCATC...................................................................................................................................................................................................................................... | 18 | 2 | 5 | 0.60 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................GCTTCAGTAGCTCCGTAGGA................ | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................GCTAGAATTGTTGTTGCCAAGT................................................................................................................................................ | 22 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .TAAAATCTAACGTTGCCATC...................................................................................................................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...AAATCCAACGTTGCCATCGG.................................................................................................................................................................................................................................... | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..TAAATCCAACGTTGCCATCG..................................................................................................................................................................................................................................... | 20 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............TGTCAGGTGGGGATAAAT........................................................................................................................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................ACAATTAGACTTCTTTGA................................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AAATTTAGTTTACAACGGTAGACCCCTATTTACGTGTATACCGTTGCGGTACGACGTTAATCTGAAGCAACTTACAATGGTAGGGCGAGCTTAACCGCAACGGTTCAACTTGAAGTTGAGAGGTATATAACTTTGTAAAGGAACCTTAAGATTGAACCTCTGAATTAATTTGTGAGAAGGGAAGAGAGACGGTGGAATGTCAAGGGACGGGGTCTCGATGTCGTCGAGGCAGCCTGGCGACGTCAGCTCGC
**************************************************..((((....(((.......((.((((.........(((.((((((.(((.(((((......(((((...........))))).......)))))))).)))).)).))).........)))).))......)))....))........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
GSM1528803 follicle cells |
M027 male body |
V047 embryo |
M028 head |
M047 female body |
SRR060689 160x9_testes_total |
SRR060655 9x160_testes_total |
SRR060668 160x9_males_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060673 9_ovaries_total |
SRR060674 9x140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................................................................................TGGGATGGTGTCTCGATGTCGT........................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................CTGGGACGGTGTCTCGATGTCGTCG......................... | 25 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................GACGGTGACTCGATGTCGTCG......................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................GTGGAAAGTCAAGGGGCAGGG....................................... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................TAACCGCAACGGTTCAACTTGAAGTTG..................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................ATGGTGACTCGATGTCGTCG......................... | 20 | 3 | 20 | 0.95 | 19 | 3 | 0 | 2 | 0 | 2 | 3 | 3 | 4 | 0 | 2 | 0 | 0 |
| ................................................................................................................................................................................................................GGTGACTCGATGTCGTCG......................... | 18 | 2 | 14 | 0.64 | 9 | 0 | 0 | 1 | 0 | 5 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................GGGGAATGTCGAGGGACGGAG....................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................GATGGTGACTCGATGTCGTCG......................... | 21 | 3 | 11 | 0.27 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 1 |
| ............................................................................................................................................................................................................................TCGTCGAGTCAGCCTGGA............. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............CAGGAGACCACTATTTACGT........................................................................................................................................................................................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................GCAGCCTAGGGATGTCAGC.... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............AACAGGAGACCACTATTTAC.......................................................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................TTTGTGAAGGACCCTTAA...................................................................................................... | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................................................................................GAGACGGGGACTCGATG............................... | 17 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:4137748-4137998 + | dvi_14684 | TTTAAATCAAATGTTGCCATCTGGGGATAAATGCACATATGGCAACGCCATGCTGCAATTAGACTTCGTTGAATGTTACCATCCCGCTCGAATTGGCGTTGCCAAGTTGAACTTCAACTCTCCATATATTGAAACATTTCCTTGG-----AATTCTAACTTGGAGACTTAATTAAACACTCTT------------CCCTTCTCTCTGC--CACCTTACAGTTCCCTGCCCCAGAGCTACAGCAG------------------CTC--CGTCGGA-C---------CGCTGCAGT-CGAGCG |
| droMoj3 | scaffold_6359:2284152-2284330 - | AACT------------------------------------------------------------------------------------------GGCGCTGTCAGACAG-TTTTAGACTTCTAAAATGGCAAAACTTTTTATTGCTGATTAACTATAAGTGGGAGAAATACGTTAACATGATTTGTTTATATATATTCTTTTTTCTTT--CA-TTTTCAGTTCATTGCCTCAAAGCAGCTTCAG---------------CAGCTA--TATCGGT-C---------CGCTGCAGT-CGAGCG | |
| droGri2 | scaffold_14853:6396161-6396217 - | CTTG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTTCCTTGCCGCAGAGCTACATCAG------------------CTA--CATTGGT-C---------CGCTGCAGT-CGAGCG | |
| droWil2 | scf2_1100000004515:3904511-3904596 - | TTTC---------------------------------------------CC-------------------------------------------G---TT------------------------------------------------------------------------------------------------------TCCTGGCATTCATTGCAGATCCTTGAGGCACAGCTATTCCAG------------------CTC--ACTTAGT-CCCCTGAACCAATTGCAGA-CGAGTG | |
| dp5 | 2:11339737-11339769 - | CACA------------------------------------------------------------------------------------------GT--------------------------------------------------------------------------------------------------------------------------------------------TGCTGTGG------------------C----------T-C---------CGCCGCATCCCCAGCA | |
| droPer2 | scaffold_0:9176259-9176297 + | CACA------------------------------------------------------------------------------------------GT--------------------------------------------------------------------------------------------------------------------------------------------TGCTTTGG------------------CTC--CGTCGC------------ATCCCCAGCAGCAGCA | |
| droBip1 | scf7180000396572:151682-151730 + | AGCA------------------------------------------------------------------------------------------GC-----------------------------------------------------------------------------------------------------------------------------------------------AGCAACACCAACAGATCCCGCAGC-------------------AGCACCAACAGCTCCAGCA | |
| droKik1 | scf7180000302685:696993-697026 - | CGGG------------------------------------------------------------------------------------------GC--------------------------------------------------------------------------------------------------------------------------------------------CACCGCAG------------------C-------------------AGCCGCCGCAGCAGCAGCA | |
| droFic1 | scf7180000454072:3187346-3187382 + | CCGG----------------------------------------------T-------------------------------------------GC--------------------------------------------------------------------------------------------------------------------------------------------CGCCGCAG------------------TCC------ACC-T---------CGGTGCTCT-TGGACC | |
| droEle1 | scf7180000491272:509493-509531 - | TGCC---------------------------------------------CA-------------------------------------------GC--------------------------------------------------------------------------------------------------------------------------------------------AGCAGCAG---------------CAGC-------------------AACATCACCAGCAGCAGCA | |
| droRho1 | scf7180000776532:36102-36105 - | A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCA | |
| droBia1 | scf7180000302136:645367-645418 + | GTTG------------------------------------------------------------------------------------------CT---------------------------------------------------------------------------------------------------------------------------------GGTCGCAGTGTTGCTGCTG------------------CGT--TGTTGCTGC---------TGCTGCTGC-TGGGCC | |
| droTak1 | scf7180000415352:1047926-1047984 - | CAAC---------------------------------------------AT-------------------------------------------GCCGCT------------------------------------------------------------------------------------------------------GCCTACC---------------------------AGCAGCAGCACAA---------T---ATGTATGC---C-C---------AGCAGCAGCAGCAGCA | |
| dm3 | chr2R:7415146-7415177 - | CAAT------------------------------------------------------------------------------------------GG--------------------------------------------------------------------------------------------------------------------------------------------CCCTGG----------------------------AGGA-T---------CGCTGCAGT-CCACAG | |
| droSim2 | 3r:7752251-7752291 + | GAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACAGCAGCACCA---------ACAGC-------------------AACAGCTGCGGTACGGCCA | |
| droSec2 | scaffold_12:646672-646712 + | GAGC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACAGTAGCACCA---------ACAGC-------------------AACAGCTGCGGTACGGCCA | |
| droYak3 | 3R:8952837-8952877 + | TGCC------------------------------------------------------------------------------------------C---------------------------------------------------------------------------------------------------------------------------------------CAGGGC----GCTA------------------CTC--CGTAGTA-C---------TGCGGCATC-TGGGCG | |
| droEre2 | scaffold_4929:9006076-9006112 - | AGCA------------------------------------------------------------------------------------------AC-----------------------------------------------------------------------------------------------------------------------------------------------AGCTGC---C---------GCGGC-------------------GGCAGCTGCAGCAGTGGTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:45 PM