
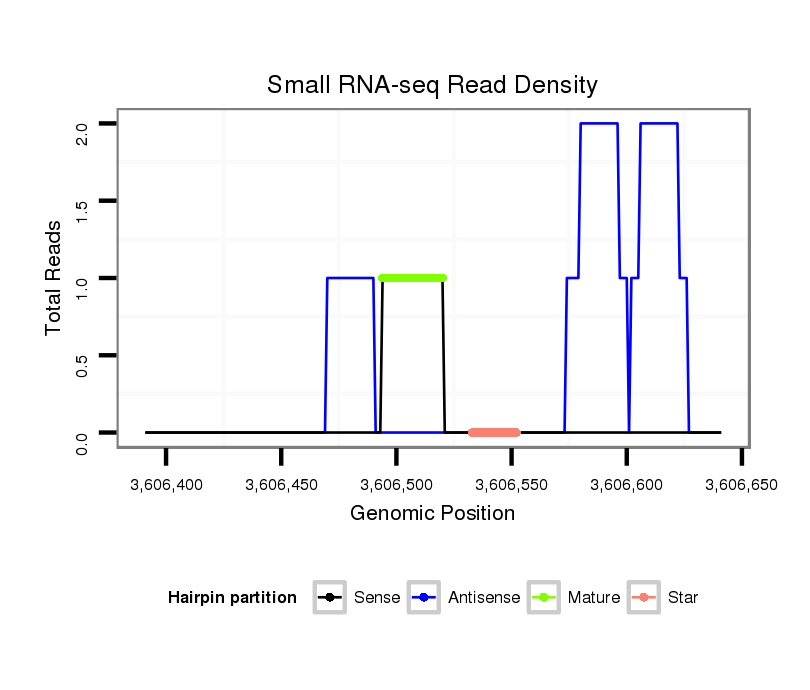
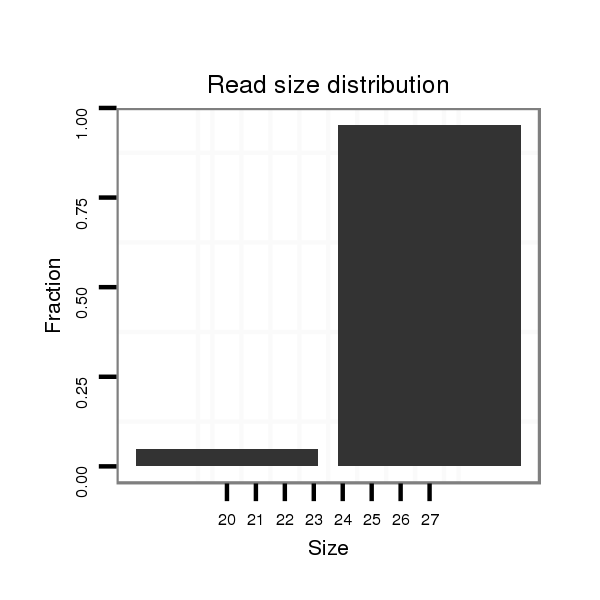
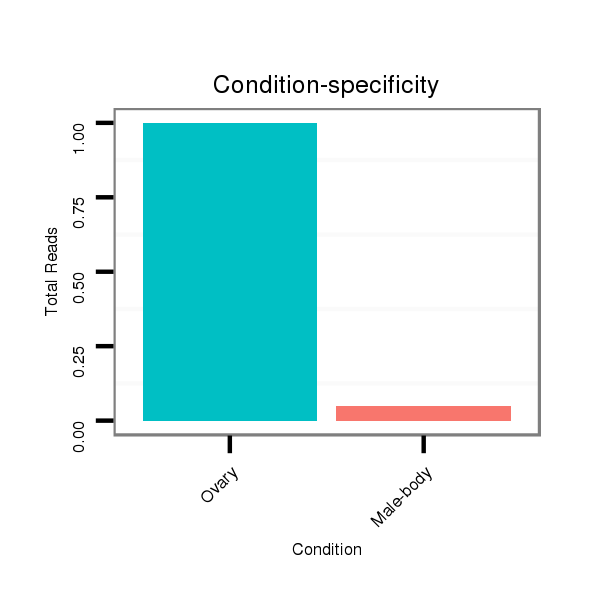
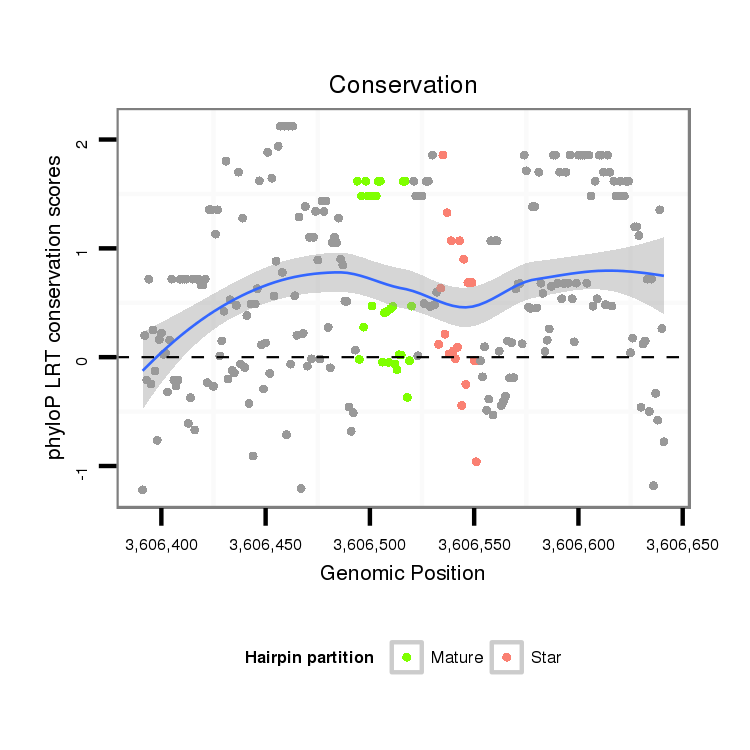
ID:dvi_14641 |
Coordinate:scaffold_13042:3606441-3606591 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -1.0 | -0.6 | -0.6 |
 |
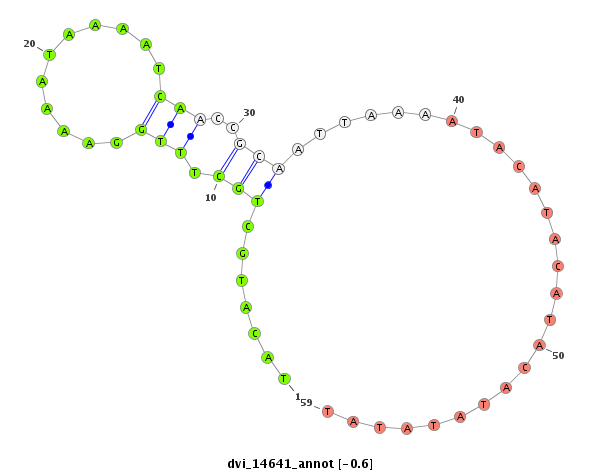 |
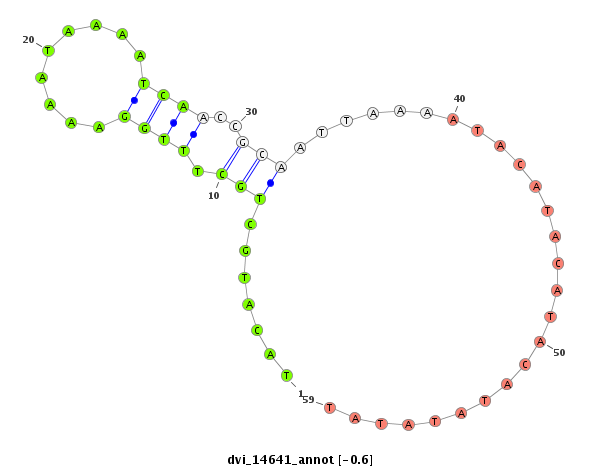 |
intron [Dvir\GJ15970-in]; CDS [Dvir\GJ15970-cds]; exon [dvir_GLEANR_16396:2]; intron [Dvir\GJ15970-in]
| Name | Class | Family | Strand |
| (TC)n | Simple_repeat | Simple_repeat | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------##############################-------------------- AAATGTATAGTTCAATTATTAACGACAAGCTCTTATATTATATACATATATCTCTATCTCTTTCTCTCTCTCTCTCTCTATTTCTTTCTTCCTGTACAACCTTTACATGCTGCTTTGGAAAATAAAATCAACCGCAATTAAAATACATACATACATATATATATATCTCTTTATTACAATAACAACTTTCCTCTTGAAAAGCTCTACTAAAAAAACAACAACTACAACCAGGTATTATGCTCTATGAAAAT *******************************************************************************************************.......(((....((........))....)))..........................***************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060677 Argx9_ovaries_total |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
V047 embryo |
V116 male body |
SRR060665 9_females_carcasses_total |
M047 female body |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................TACATGCTGCTTTGGAAAATAAAATCA......................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................CATGCTGCTCTGGGAAATAAA............................................................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................CATGCTGCTCTGGGAAAT................................................................................................................................ | 18 | 2 | 7 | 0.29 | 2 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................CTTTACAAGCTGCCATGGAA................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...........................................ACACATATCGATATCTCTTTCT.......................................................................................................................................................................................... | 22 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................CATGCTGCTCTGGGAAATGA.............................................................................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................................................................................................AACCGGGTATTTTGTTCTAT...... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................ATGCTGCTCTGGGAAATGAA............................................................................................................................. | 20 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................ATACATACATACATATATAT......................................................................................... | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................TGAACACGCTGCTTTGGAA................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
|
TTTACATATCAAGTTAATAATTGCTGTTCGAGAATATAATATATGTATATAGAGATAGAGAAAGAGAGAGAGAGAGAGATAAAGAAAGAAGGACATGTTGGAAATGTACGACGAAACCTTTTATTTTAGTTGGCGTTAATTTTATGTATGTATGTATATATATATAGAGAAATAATGTTATTGTTGAAAGGAGAACTTTTCGAGATGATTTTTTTGTTGTTGATGTTGGTCCATAATACGAGATACTTTTA
*****************************************************************************************.......(((....((........))....)))..........................******************************************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060679 140x9_testes_total |
SRR060670 9_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060678 9x140_testes_total |
SRR060681 Argx9_testes_total |
SRR060667 160_females_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .............................................................................................................................................................................................GGAGAACTTTTCGAGATGATT......................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................TTTTGTTGTTGATGTTGGTCC................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................GTTGTTGATGTTGGTCCATAA............... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................TTGAAAGGAGAACTTTTCGAGAT............................................. | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................TAAAGAAAGAAGGACATGTTG....................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................CTGTTGTTGATGTTGGTCCATA................ | 22 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................TAACGAACGAAGGACATGTTG....................................................................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................TCGAGAAGAGAAAATATGTAT........................................................................................................................................................................................................... | 21 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................TATGTATGTATGTATATATAT........................................................................................ | 21 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........................................................................................................................................TTTTATGTTTGTATGTATA............................................................................................. | 19 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:3606391-3606641 + | dvi_14641 | AAA--TGTATAGTTCAATTATTAACGA----CAAGCTCT----TAT-ATT-------------------AT----------AT------------ACATATATCTCT----------AT-----CTCTT--TCTCTCTCTCTCTCTCTATTTCTTTCTTCCTG----------T--------------------------------------ACAACCTTTACATGCTGCTTTGGAAAATAAAATCAACCGCAATTAAAATACATACATACATATATATATATCTCTTTA-T--TACAATAACAACT---TTCCTCTT---G---------------------A---AAAGCTCTACTAAAAAAACAACAACTACAACCAGGTATT--------------------------------------------ATGCTCTATGAAAAT |
| droMoj3 | scaffold_6308:831029-831300 + | AAA--TGTACAGTGTAATAATTAAGAAAATAAAAGCTCTAACTTAT-ATTAATATATATAATCAAAATTATGAATATATA----------------------TTAAAACATATTTAAAT------CTTT----TCTCTATTT----------CTTTCTT----CCTATTACCT----------------------------------------------ATACATGCTGCTTGGGAAA-TAAAAAAAACTGCTATTAAAATACATATATA------TCTATAATTATTAT-CTATACTATAATAACT---TTCCTCAC---G---------------------AAAAAAAGCTCTACTAAAAAAACAACAACTACAACCAGGTATT------------------------------------------CAATGCTCTCTA-CAAA | |
| droGri2 | scaffold_14853:10133963-10134211 + | AATAATGCACAGATCAACTGTTAACAA----AAAGCTTTATCTTCTAAAT-ATGTG------------TAT----------AT------------ACATATATTTATATAT------ATACAATATCTT----TCTTTATTT----------CTTTCTT----------CCCTT--------------------------------------ACAATAATTACATGCTGCTTTGGAAA-TAAAAACAACTGCAATTAAAATACATACCTAAA--------CATCTCTTTAACTGATTTATAATA-TT---TTCCTCTT---G---------------------A---AAAGCTCTACTAAAAAAACAACAACTACAACCAGGTACT-------------------------------------------------CTATCAAATA | |
| droWil2 | scf2_1100000004921:2233959-2234151 - | TATT--T---------------------------------------------------------------T----------TT------------CTATCTCTCTCT----------GT-----CTCTC--TCTCTCTCTCTCTCTCTATCTCTCTCTATCTG----------TAGTGGATATTTTTGATTTTCAGTCTTAGAGTACGCCTCAC------------------------------------------AATATACACACAAACACATACGCGCGCTTTTTTT-TTGTTTTTGATCAATT---TTCCCCCA----AAAATG---------------AAAAAATGAACCAACGAAAAGA---AAACTAC---------------------------------------------------------------------- | |
| droPer2 | scaffold_63:298095-298325 - | TATG-------------------------------------------AAG-ATGTG------------TAT----------TTATCTCG--ATTGCTATA-----------------A--------------TTCTCTATTTCCCGTTC----TT--------TTTATCGCCT-----------------------------------------------TACATGCTGCTTGAATTGAATTAATTTACCGCAATCATTTTATAT-----------------------------AACTATGATAATCTCG--TCTTCTATCG---------------------AACAAAAGCTCTACCAAAAAAACTACAACAACAACCAGGTACAAGAAATATCTAACTAAAAAAGATTATTTTTTGTTTCTGTTTTTAATGGTT----TAAAT | |
| droAna3 | scaffold_13417:1139917-1140073 + | T-------------------------------------------------------------------------------------------------------------------------------T--TCCCTCTATTTC----------TC--------TCTATTCTCT-----------------------------------------------TCCCTGCTGCTTGGATTACGATAATCAACCGCAATTATTTCA--A-----------------------------AACTTTAATAATCTTC--TCTCCTATCTAAAAAAAAAAAATAAATCAAAAATAAAAGCTCTACTAAAAAAACAACAACAACAACCAGGTA------------------------------------------------------------- | |
| droBip1 | scf7180000395529:233929-234148 - | GCT----------------------------------CT----TGT-AAG-A------------------------TACCTTTATTACCAT-TTTTAATA------------AATAA--------CTTT--TCCCTCTATTTC----------TC--------TCTATTCTCT-----------------------------------------------TCCCTGCTGCTTGGAATACGATAATCAACCGCAATTATTTCA--A-----------------------------TACTATAATAATCTTC--TCTCCTATTTAAAAAAAA----ATAAATA--T---AAAGCTCTACTAAAAAAACAACAACAACAACCAGGTACAAAAAAAA-ATAACTAAA---------------------------ATGTAT---ACCAAC | |
| droKik1 | scf7180000302696:443203-443405 + | TATA--TTTGTA-------------------------TT----TG-----------------------TATGTATATACC----------------------CTTTTACCTAACTTAAT------ACTCTCTCTCTCTTTTTT----------CC--------TCTATTCTCT----------------------------------------------TTTCCTGCCGCTTGGAATACTTAAAACAACCGCAATTATTATA--A-----------------------------TATCTTAATAATCTCCTTTCTCCTATCTAAAAACAA----TGCAATATAAAAAAAAGCTCTACTAAAAAAACAACAACAACGACCAGGTA------------------------------------------------------------- | |
| droSec2 | scaffold_21:504843-504923 + | CAAA--ATGCAATTTG----------------------------------------------------TTT----------GCAGCTCT--CTCTCTCTCTCTCTCT----------AT-----CTCTC--TCTCTCTCTCTCTCTCTCTCTCTTTCTTTCTG----------T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4784:3523976-3524011 + | ACC--------------------------------------------------------------------------------------------------------------------------TCTT--TCTCTCTCTCTCTCGCTC----TT--------TCTCCCATC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:24 PM