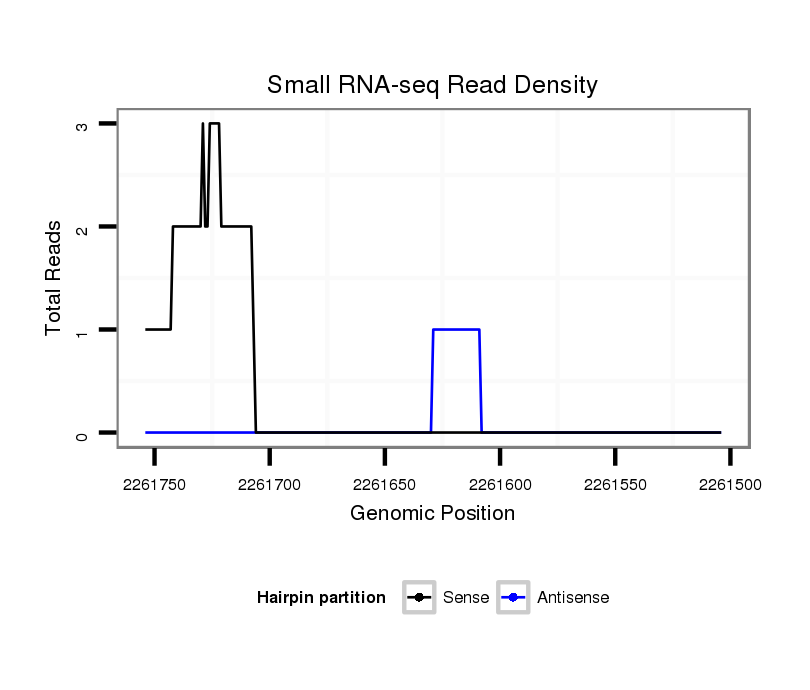
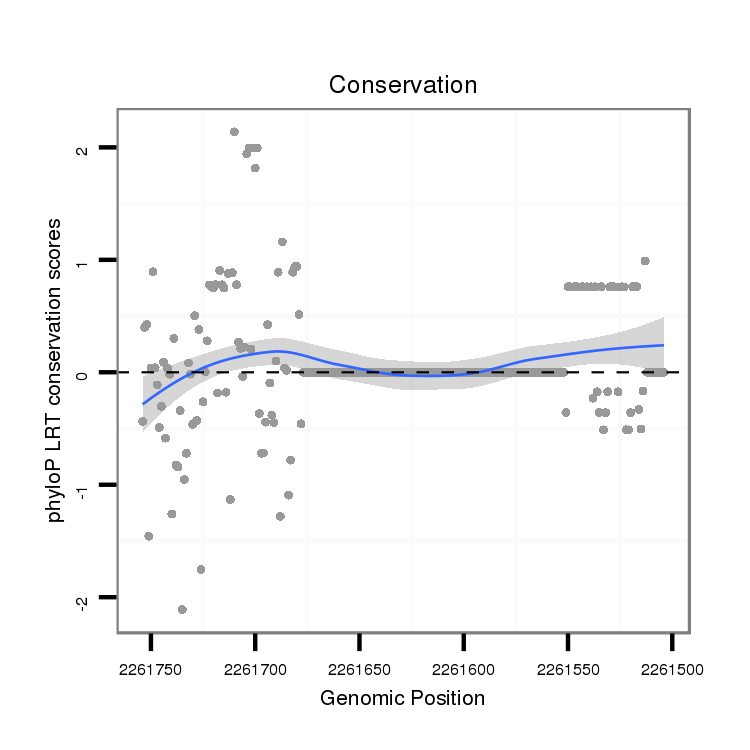
ID:dvi_14395 |
Coordinate:scaffold_13042:2261554-2261704 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_16119:4]; CDS [Dvir\GJ15679-cds]; intron [Dvir\GJ15679-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CAGGCGCAGCGACAGCTCCCTCAACAGCGACATCCGTTGGATTAAGTCATGTGAGTACAAAAATCGGATGCAGAATCTATTGGCACCTTTAATCTAGGAAAGATCCATTGAATCATTAAAAAAGGGGATCAAAGGGTTATTGATTAGGGCGATAGGCCTGGTCTGTTTGTCTGCAACATAGCTCCAAAATTATTGCCCACTTTAATGTTCGACACATACAAGTTAATGGCACCGCAATCCCTCCTCTGCAA **************************************************.......................((((...((((.(((((...........(((((...................))))))))))))))..)))).(((((((((...(((.(((((((....))).))))..)))...)))))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060667 160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
V047 embryo |
SRR060679 140x9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CAGGCGCAGCGACAGCTCCCTCAACA................................................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................GACATCCGTTGGATTAAGTC........................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............CAGCTCCCTCAACAGCGACAT.......................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................AGCGACATCCGTTGGATTAAGT............................................................................................................................................................................................................ | 22 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................CAGAATCCATCGGCACCTTT................................................................................................................................................................. | 20 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................TACAGGATAATGGCACCGC................ | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................ATTCGGGTGATAGGCGTGG.......................................................................................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................CATCCGCTGGATTAAGGCT.......................................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GTCCGCGTCGCTGTCGAGGGAGTTGTCGCTGTAGGCAACCTAATTCAGTACACTCATGTTTTTAGCCTACGTCTTAGATAACCGTGGAAATTAGATCCTTTCTAGGTAACTTAGTAATTTTTTCCCCTAGTTTCCCAATAACTAATCCCGCTATCCGGACCAGACAAACAGACGTTGTATCGAGGTTTTAATAACGGGTGAAATTACAAGCTGTGTATGTTCAATTACCGTGGCGTTAGGGAGGAGACGTT
**************************************************.......................((((...((((.(((((...........(((((...................))))))))))))))..)))).(((((((((...(((.(((((((....))).))))..)))...)))))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|
| ..........................................................................................................................................................TCGGACAAGACATACAGACGTT........................................................................... | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 |
| .............................................................................................................................CCTAGTTTCCCAATAACTAAT......................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 |
| ......GTCGCTGTCGAGGTGGTTGTCG............................................................................................................................................................................................................................... | 22 | 2 | 2 | 0.50 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:2261504-2261754 - | dvi_14395 | CAGGCGCAGCGACAGC----TCCCTCAACAGCGACATCCGTTGGATTAAGTCATGTGAGTACAAAAA-------TCGGATGCAGAATCTATTGGCACCTTTAATCTAGGAAAGATCCATTGAATCATTAAAAAAGGGGATCAAAGGGTTATTGATTAGGGCGATAGGCCTGGTCTGTTTGTCTGCAACATAGCTCCAAAATTATTGCCCACTTTAATGTTCGACACATACAAGTTAATGGCACCGCAATCCCTCCTCTGCAA |
| droMoj3 | scaffold_6328:1298824-1298888 + | CAAGCGCAGCGGCAAC----TCCGACCCCGACGAGATCCGCTGGCTGCAGTCGAGTAAGTATGGAAG-------CC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droGri2 | scaffold_14853:1436165-1436240 - | CAGGAGCAGCAGCAGC----TGCAACAACAGCAACATCAACAATGGAAAATAACG-----CCGGAAA-------TCGCAATTCGAATT-----------------------------------------------------------------------------------------------------------------------------------------------------------------TATT--------- | |
| droWil2 | scf2_1100000004521:5542269-5542300 + | CAGCAGCAGCGGCAGC----AACAGCAGCAGCGGCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XL_group1a:6481329-6481389 + | CAGGCAGGAGGCG-AATGCACGGCTAAAGAGCACAGCCCGATGGATTAAGTTGCGTGAGTAC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302696:308272-308353 - | TCGCCACAGCGACACG----CATCTGAATACCAACGCCCGGTGGCTGAAGACGCGTAAGTAATGATGATAAAAAATGAATGGTGAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454072:749897-749952 - | TCGCCATGATGACACC----GATGGGTCAACAGCCGCCCGCTGGCTGAAGACGCGTAAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491044:568541-568593 + | CCGGCACAATGATGAC----GGCA---CAATATCCGCCCGCTGGCTGAAGACACGTAAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000776942:251223-251286 + | CCGTCACGGTGACGAC-------GGGACGATCTCCGCGCGCTGGCTAAAGACACGTAAGTGCATCG-----------CATGC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droBia1 | scf7180000301760:2734207-2734266 - | CCGCCACATGGACTAC----GGGAAAACGATCACCTCCCGCTGGCTAAAGACGCGTAAGTGAAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droTak1 | scf7180000413955:120913-120968 + | TCGGCACAGTAGCTAC----GACAATTCGGTCTCCGCCCGCTGGCTAAAGACGCGTAAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chrX:7553316-7553371 + | AAATTATTGTTTATGC----C-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATGTTCGACACAAATCACGCAATGACACGCAAATTCCT--------- | |
| droSim2 | x:6265800-6265863 + | CCGCCACAAGGACAGC----AACAAGACGACCGAAGCCCGCTGGCTGAAGGCGCGTAAGTTTTGAAA-------T------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_4:132739-132794 - | CCGCCACAATGACAGC----AACAAGACGACCCAAGCCCGCTGGCTGAAGGCGCGTAAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | X:2625020-2625081 + | TCGCCACAGTGACACC----AACGAAACGACCGACGCCCGCTGGCTGAAGGCGCGTAAGTGTAGAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4690:15588903-15588964 + | CCGCCACAGTGACACC----AACAAGACGACCGACGCCCGCTGGCTGAAGGCGCGTAAGTGTAGAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:17 AM