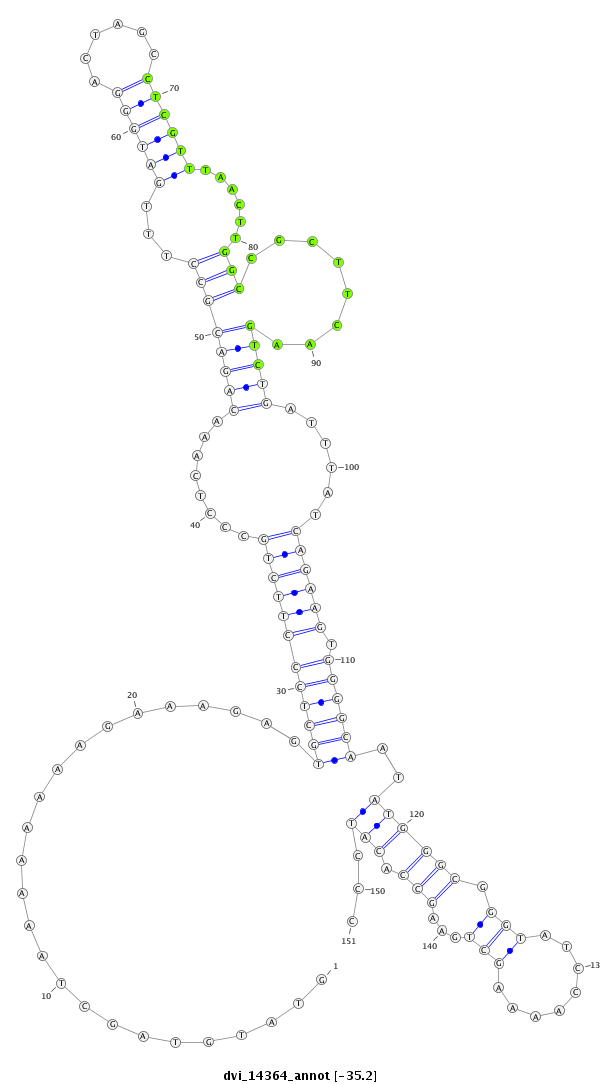
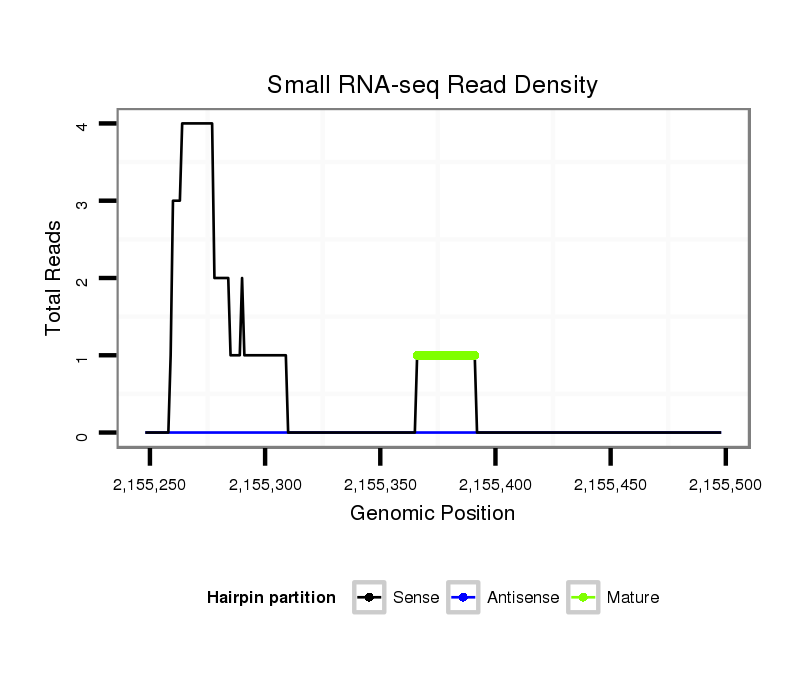

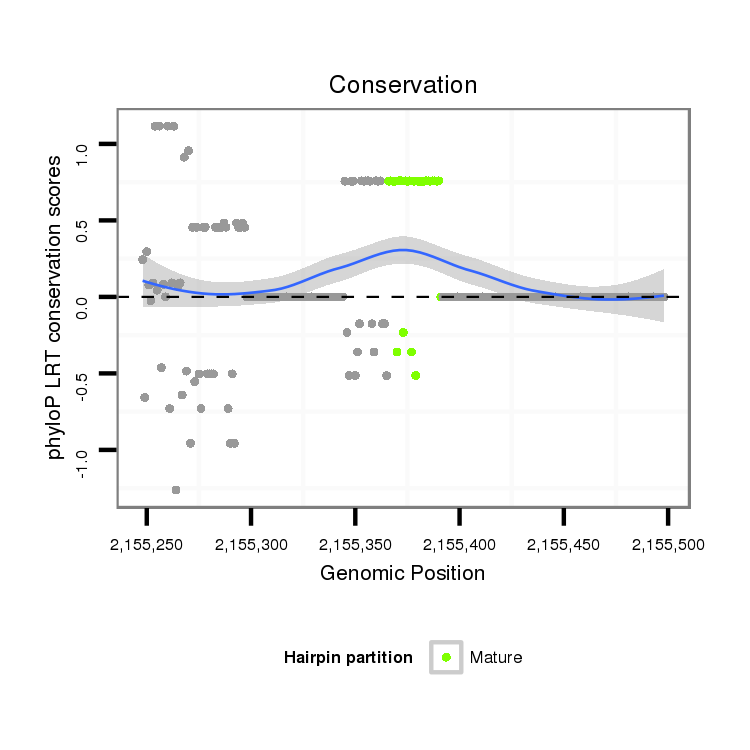
ID:dvi_14364 |
Coordinate:scaffold_13042:2155298-2155448 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -34.9 | -34.9 | -34.8 |
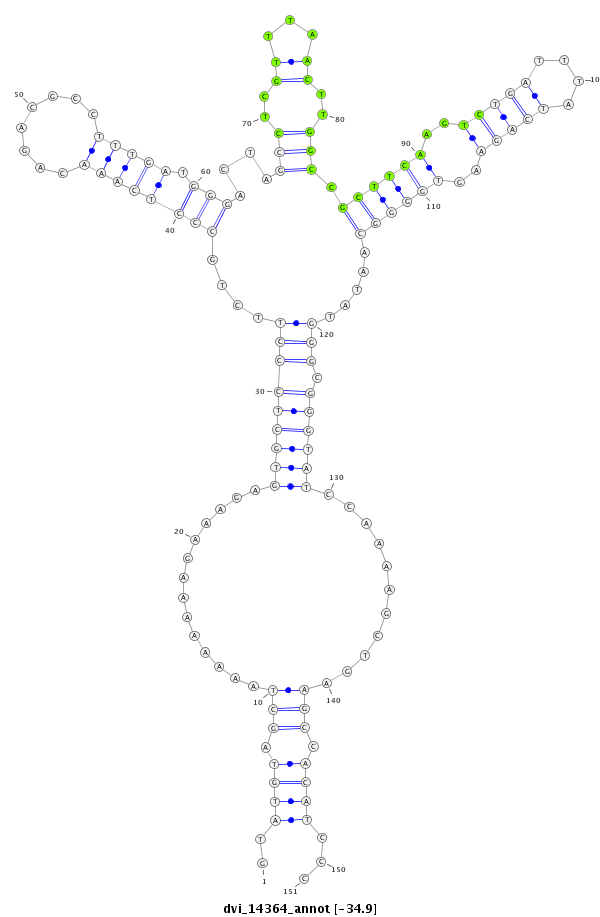 |
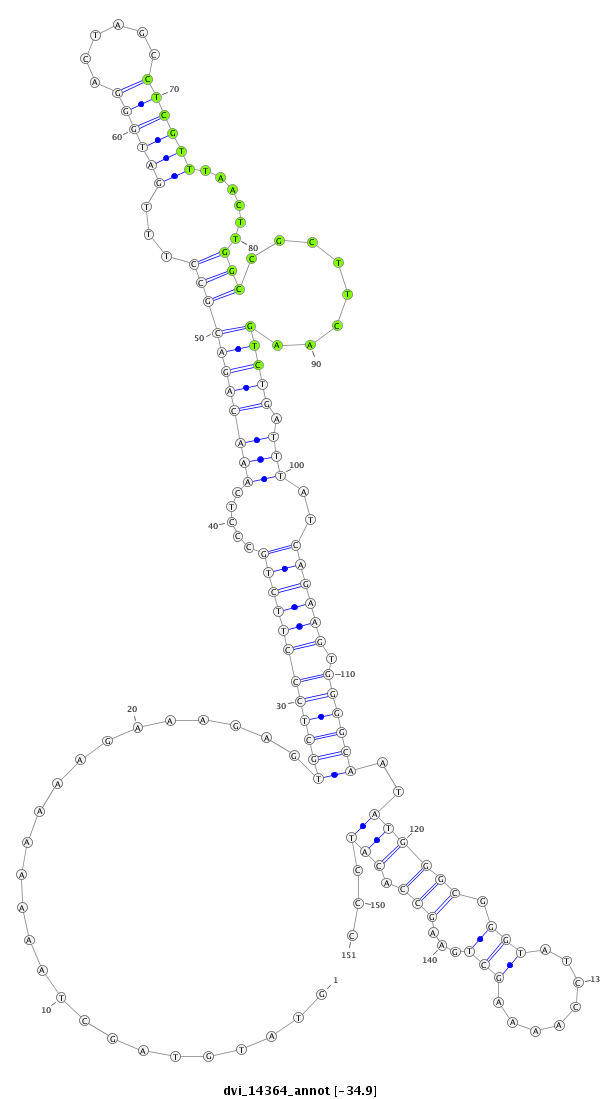 |
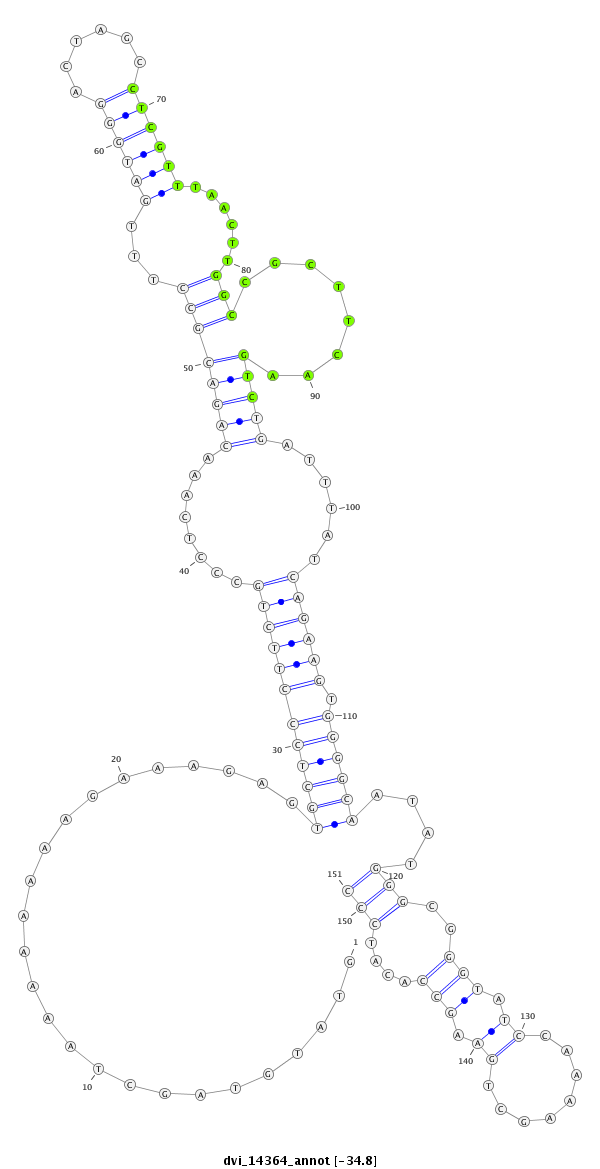 |
CDS [Dvir\GJ15883-cds]; exon [dvir_GLEANR_16316:2]; intron [Dvir\GJ15883-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CTGTTCCAGTTCCTGCTCCTGGACCTGGCCCAATGGCGCACACGGAGCAGGTATGTAGCTAAAAAAAAGAAAGAGTGCTCCCTTCTGCCCTCAAACAGACGCCTTTGATGGGACTAGCCTCGTTTAACTTGGCCGCTTCAAGTCTGATTTATCAGAAGTGGGGCAATATGGGCGGGTATCCAAAAGCTGAAGCCACATCCCAGATTTCGTGCAACTAGCTCGTATAGTTCTCGAGTTATGCTTGAAGAAAA **************************************************.........................((((((((((((........((((((((...((((((......))))))......)))........)))))......)))))).))))))..((((((.(((........)))...))).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060686 Argx9_0-2h_embryos_total |
M028 head |
M061 embryo |
SRR060681 Argx9_testes_total |
M047 female body |
SRR060687 9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR060678 9x140_testes_total |
SRR060682 9x140_0-2h_embryos_total |
V047 embryo |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................ACGGAGCAGCTAGGTAGCT............................................................................................................................................................................................... | 19 | 2 | 1 | 3.00 | 3 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............CTGCTCCTGGACCTGGCC............................................................................................................................................................................................................................. | 18 | 0 | 1 | 2.00 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........CCTGCTCCTGGACCTGGCCCAATGGC...................................................................................................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................GGGGATGTAGCTCAAAAAAAGAA.................................................................................................................................................................................... | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TCCTGGACCTGGCCCAATGGCGCACAC................................................................................................................................................................................................................ | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................CGGAGCAGGTATGTAGCTAA............................................................................................................................................................................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................CTCGTTTAACTTGGCCGCTTCAAGTC........................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................CAATGGCGCACACGGAGCAGAAT...................................................................................................................................................................................................... | 23 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................GGGGATGTAGCTCAAAAAAA....................................................................................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................GGGCACTATGGGTGGGTA......................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................CGGAGCAGTTAGGTAGCT............................................................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................GGGGATGTAGCTAAAAAAA........................................................................................................................................................................................ | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................TATGAAGGTAAAAAAAAGAAA................................................................................................................................................................................... | 21 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................TAGGGCTCTATGGGCGGGT.......................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........GTTAATGCTCCGGGACCTG................................................................................................................................................................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........TCCTGCTCCTGGACCTGC............................................................................................................................................................................................................................... | 18 | 1 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................TTGATGGCACTAGTCTAG................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GACAAGGTCAAGGACGAGGACCTGGACCGGGTTACCGCGTGTGCCTCGTCCATACATCGATTTTTTTTCTTTCTCACGAGGGAAGACGGGAGTTTGTCTGCGGAAACTACCCTGATCGGAGCAAATTGAACCGGCGAAGTTCAGACTAAATAGTCTTCACCCCGTTATACCCGCCCATAGGTTTTCGACTTCGGTGTAGGGTCTAAAGCACGTTGATCGAGCATATCAAGAGCTCAATACGAACTTCTTTT
**************************************************.........................((((((((((((........((((((((...((((((......))))))......)))........)))))......)))))).))))))..((((((.(((........)))...))).)))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
V053 head |
M047 female body |
|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................................................................AGGGGCTAAAGCACGGTCAT.................................. | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 |
| ...............................................................................................................................................................................................CGGTGTAGGGCCTACAGGAC........................................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 |
| ...................................................................................................................................CGGCTAAGTTCGGACTAG...................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:2155248-2155498 + | dvi_14364 | CTGTTCCAGTTCCTGCTCCTGGACCTGGCCCAATGGC-GCACACGGAGCAGGTATGTAGCTAAAAAAAAGAAAGAGTGCTCCCTTCTGCCCTCAAACAGACGCCTTTGATGGGACTAGCCTCGTTTAACTTGGCCGCTTCAAGTCTGATTTATCAGAAGTGGGGCAATATGGGCGGGTATCCAAAAGCTGAAGCCACATCCCAGATTTCGTGCAACTAGCTCGTATAGTTCTCGAGTTATGCTTGAAGAAAA |
| droMoj3 | scaffold_6540:24490764-24490814 - | CAGCTCCAGCAGCAGCGCCGGTAGCAGAACCGGCAGCAGCACCGACAGCAG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000391693:42509-42531 - | CCATTTCAGTTCCTACGCTTGGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000299277:254259-254277 + | CTATGCCGGCTCCCGCCTC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:6215080-6215128 + | ACA-----------------------------------------------------------------------------------------------GTGGCGGCTGATGATACTGAGCTCGGTTTACTGGCCCGCTTCAAGT------------------------------------------------------------------------------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
Generated: 05/17/2015 at 04:03 AM