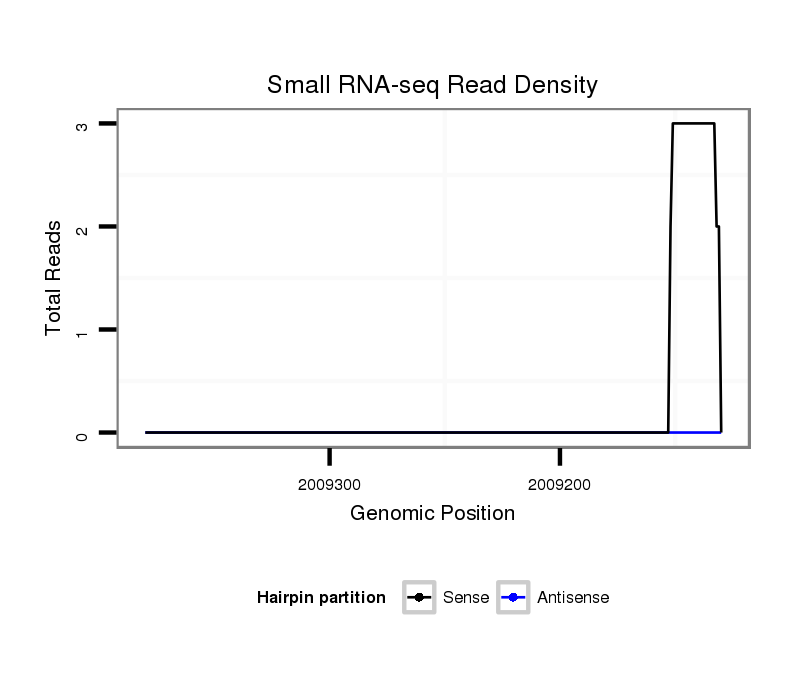
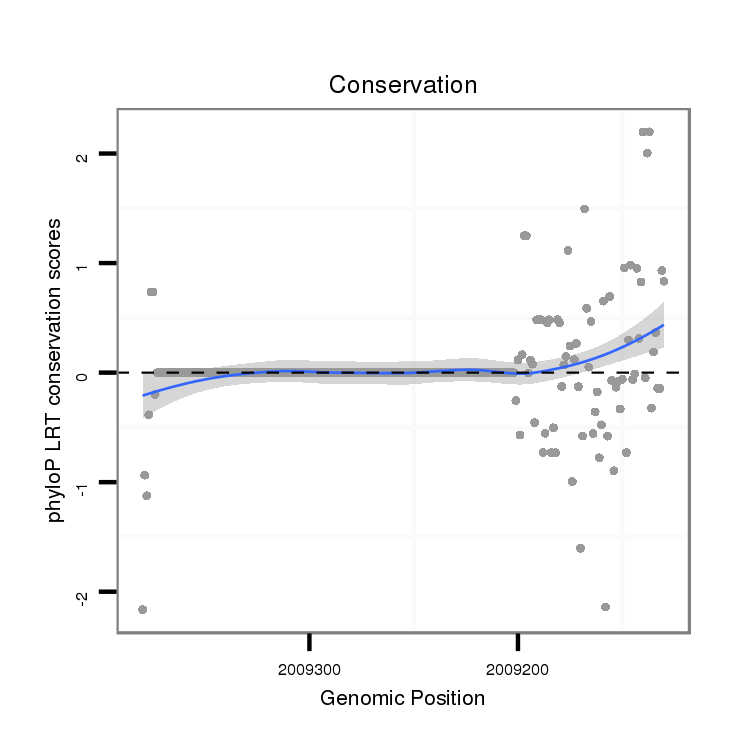
ID:dvi_14353 |
Coordinate:scaffold_13042:2009180-2009330 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_16134:2]; CDS [Dvir\GJ15696-cds]; intron [Dvir\GJ15696-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CACAAAGTATTTTTCATGGAAAACATTTTTTGGTCAACAGGTTGCTTTCAGGTTTGAACCGGCTTGTTTCACACGCGCCAGCACAATCTAGTTTGCGTTGCGATGTTTTCTCCTGGAAAATAATTTCAGGTCCGAGTCAACAAAGGAAATTTAGTAAATCGTCGAATGGATCTGAACAATATAAATAACATACTCTTGCAGAATCATGAGGAGAAGCGAAAGCTGAAGAAGAAGAGGAGGAGGAGGAAGAG **************************************************(((....)))((..((((((.((.((((.(((........))).)))))).))((((((((...))))))))..((((((((((..((.((...................)).)).))))))))))..........))))..)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060657 140_testes_total |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060658 140_ovaries_total |
GSM1528803 follicle cells |
SRR060655 9x160_testes_total |
SRR060669 160x9_females_carcasses_total |
M027 male body |
SRR060687 9_0-2h_embryos_total |
M047 female body |
SRR060659 Argentina_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................................................................................................................................................................................................................AGAAGAGGAGGAGGAGGAAGA. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................AAGAAGAGGAGGAGGAGGAAGA. | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................AAGAAGAGGAGGAGGAGGAA... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................CATGAGAAGAAGCGAAAGCTGAAG....................... | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................................AGAGGAGGAGGAGGAAGA. | 18 | 0 | 5 | 0.40 | 2 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................AGAAGAAGAGGAGGAGGAAGAAGA. | 24 | 1 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................AAGAGGAGGAGGAGGAAG.. | 18 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................AAGAGGAGGAGGAGGAAGA. | 19 | 0 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................AGGGGAAGCGAAAGCTAA......................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................................TGAAGAAGAAGAGGAGGA.......... | 18 | 0 | 20 | 0.15 | 3 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................GATATTTTCTCCTGG....................................................................................................................................... | 15 | 1 | 20 | 0.15 | 3 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................................AGAAGAGGAGGAGGATGTAGC. | 21 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................GAAGAAGAGGACGCGGATGAAGA. | 23 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................AATTCCGTGAATCGTCGAA..................................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................GAAAAAGAAGACGAGGAGGACG..... | 22 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................TTTGAACCGGGATGTTTGA.................................................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................GTAAGGGAGGAGGAGGAAGA. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................AATTCAGTGAATCATCGAAT.................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................................................................................................................TTCCGTGAATCGTCGAAT.................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...............................................................................................................................................................................................................................TTTAGAAGAAGAGCAGGAGGA....... | 21 | 3 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................CGATATTTTCTCCTCGAAA.................................................................................................................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................................AAGAAGAGGAGGAGGATGT.... | 19 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GTGTTTCATAAAAAGTACCTTTTGTAAAAAACCAGTTGTCCAACGAAAGTCCAAACTTGGCCGAACAAAGTGTGCGCGGTCGTGTTAGATCAAACGCAACGCTACAAAAGAGGACCTTTTATTAAAGTCCAGGCTCAGTTGTTTCCTTTAAATCATTTAGCAGCTTACCTAGACTTGTTATATTTATTGTATGAGAACGTCTTAGTACTCCTCTTCGCTTTCGACTTCTTCTTCTCCTCCTCCTCCTTCTC
**************************************************(((....)))((..((((((.((.((((.(((........))).)))))).))((((((((...))))))))..((((((((((..((.((...................)).)).))))))))))..........))))..)).......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR1106721 embryo_10-12h |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| .............................................................CGGACAAAGTGTGCGGGGT........................................................................................................................................................................... | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................TCTTCCCTTTCGACTTTTCCTTC................. | 23 | 3 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 |
| ............................................................TCGGCCAAAGTGTGCGCGGTC.......................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| .....................................................................................................CTACAAAAGAGGTGCTTATA.................................................................................................................................. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 1 | 0 |
| ................................CAGTTGTCCAACGAAAAG......................................................................................................................................................................................................... | 18 | 2 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 |
| .............................................................CGACGAAAGTGTGCGTGGTC.......................................................................................................................................................................... | 20 | 3 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:2009130-2009380 - | dvi_14353 | CACAAAGTATTTTTCATGGAAAACATTTTTTGGTCAACAGGTTGCTTTCAGGTTTGAACCGGCTTGTTTCACACGCGCCAGCACAATCTAGTTTGCGTTGCGATGTTTTCTCCTGGAAAATAATTTCAGGTCCGAGTCAACAAAGGAAATTTAGTAAATCGTCGAATGGATCTGAACAATATAAATAACATACTCTTGCAGAATCATGAGGAGAAGCGAAAG----------CTGAAGAAGAAG-AGGAGGAGGAGGAAGAG |
| droMoj3 | scaffold_6359:3902755-3902830 + | ACAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TAAAAATGCAATAAACTGAAAGGGTCACAAGCCGCAGCAAAAG----------CAGAAGAAGAGG-AGGAAGAAGAAGAAGCG | |
| droGri2 | scaffold_14832:2545-2586 + | GAAG-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACGAAATGG----------ATGAGGAAGAGG-AGGAGGAGGAGGAGGAG | |
| droWil2 | scf2_1100000004515:854026-854073 + | CAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGAAAAACA-----------------------------GTGG----------CTAAAAGAGAAGGAGGAGGAGGAGGAGCAG | |
| droPer2 | scaffold_4:3559742-3559767 - | AGGA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGG-AGATGAAGGAGCAGCAG | |
| droKik1 | scf7180000302698:593000-593042 + | AGAG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGGAGGAAG----------ACGAGGATGAGG-AGGAGGACGATGAAGAG | |
| droFic1 | scf7180000453839:2002029-2002095 - | GCGGAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGAAAACA----------------------GGAAACGAAACTTCAAGCCCAGCGAAGAAGAAG-ACAAGGAAGAACTAGAG | |
| droRho1 | scf7180000777356:7045-7094 + | CGGA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACCGGAAAGAGAA---GCGG----------CCGCAAGGGAAG-TAGAAGAGGAAGCAGAG | |
| droEug1 | scf7180000409474:440241-440290 - | AGCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCATGAGGAAGACGAGG----------ATGAGGAAGAGG-AGGAGGAGGAGGAGGAG | |
| droYak3 | 3L:17455116-17455172 + | GCAG-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACCGGAAACGGAAGCGACAGTAG-------CAGGCCGGAAAC-AGGAGGAGGAGGAAAAA | |
| droEre2 | scaffold_4784:17212668-17212721 + | GCAG-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AACCGGAAACGGAAGCGACAG----------TCGCACACCGGA-AACAGGAGGAGGAAAAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:59 AM