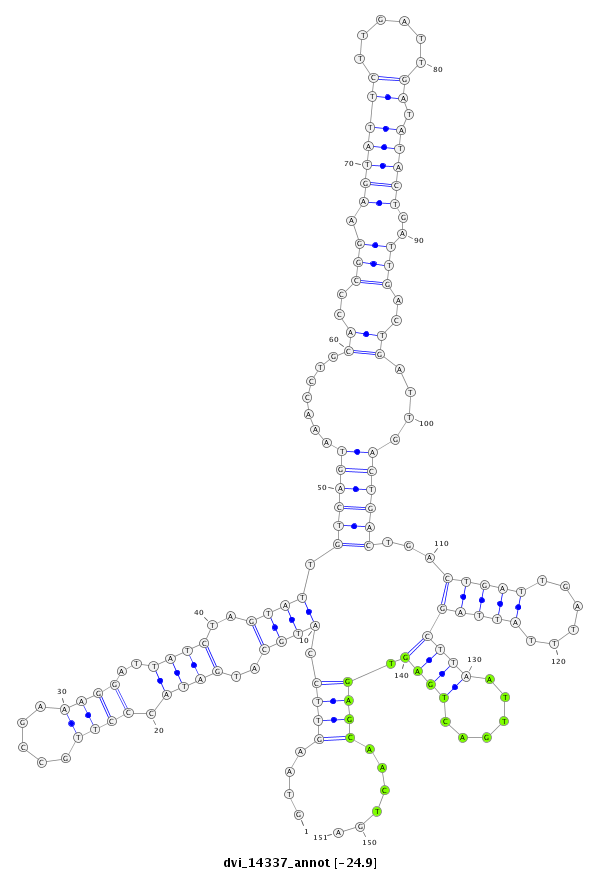
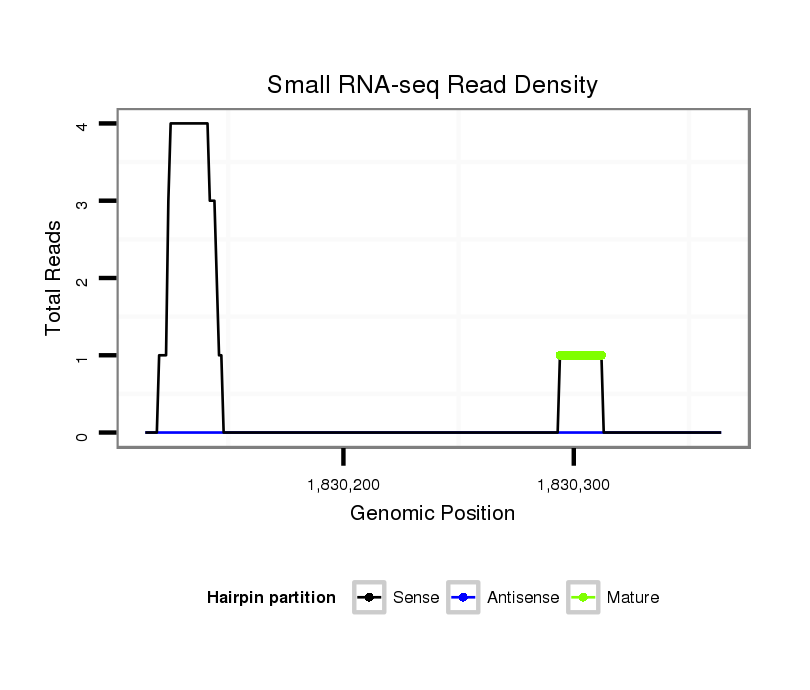
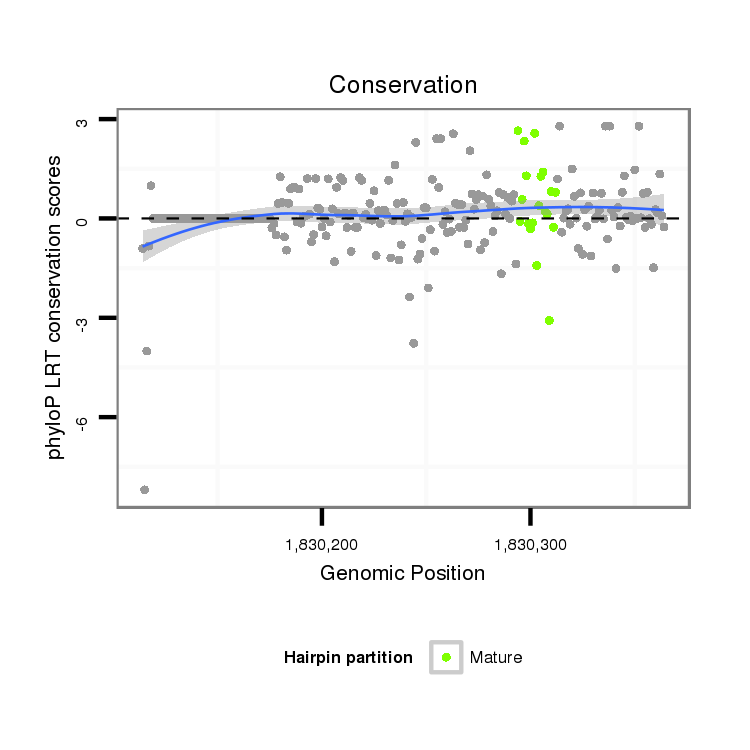
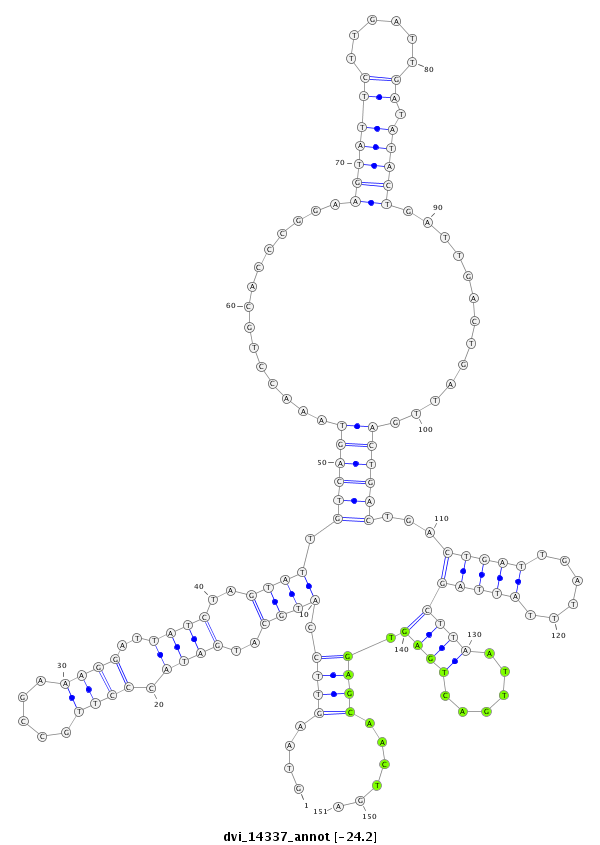
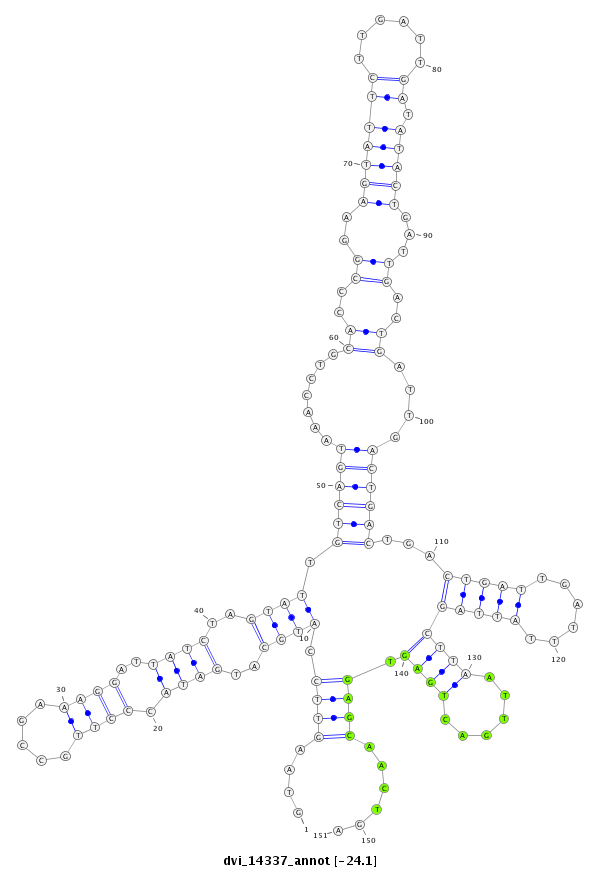
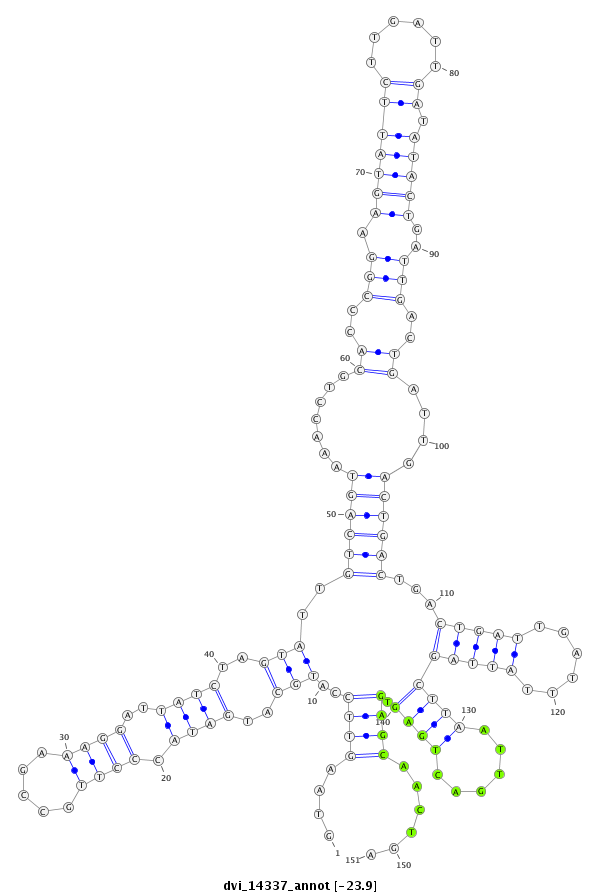
ID:dvi_14337 |
Coordinate:scaffold_13042:1830164-1830314 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -24.2 | -24.1 | -23.9 |
 |
 |
 |
CDS [Dvir\GJ15870-cds]; exon [dvir_GLEANR_16302:3]; intron [Dvir\GJ15870-in]
| Name | Class | Family | Strand |
| (ACTG)n | Simple_repeat | Simple_repeat | + |
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- CACCAGCGGACTTTGCCGATGGCACCTTCTCCTTGTCCAATATTGGCATCGTAAGTTCCATGCATGATACCCTTGCCGAAAGGATTATCTAGTATTGTCAGTAAACCTGCACCCGGAAGTATTCTTGATTGATATACTGATTGACTGATTGACTGACTGACTGATTGATTTATTAGCTTAATTGACTGAGTGAGCAACTGAACGATTGTCTTGCTCGTTGCTTGACTAATTGACTTACTGATTGTTTGATT **************************************************....((((.((((..((((.((((.....))))..))))..)))).((((((.......((..(((.(((((((......)).)))))..)))..))....))))))...(((((......)))))((((......)))).))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060686 Argx9_0-2h_embryos_total |
GSM1528803 follicle cells |
M028 head |
M061 embryo |
SRR060655 9x160_testes_total |
SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060672 9x160_females_carcasses_total |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
V047 embryo |
V116 male body |
SRR060661 160x9_0-2h_embryos_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..................................GTCCAATATTGGCATCGTGGGC................................................................................................................................................................................................... | 22 | 3 | 1 | 2.00 | 2 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........CTTTGCCGATGGCACCTTCTC............................................................................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........CTTTGCCGATGGCACCTTCTCCTT......................................................................................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................CCTGTACCCGAAAGTATTCATG............................................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......CGGACTTTGCCGATGGCACCTT............................................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........TTTGCCGATGGCACCTTCTCC........................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................ATTGACTGAGTGAGCAACT.................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................GTCCAATATTGGCATCGTGGG.................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................AACAAGCACCCTGAAGTAT................................................................................................................................. | 19 | 3 | 18 | 0.17 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................ATACGGATTGACAGATTGAG.................................................................................................. | 20 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................................................................ATAAGGATTGACAGATTGACTG................................................................................................ | 22 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................ATATTAGCTTAATTGATAGAG............................................................. | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................ATACGGATTGACAGATTGA................................................................................................... | 19 | 2 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................AGTAAGCCTGCTCCGGGA...................................................................................................................................... | 18 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................GACTGATTGACTGATTGATTTAT.............................................................................. | 23 | 1 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................ATACGGATTGACAGATTG.................................................................................................... | 18 | 2 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................TATAAGGATTGACAGATTGA................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........TTTTGCGGATGGCACCTTT.............................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ....................................................................................................................................................................GTGATTTGTCAGCTTAATTG................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
GTGGTCGCCTGAAACGGCTACCGTGGAAGAGGAACAGGTTATAACCGTAGCATTCAAGGTACGTACTATGGGAACGGCTTTCCTAATAGATCATAACAGTCATTTGGACGTGGGCCTTCATAAGAACTAACTATATGACTAACTGACTAACTGACTGACTGACTAACTAAATAATCGAATTAACTGACTCACTCGTTGACTTGCTAACAGAACGAGCAACGAACTGATTAACTGAATGACTAACAAACTAA
**************************************************....((((.((((..((((.((((.....))))..))))..)))).((((((.......((..(((.(((((((......)).)))))..)))..))....))))))...(((((......)))))((((......)))).))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060665 9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060666 160_males_carcasses_total |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|
| ................................AACAGGGTATAAACGTAGCAA...................................................................................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 |
| ...................................CCGTTATAACCGTAGCACTC.................................................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................AACTGACTAACTGAATGACTGACT....................................................................................... | 24 | 1 | 20 | 0.10 | 2 | 0 | 0 | 2 | 0 | 0 |
| ..........................................................................................................................................CTAACTGACTAACTGAATGACTGACTA...................................................................................... | 27 | 1 | 17 | 0.06 | 1 | 0 | 0 | 1 | 0 | 0 |
| .........................................................................................................................................ACTAACTGACTAACTGAATGACTGACT....................................................................................... | 27 | 1 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................................................TGACTAACTGACTAACTGAATGAC............................................................................................ | 24 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 |
| ..........AAAACGACTACCATGGAAG.............................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:1830114-1830364 + | dvi_14337 | CACCAGCGGACTTTGCCGATGGCACCTTCTCCTTGTCCAATATTGGCATCGTAAGTTCCATGCATGATACCCTTGCCGAAAGGATTATC-TAGTA-----------------------------------------------------TT------------GTCA-GTAAACCTGCACCCG-----GAAGTATTCTTGATTGATATACTGATTGACT--------GATTGACTGACTGACTGATTGATTTATTAGCTTAATTGACTGAGTGAGCAACTGAA----CGAT---------------------TG----------------TCTTGC--------TC----------------GTTGCTTGACTAATT----------------GA-------CTTACTGATTGTTTGATT- |
| droMoj3 | scaffold_6540:30384004-30384202 - | ACG-----------------------------------------------------------CACGATTGCCTTGCTGGAAAAATTGTAGTAGAAGCAGTAGTAGTAGTAAAATAGACAT------------------------------------------------------TGCACGCA-----GAA-GATTGTTGATTGACTG----ACTGAGACACT----GACTGATTG----TCTGACGGAATGGCTGA-TTGATTGAATGAATGATCCACCGAC----TGAT---------------------TG----------------GTTGGT--------AA----------------AATAACTAACTAGCT----------------GG-------CTTATTAATAAGGTGATT- | |
| droGri2 | scaffold_15126:2881955-2882102 - | CAAAA---------------------------------------------------------------------------------------------------------------AGCCAACAGCCAAAAGCCAACTGGCTG------------------------------------------------------------ACTGACTGACTGACT--------GGCTGACTGGCTGACTGACTGACTGAC-----------------TGACTAACTGAC----TGAC---------------------TG----------------ACTGAC--------TG----ACTGACTGACTGACTGACTGACTGGTT----------------GA-------GTTACTGCTTGACAAACCG | |
| droWil2 | scf2_1100000004511:6362220-6362313 + | CTG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACTGACTGAGTGAATGA-CTGACTGACTGACTGCCTGAATGAG----TGAC---------------------TG----------------ACTTGT--------TA----------------ACTGACTGACTAACA----------------GC-------CTGACTGACTGTTTGACT- | |
| dp5 | XL_group1e:5228709-5228893 + | TGA-----------------------------------------------------------------------------------------------------------------------------------------CGGATAGATTGACTGACTGACAGATAAAAGGAC----TCACT-----GAAGGATAGATGAT----------------------------TG--------ACTCAATGACTGACTGA-CTGAAGGATATATTGACTAACTGAT----GGTTGGACTTCATTCGGTAAATGAATGAATGACGGTTGGTTGGTTGGAC--------TC----------------ACTGACTGACGGATT----------------GA-------CGGATTGACTGATTGACT- | |
| droPer2 | scaffold_15:638346-638454 - | TTG-----------------------------------------------------------------------------------------------------------------------------------------CTG------------------------------------------------------------AATGACTGACTGACT--------GACTG----------------ACTGACTGA-CTGACTGACTGACTGACCGATTGAC----TGAC---------------------TG----------------TTTGAT--------TC----------------ACAGACTCACTGAGT----------------GA-------ATGACTGAATGAGTGACT- | |
| droAna3 | scaffold_13335:1748853-1748974 - | TTG-----------------------------------------------------------AT-----------------------------------------------------------------------------TG---------------------------------------------------------ACTATCTGCGGATTGACT--------AACTGATTGATTGACTATCTGACTGATTGA-CTAACGGACTGATTGACTAACTGAC-----------------------------TG----------------ATTGAC--------TA----------------ACTGACTGATAGACT----------------GA-------CTGACTGACTGACCG---- | |
| droBip1 | scf7180000394714:86151-86292 - | AATA--------------------------------------------------------------A-----------------TTATT-TTGT-------------------------------------------------------------------------------------------------GATTTTTTTTTGATAA----ACTCACAAACTGAATGATTG--------ACTAATTGAATGATTGA-CTAACTGAATGATTGACTAACTGAT----TGAT---------------------TG----------------ATTGAC--------TA----------------ACTGACTAACTGACT----------------GA-------CTGACTGACTGACTGACCG | |
| droKik1 | scf7180000302802:55720-55900 - | ACC--------------------------------------------------------------------CTTTCCGAAGGAATAACTGTAGGA-----------------------------------------------------TTGACTATTTCACTGATTTCAGGACTAT----TTTACTAATCGCGTTACTAC--------T--------T--------GTTTCACTAATTGTTTCACTAACGGTTTCA-CTAACTGTCTCACTAACTGTCTCAC----TAAC---------------------TG----------------TCTCAC--------TA----------------ACTGTCTCACTAACT----------------GT-------CTCACTAACTGTCTCACT- | |
| droFic1 | scf7180000453872:503546-503649 - | TTG-----------------------------------------------------------CC-----------------------------------------------------------------------------TG--------------------------------------------------------------------ATTGACT--------CACTG----------------GCTGACTGA-CTGACTGACTGATTGACTAATTGACTAAATGAC---------------------TG----------------ATTGAC--------TAACTG------------GCTGATTGACTTGCT----------------GA-------CTGACTGACTGA------- | |
| droEle1 | scf7180000490276:4206-4334 - | TTA-----------------------------------------------------------------------------------------------------------------------------------------TTG--------------------------------------------------------ATTTATTGATTTATTGATT--------TATTGATTTATTGATTTATTGATTTATTGA-TTTATTGATTTATTGATTTATTGAT----TTAT---------------------TG----------------ATTTAT--------TG----------------ATTTATTGATTTATT----------------GA-------TTTATTGATTTATTGATT- | |
| droRho1 | scf7180000779914:688103-688227 + | TGA-----------------------------------------------------------------------------------------------------------------------------------------TTG------------------------------------------------------------ACTGACTGACTGACT--------GACTGACTGACTGACTGACTGACTGATTGA-CTAACTGACTGATTGACTAACTGAC----TGAT---------------------TG----------------ACTAAC--------TG----------------ACTGATTGACTAACT----------------GA-------CTGACTGACTGACTGACT- | |
| droBia1 | scf7180000301760:732267-732383 - | TGG-----------------------------------------------------------------------------------------------------------------------------------------GTG------------------------------------------------------------ACAGACTGACTGACT--------AGCTGACCAATTGGCTGACTGACTGAGAGG-CCAAC--------AGACAGACTGAC----TGGC---------------------TG----------------ACTGGC--------TG----------------ACTGATTGACTGACT----------------GA-------CTGACTGATTGATTGACT- | |
| droTak1 | scf7180000415734:40335-40463 - | TTT-----------------------------------------------------------------------------------------------------------------------------------------TTA--------------------------------------------------------ATTGACTGACAGACTGACA--------GACTGACAGACTGACAGACTGACAGACTGA-CAGACTGACAGACTGACAGACTGAC----AGAC---------------------TG----------------ACAGAC--------TG----------------ACAGACTGACAGACT----------------GA-------CAGACTGACAGACTGACA- | |
| droEug1 | scf7180000409540:11556-11680 - | TGA-----------------------------------------------------------------------------------------------------------------------------------------CTG------------------------------------------------------------ACTGACTGACTGACT--------GACTGACTGACTGACTGACTGACTGACTGA-CTGACTGACTGACTGACTGACTGAC----TGAC---------------------TG----------------ACTGAC--------TG----------------ACTGACTGACTGACT----------------GA-------CTGACTGACTGACTGACT- | |
| dm3 | chrUextra:10723625-10723753 + | TGA-----------------------------------------------------------------------------------------------------------------------------------------CTA------------------------------------------------------------AATGACTAAATGACT--------AAATGACTAAATGACTGAATGACTAAATGA-CTAAATGACTAAGTGACTAAGTGACTAAATGAC---------------------TA----------------AATGAC--------TA----------------AATGACTAAATGACT----------------AA-------ATGACTAAATGACTAAAG- | |
| droSim2 | 3r:9922312-9922454 - | CAG-----------------------------------------------------------------------------------------------------------------------------------------TTT--------------------------------------------------------AATTACGCCCAGCATGATT--------AACTGATTAACTG--------GCAAACTGA-CTAACTGACTGACTGACTGACTGAC-----------------------------TA----------------ATTGACTAACTGACTA--------------------ACTAACTGACTAACTGACTGACTGCCTGGCTGCTGGCCAGTTAGCTACTTGGC-- | |
| droSec2 | scaffold_37:101998-102178 + | TTG-----------------------------------------------------------AC-------------TAAGTGA----------------------------------------------------------------CTAACTGACTAACTGACA-GAAAACTAG----CTGACTGGACGACTTAGCGACTAACTGACCAAATGACT--------AGCTGGCTACCTGACAAAGTGACTAACTGA-CTAACTGACAGACAGACTAGCTGAC----TAGC---------------------CG----------------ACTTAG--------CG----------------ACGAACTGACAAACT----------------GA-------CTAGCTGGCTAACTGACA- | |
| droYak3 | X:10569159-10569302 + | CAGTA-------------------------------------------------------------A-----------------CTGC---TAC--------C-------------AGTCGTCT----------------------------------------------------------------------------GCCAGAGACGTATTGACT--------GACTGACTGACTAACTGACTAACAGATAGA-CTAACTGACAGACTGACTAACGGAC----AGAC---------------------TG----------------ACTAAC--------GG----------------ACAGACTGACTAACG----------------GA-------CAGACTGACTAACAGACT- | |
| droEre2 | scaffold_4690:8212263-8212389 + | CC-------------------------------------------------------------------------------------------------------------------------------------------TG--------------------------------------------------------ATTGACTCACTGACTGAAT--------GACTAGCTGGCTGACTAGCTGGCTGACTAA-CTGACTGACGGACCGACTGACTGAC----TGAC---------------------TG----------------ACTGAC--------TG----------------ACTGACTGACTGACT----------------GA-------CTGACTGACTGACTGACT- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 03:43 AM