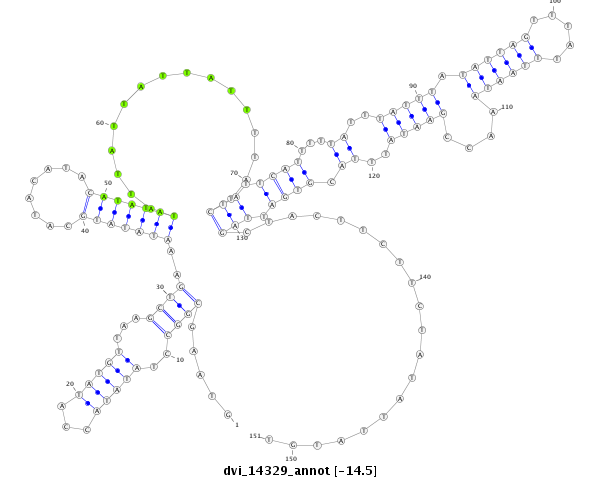
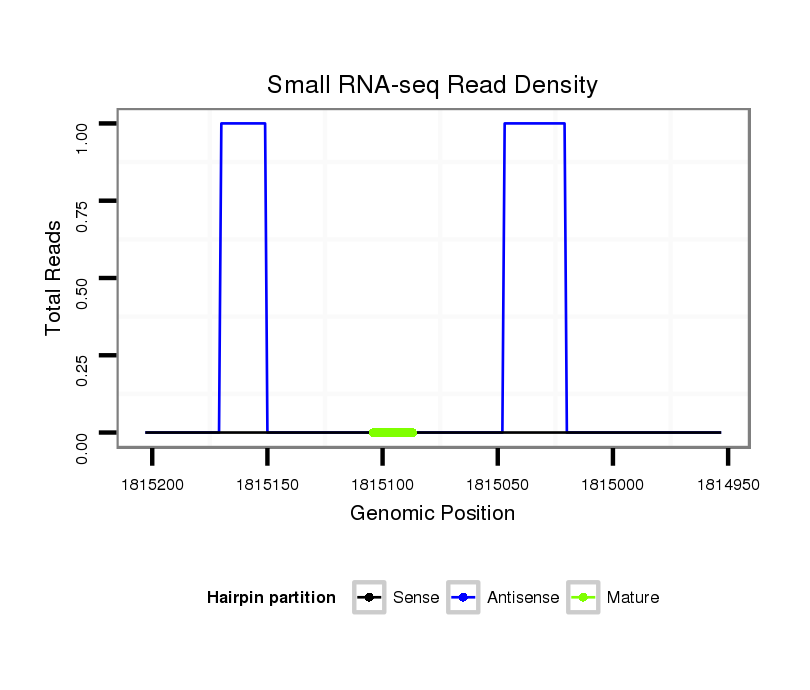
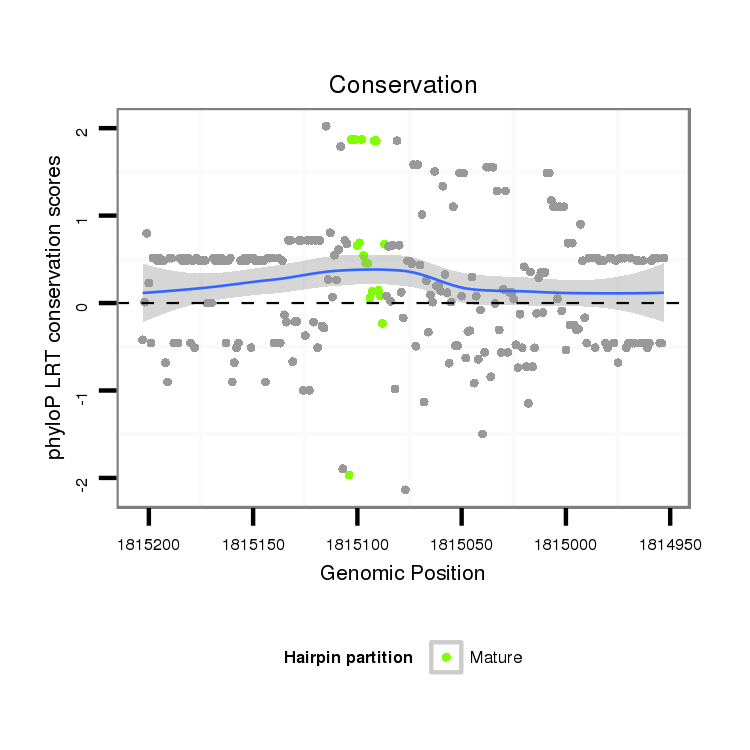
ID:dvi_14329 |
Coordinate:scaffold_13042:1815003-1815153 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -14.1 | -14.0 | -13.9 |
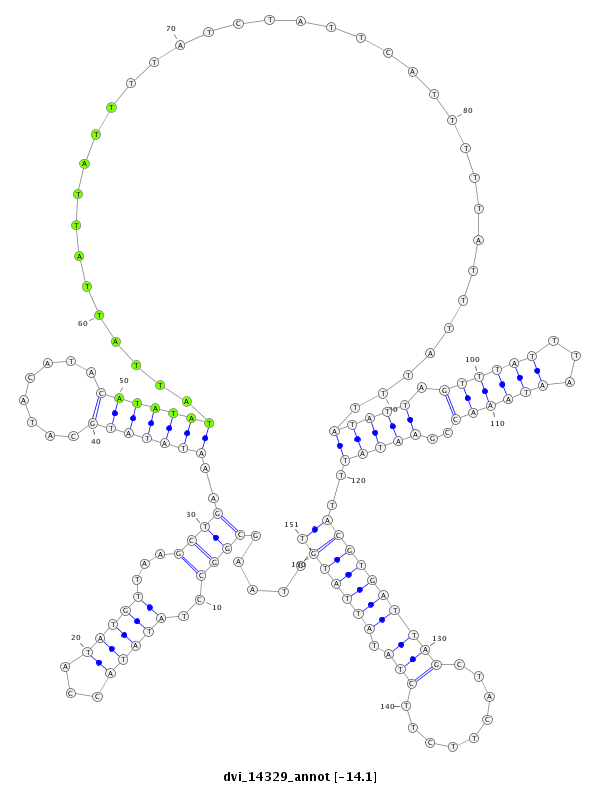 |
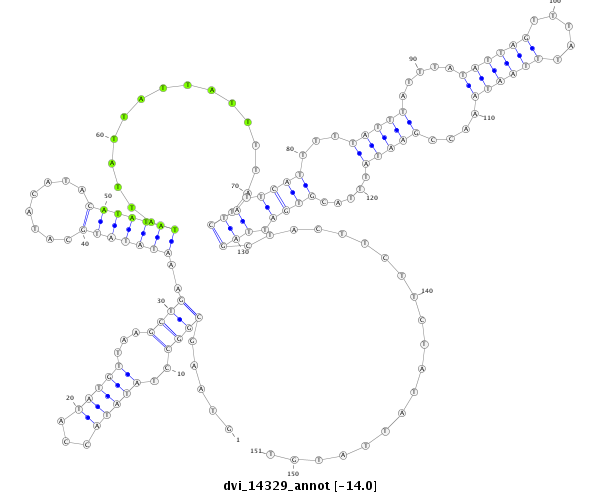 |
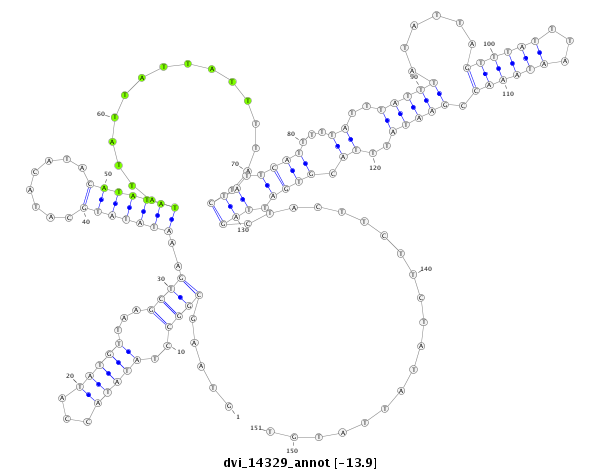 |
CDS [Dvir\GJ15704-cds]; exon [dvir_GLEANR_16141:1]; intron [Dvir\GJ15704-in]
| Name | Class | Family | Strand |
| A-rich | Low_complexity | Low_complexity | + |
| ----------------------############################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TTACCAACACCGGTTGCGTGAGATGTTCGCCAACACAGTGCCTGCAAGCAGTAAGCGGCCTATATACCATATGTTAAGCTGAAATATATGCATACATACATATATATTATTATTATTTTATCTATTCATTTTTATTTATTTATATTAGTTTATTTAATAAACCGAATATTTACGTGATTAGCTACTTCTTCTATATTATGTATTTCACTTAACCTTAAAGCCCATAAGTAATAGTTGGCGCTTGCAATCGT **************************************************.....((((..(((((...)))))...))))..(((((((........)))))))................(((.((((...((..(((((.((((((.....))))))....)))))..)).)))).)))....................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060687 9_0-2h_embryos_total |
V053 head |
SRR060663 160_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
M047 female body |
SRR060684 140x9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .....................................GTGCGTGCAAGCAGTAAGT................................................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................TACATCCATATATATTAATATTATG...................................................................................................................................... | 25 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................CGCAAACAGAGTGCCTGGAA............................................................................................................................................................................................................ | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........CCGTTGCGTGAGATTTTGGC............................................................................................................................................................................................................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............GCATGCGATGGTCGCCAACA........................................................................................................................................................................................................................ | 20 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................................................GGCCGATTATTTACGTGAT......................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........CGTGTCGTGAGATGTTCG.............................................................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................ATATATATTATTATTATT...................................................................................................................................... | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AATGGTTGTGGCCAACGCACTCTACAAGCGGTTGTGTCACGGACGTTCGTCATTCGCCGGATATATGGTATACAATTCGACTTTATATACGTATGTATGTATATATAATAATAATAAAATAGATAAGTAAAAATAAATAAATATAATCAAATAAATTATTTGGCTTATAAATGCACTAATCGATGAAGAAGATATAATACATAAAGTGAATTGGAATTTCGGGTATTCATTATCAACCGCGAACGTTAGCA
**************************************************.....((((..(((((...)))))...))))..(((((((........)))))))................(((.((((...((..(((((.((((((.....))))))....)))))..)).)))).)))....................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060673 9_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................GTGTCACGGACGTTCGTCAT...................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................TATTTGGCTTATAAATGCACTAATCGA.................................................................... | 27 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................ATGATAGTAAAATAGATAAGT........................................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................AGAGACTTGGGATTTCGGGT........................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................ATAATAATAATAAAGTAGA................................................................................................................................ | 19 | 1 | 12 | 0.17 | 2 | 0 | 0 | 0 | 1 | 1 | 0 | 0 |
| .......................................................................................................TATTATAATAATAATATAGATAA............................................................................................................................. | 23 | 2 | 14 | 0.14 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_13042:1814953-1815203 - | dvi_14329 | TTACCAACACCGGTTGCGTGAGATGTTCGCCAACACAGTGCCTGCAAGCAGTAAGCGGCCT--------------------------------------------------------ATATACCATATGTTAAGCTGAAATATATGCA--TACATACATAT--ATATTATTATTATTTTATCTATTCATTTTTA--------------------T--------TTATTTATAT------------------TAGTTTATTTAATAAACCGAATATTTACGTGATTAGCTAC-----------TTCTT-CTATATTATGTATTTCACTTAACCTTAAAGCCCATAAGTAAT---AGTTGGCGCTTGCAATCGT |
| droMoj3 | scaffold_6500:16690588-16690710 + | TAAT------------------------------------------------------------------------------------------------------------------------GTATTTTAATTTTAAA-AAGTAT----ATACATTTAT--ATATTATAATTATTTCATTTAATT----------------------------TTTTTAGTGTATTT----------------------TAT----TTTTTATTATATCATGTTTGAATTATTTTCAAT-----------ATTCT-TTATATT--------------A------------------------------------------ | |
| droGri2 | scaffold_14853:3199238-3199468 - | TTACTAACACCTCTTATATGAGACGATCGCC---ACAGTGTCTCATGGCAGTTAGCGGCGTATCTCATATTGGTTTTGTACAATGTAATTGATCATTTCTGTGTGTAAATATTGAATATGCATCAAATTCCAAATTAATAAATGTAT----ATACATGTAT--AAA------------------------------TGTATATTTTATTTAAGT-TTGTTAACAAG-----------------------------------------------------------------------------------------------------CAATTTTATAGCCTTTAGGGAATTTATATCAGCACCAACAATTAT | |
| droWil2 | scf2_1100000004511:4779272-4779431 + | ATAT------------------------------------------------------------------------------------------------------------------------A-------------------TATA--TATATATATAT--ATATATTTATATATATATATATAAATTTGTGTGCGTG----------------------------TGTATGTATGTGCGTGTGGATGACTGTTCGTTTGTTTGACTGTATGTATGCGTGGCGAAAATTCGTTGCCACGATTTGCCTATAATTATGTGTGTTATATAAC----------------------------------------- | |
| dp5 | XR_group6:10332915-10332997 + | T-AT------------------------------------------------------------------------------------------------------------------------T-------------------TATA--TATATATATAT--TTATATTTATTATTATATTTATTTATTTATAT---------------------------------------------------------------TATTTATTTTTTCATAT--------------AT-----------ATTTT-TTTTATT--------------A------------------------------------------ | |
| droKik1 | scf7180000302517:608923-608969 - | ATTT------------------------------------------------------------------------------------------------------------------------G-------------------TATT--AT------------------TATTATTATGATTTATTATTTTTA--------------------T--------ATATATATTT------------------T------------------------------------------------------------------------------------------------------------------------ | |
| droEug1 | scf7180000409048:159855-159896 + | T-AT------------------------------------------------------------------------------------------------------------------------A-------------------TATA--TATATATATAT--ATATATATATTAGATTTTCTATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dm3 | chrX:14973156-14973217 - | ATAT------------------------------------------------------------------------------------------------------------------------A-------------------TGTA--TGTATGTATAT--ATACATATATTCATATGTATATACATTTATATATGTA----------------------------TACAT------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_53:125154-125257 + | T-AT------------------------------------------------------------------------------------------------------------------------A-------------------TATTATTA--CATTTATATATA------------------TTATATTATA--------------------T--------TTTATTATAT------------------TATATATTATTAATATATATATATATATGTAATTTAATAA-----------TTATA-TTATAT-ATATATA-------T------------------------------------------ | |
| droYak3 | X:9164749-9164817 - | AAAT------------------------------------------------------------------------------------------------------------------------A-------------------TATACATA--GATGTATACATATATTAATACATATGTATATATATATATA--TGTC--------------------------------------------------------------------TTTATGT--------------AT-----------ACT------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/17/2015 at 03:40 AM