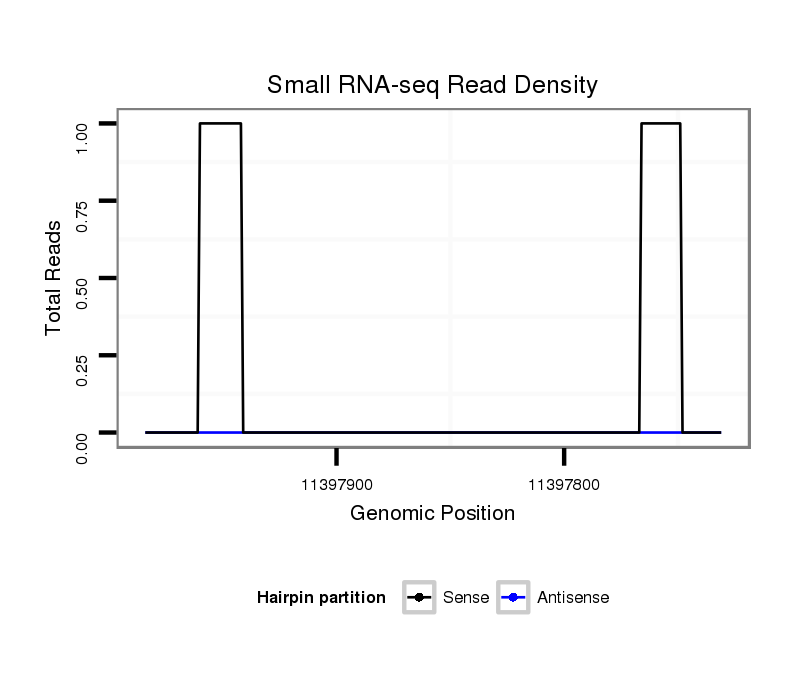
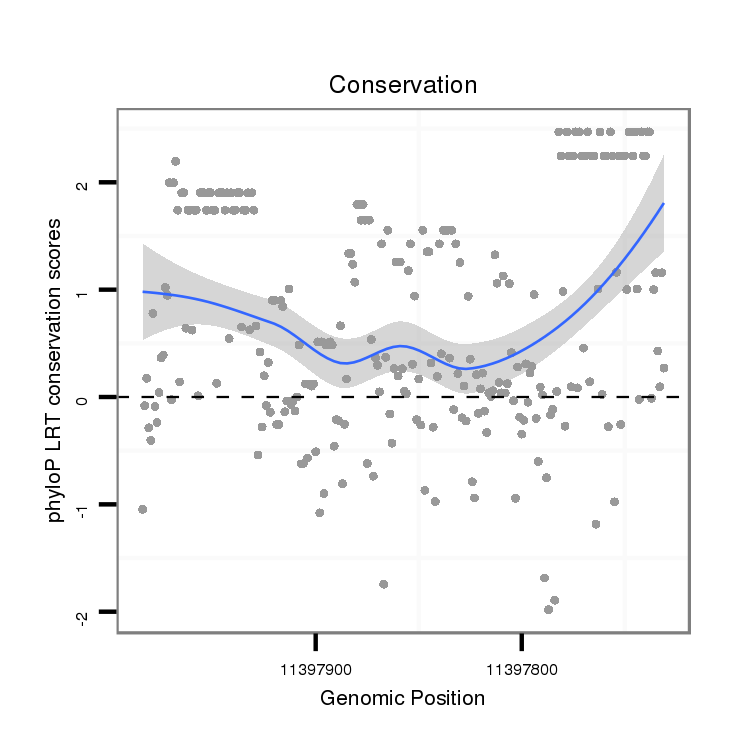
ID:dvi_14005 |
Coordinate:scaffold_12970:11397781-11397934 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_3441:3]; CDS [Dvir\GJ18609-cds]; exon [dvir_GLEANR_3441:2]; CDS [Dvir\GJ18609-cds]; intron [Dvir\GJ18609-in]
No Repeatable elements found
| ##################################################----------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CTACTATCGGGACGCCACCAAGCCGCCCAATCATCGTAAAGTCTGCAAACGTAAGTGCTGTTAAAGCTGTCACCTGGTGATGTTAAGACGCTGCTCGCTCATCAATCAGCGGCCTAGTTAGTTGTCAACTGGGAAAATATCTTTCTAATGATGACACCTTCTTTCCTTCCCTCTCTCTCTCTTTTCTTCTACCCTGTCTCATAGGCTGCGACTGCCATCCCGTGGGCTCGACTGGCAAGACCTGCAATCATCTG **************************************************...(((((.(((..(((.(((((...))))))))..))))).)))((((........)))).(((((((.......)))))))..............((((.((((......................................))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060657 140_testes_total |
SRR060670 9_testes_total |
V053 head |
SRR060656 9x160_ovaries_total |
SRR060660 Argentina_ovaries_total |
SRR060689 160x9_testes_total |
M027 male body |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................TGGGATGTTAAGACGGTGCTCG............................................................................................................................................................. | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................CCCGTGGGCTCGACTGGC.................. | 18 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................GCCCAATCATCGTAAAGTC................................................................................................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................AAACCCTGTCTCCTAGGCTGC............................................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................TTCTGTCCTTGCCTCTCTCT............................................................................ | 20 | 2 | 6 | 0.33 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| ..................................CGTAAAGTCTGGAAACGCGA........................................................................................................................................................................................................ | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................GCCGGGGGCTCGACTGGGAA................ | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................................GCCGTACCGTGGGTTCGA....................... | 18 | 3 | 16 | 0.13 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 |
| ......................................................................................................................................................................................................................CTATCCGGTGGGCTGGACT..................... | 19 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...................................................................................................................................................ATGATGACAGCTTCTTGCT........................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GATGATAGCCCTGCGGTGGTTCGGCGGGTTAGTAGCATTTCAGACGTTTGCATTCACGACAATTTCGACAGTGGACCACTACAATTCTGCGACGAGCGAGTAGTTAGTCGCCGGATCAATCAACAGTTGACCCTTTTATAGAAAGATTACTACTGTGGAAGAAAGGAAGGGAGAGAGAGAGAAAAGAAGATGGGACAGAGTATCCGACGCTGACGGTAGGGCACCCGAGCTGACCGTTCTGGACGTTAGTAGAC
**************************************************...(((((.(((..(((.(((((...))))))))..))))).)))((((........)))).(((((((.......)))))))..............((((.((((......................................))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060658 140_ovaries_total |
SRR060679 140x9_testes_total |
SRR060664 9_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060666 160_males_carcasses_total |
V116 male body |
SRR060673 9_ovaries_total |
V047 embryo |
M047 female body |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
SRR060663 160_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................AGCATTTCAGACCTTTGC........................................................................................................................................................................................................... | 18 | 1 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................TGGTTCGAAGGGTCAGTAG........................................................................................................................................................................................................................... | 19 | 3 | 20 | 0.85 | 17 | 0 | 0 | 0 | 1 | 2 | 5 | 0 | 0 | 0 | 1 | 2 | 2 | 1 | 1 | 1 | 1 |
| ............................................................................TACTACAATTCTGGGACG................................................................................................................................................................ | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................GGAAGAAAGGTAGGTAGAGAG............................................................................. | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................GGAAGAATGGAAGGGAGCGAT............................................................................. | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........CTGGCGGTGGTTCCGCGGGTTA............................................................................................................................................................................................................................... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................................GGCACCCGAGCTGACAGA................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................CGCATTTCAGACCTTTGC........................................................................................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................GGAGAGGTAGAGAACAGAAGA................................................................ | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................GGTAGAGAACCCTAGCTGA..................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................CGTAGTTACCCGCCGGATC......................................................................................................................................... | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................................CCCATCTGGACGTTAGAA... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................AAGAGGAGGGGACAGAGTAG................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:11397731-11397984 - | dvi_14005 | CTACTATCGGGACGCCACCAAGCCGCCCAATCATCGTAAAGTCTGCAAACGTAAGTG-CTGTT-------------------------------------------------------------------AAAGCTGT---------------------------------------------------CACCTGGTGATGTTAA---GACGCTGC-----TCGCTCATCAATCAGCGGCCTA-------------------GTTAGTTGTCA--------------ACTGGGA-------AAATAT----------------------CTT----TCTAATGATG---ACACCTTCTTTCCT----------TCCCTCTCT-----------CTC-----------TCTT--------------T--TCTTCTACCCTG-------------------T---------------C-TCATAGGCTGCGACTGCCATCCCGTGGGCTCGACTGGCAAGACCTGCAATCATCTG |
| droMoj3 | scaffold_6473:9893199-9893441 + | CTACTACCGGGATGCCACCAAGCCACCCAATCATCGCAAAGTGTGCAAACGTAAGTAGCAGCT------------------------------------------------------------------------------------------------------------------------------------------------GT-----------TTTGGCCATCAATCAGGGGCCTA-------------------GTTAACTGTCACACA-------------GC---------ACAGAT----------------------GTCTCTCTCTAATGATG---ACTC--TCCCTCTC-TCC-TCTCTCCGCTCTCTTCTGTTTGTGTCTC-----------TCTT--------------T--TGCTCTA-------------------------------------TCTC-TTTTAGGCTGTGACTGTCATCCCGTGGGCTCCACCGGCAAGACGTGCAATCATCTG | |
| droGri2 | scaffold_15203:3382385-3382679 - | CTACTATCGGGATGCCACCAAGCCGCCCAATCATCGCAAAGTCTGCAAACGTAAGTG-A-------------------------------------------------------------------ATGTGAAGCTGTTTATCCTG-------------------------------------------------GCAAAGCTTACCGCAGGCTGTTGGCTCCGGTCATCAATCAGCGGAC----------------AAC-AGCTAACTGTCA--------------ACTGG---------ACACAT----------------------GTC----TCTAATGATG--GACTC--TCACTCCC----------CCCCTCTCT-----------CTCTCTCTCTCTC------------------------------TCTGTCTCTCTCTCTCTCTCTCCATTTGTTATTTGATATGTATGCAGGCTGCGACTGTCATCCTGTAGGTTCCACTGGCAAGACCTGCAATCATCTG | |
| droWil2 | scf2_1100000004963:4212602-4212802 + | TTATTATCGTGATGCCACCAAGCCGCCAAATCATCGTAAAGTCTGCAAACGTAAGGA-----------------------------------------------------------------------------------------------ATACTTCATAACTCATTGCCAAAGTCATCGTCATCGTCATCG--ATTGACC----GC-----------------AATC-ATCAGTGGA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGCTAATTAAGCTCT--TTTTCT-----T-------------------T---------------T-GCATAGGTTGCGATTGCCATCCAGTTGGTTCAACGGGCAAGACCTGCAATCATTTG | |
| dp5 | XL_group1e:4694961-4695317 + | CTACTATCGGGATGCCACCAAGCCGCCAAATCATCGCAAAGTCTGCAAACGTAAGTC-CCCTCCCCCCACCCCCCCATCATATTCCACATAAATAGATAGATTGAATTAGACAGCCGAGACATCGAGGATGAACGTGGGGATCCTGTGCGGAATACG----------------AAGT---GTTCCTTGATATTG--ATAAGCT----GT-----------------CATCAATCAGTGGTCT--------------------GTCAGATGCCAGATACCAGATGCCAGATGGGA--------CA-----CCCATCTTCCCTCGT-AGATGGG-----CCAATAAAGATT--------------------------------------------------CGCCACTCTTCCCGCTAATGAGGATG----TTTT---------------------------------------TATG-TTTTAGGTTGCGACTGCCATCCGGTGGGATCCACGGGCAAGACCTGCAATCATCTG | |
| droPer2 | scaffold_17:1636704-1637046 + | CTACTATCGGGATGCCACCAAGCCGCCAAATCATCGCAAAGTCTGCAAACGTAAGTC-CCCTCCCCCCA------CATCATATTCCACATAAATAGATAGATTGAATTAGACAGTCGAGACATCGAGGATGAACGTGGGGATCCTGGGCGGAATACG----------------AAGT---GTTCCTTGATATTG--ATAAGCT----GT-----------------CATCAATCAGTGGTCT------------GTCAGA-TGCCAGATGCCA--------------GATGGGA--------CA-----CCCATC-TCCCTCGT-AGATGGG-----CCAATAAAGATT--------------------------------------------------CGCCACTCTTCCCGCTAATGAGGATG----TTTT---------------------------------------TATG-TTTTAGGTTGCGACTGCCATCCGGTGGGATCCACGGGCAAGACCTGCAATCATCTG | |
| droAna3 | scaffold_12905:30506-30669 + | TCATCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCAGTGACTCC-------------------GTCAGCCGTCTGGCA-------------GG-CCATTTATCTAGATCTGTCCCCTTGTCCCGTAAGCCGC-----TCTAATGAAGATTGCAT----------------------------------------------CCCTATTTTTGTGGCT--------------------------------------------A---------------A-CCGCAGGATGTGACTGCCACCCCGTCGGCTCGACCGGCAAGACGTGCAACCACCTG | |
| droBip1 | scf7180000391635:37111-37196 - | CTCCCACTAAT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGCTACGAATTTA---------------------------------------TCTC-CCACAGCCTGCGAGTGCCATCCGATCGGCTCATCTGGAAAGATCTGCAACAGCACC | |
| droKik1 | scf7180000302344:1017509-1017668 + | TCATCA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCAGCGGTCTCGTAAGCCGTCTGTCAGACAGACA---------------------GCTGGG-CGGTTAGATAGAT----------------------GGA----CCTAATGAGGACGACAC--TGACAGCCCT--------T--GT----------------------------------------------CT--CTTCC-----TG-------------------C---------------T-CCTTAGGCTGCGACTGCCACCCGGTGGGCTCCACGGGCAAGACGTGCAACCACCTG | |
| droFic1 | scf7180000454073:369810-369932 + | A----ATGAGGATGACA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTTT-----C-TGAAGCTCCCACCTCCATTTTCAT-----------CGCCATTTTCAT--------------C----TCC-ATTTCC-------------------A---------------T-TTGCAGGCTGCGATTGCCATCCCGTGGGCTCGACGGGCAAGACGTGCAACCATTTG | |
| droEle1 | scf7180000490982:513832-513908 + | CCTTCTCCA-CC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCC--CTCG-------------------A---------------T-TCGCAGGTTGTGACTGCCATCCGGTGGGTTCGACGGGCAAGACGTGCAATCACCTG | |
| droRho1 | scf7180000767530:38295-38402 - | TC-TAATGAGGATGACA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T---GCCCGCT-------------------TCTGATTCTCTAAC-----------CCC--------------CC--TCCTCC-----A-------------------A---------------T-TCGCAGGTTGCGACTGCCACCCGGTGGGTTCGACGGGCAAGACGTGCAACCACCTG | |
| droBia1 | scf7180000302126:370971-371032 - | ATACTACCGGGATGCCACCAAGCCGCCGAATCATCGCAAAGTCTGCAAGCGTAAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415169:311988-312076 + | TC-TAATGAGGATGACA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTCCC------------------------------------------------------------------CT--CTTTCC-----C-------------------C---------------C-TCACAGGTTGCGACTGCCATCCGGTGGGTTCGACGGGCAAGACGTGCAATCACCTA | |
| droEug1 | scf7180000409008:1020951-1021016 - | TTCACC-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATGTTCACAGGTTGCGACTGCCATCCAGTGGGTTCGACGGGCAAGACGTGCAATCACCTA | |
| dm3 | chrX:14587028-14587089 - | ATATTATCGGGATGCCACTAAGCCGCCAAATCATCGCAAAGTCTGCAAGCGTGAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:13783066-13783127 - | ATATTATCGGGATGCCACTAAGCCGCCAAATCATCGCAAAGTCTGCAAGCGTGAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_32:39202-39263 - | ATATTATCGGGATGCCACTAAACCGCCAAATCATCGCAAAGTCTGCAAGCGTGAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | X:8816554-8816615 - | ATATTATCGGGATGCCACTAAGCCGCCAAATCATCGCAAAGTCTGCAAGCGTGAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4690:12395544-12395605 + | ATATTATCGGGATGCCACCAAGCCGCCAAATCATCGCAAAGTCTGCAAGCGTGAGTT-CAGCT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:19 AM