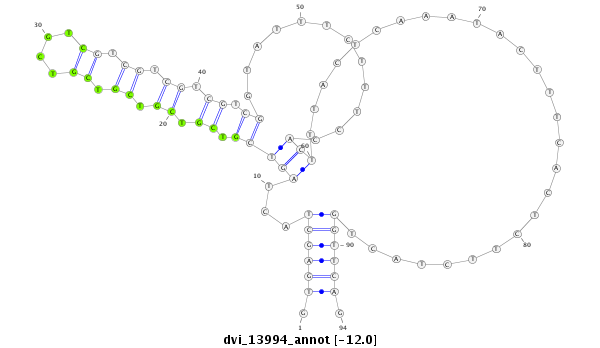
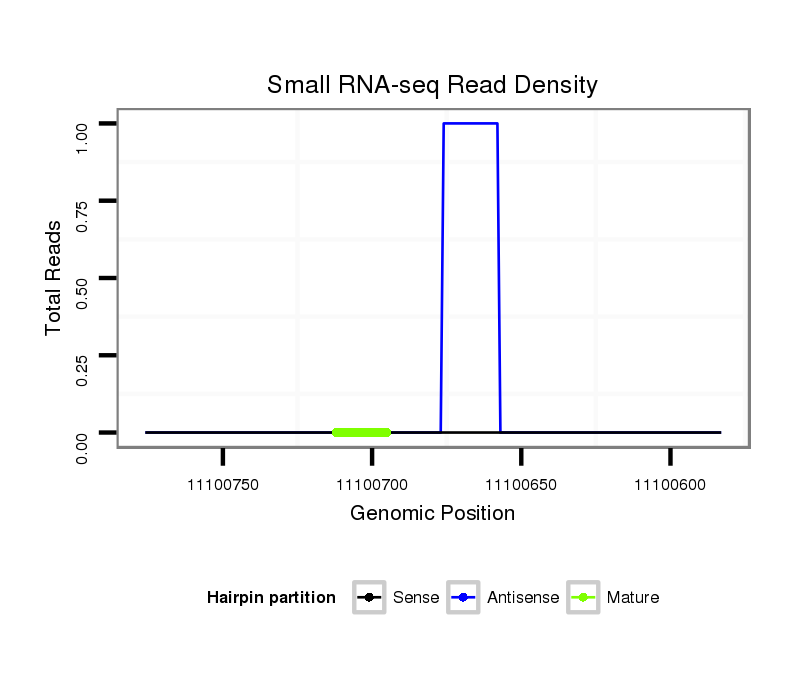
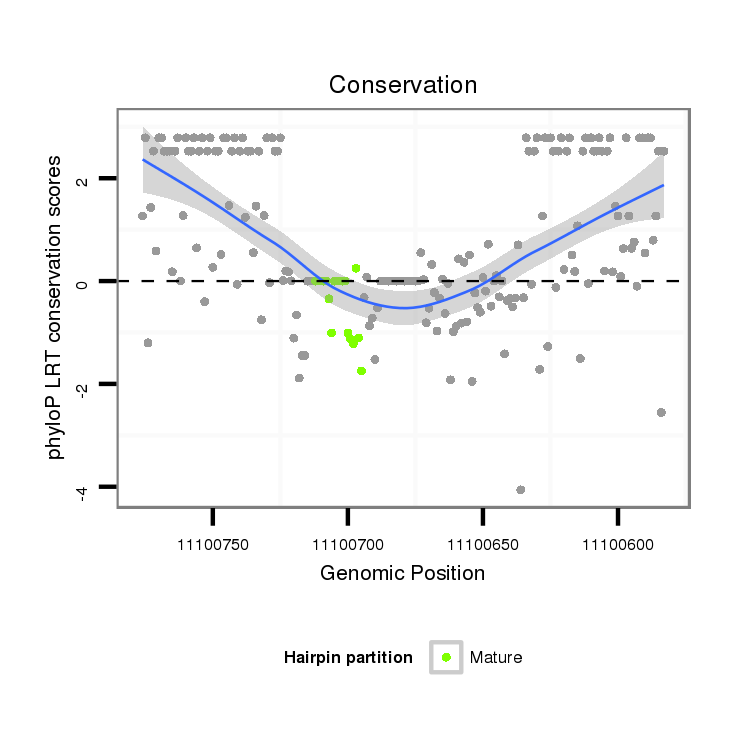
ID:dvi_13994 |
Coordinate:scaffold_12970:11100633-11100726 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.0 | -19.0 |
 |
 |
CDS [Dvir\GJ18620-cds]; exon [dvir_GLEANR_3451:4]; CDS [Dvir\GJ18620-cds]; exon [dvir_GLEANR_3451:5]; intron [Dvir\GJ18620-in]; Antisense to intron [Dvir\GJ19525-in]
| Name | Class | Family | Strand |
| (CGA)n | Simple_repeat | Simple_repeat | + |
| ##################################################----------------------------------------------------------------------------------------------################################################## GTCTCCATGGGCGAACTGGAGGTGCAGGCCAATGTGGTGCGCACACTGAGGTGAGCTACTAGTCGTCGTCGTCGTCGTCGTCGTCGTCGTCGTCGGTATTTCTTTCCACTTTACTCAAATACTTTCACTCTTCTACTGGTTCAGCGTGCTGTCAGAAGATCTGGAATGCTGCGAGACGCTGCATAACTATGCAG **************************************************.((((((...(((((.((.((.((.((....)).)).)).)).))............)))...........................)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060671 9x160_males_carcasses_total |
M061 embryo |
SRR060662 9x160_0-2h_embryos_total |
M047 female body |
V053 head |
V047 embryo |
SRR060686 Argx9_0-2h_embryos_total |
SRR060660 Argentina_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................GAGCTACCGGTCGTCGTCGTC......................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................GACGTCGTTGGTATTTCT........................................................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AGCTACTTGTCGTCGTCGTC......................................................................................................................... | 20 | 1 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................GTTCTACTGGTTCGACGTGCT............................................ | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................GTGCTGTCAGAGGCTCTGG.............................. | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................CGACTAATCGTCGTGGTCG........................................................................................................................ | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................GTGCTGCCAGAAGTTCAGGA............................. | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................CTAATGCTGCGAGACCCTG............. | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .......................................................CGACTAGTCTTCGTTGTCG........................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........GCGAACTGGAGGTGCTGC...................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................................................................GTCGTCGTCGTCGTCGTC................................................................................................................ | 18 | 0 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
CAGAGGTACCCGCTTGACCTCCACGTCCGGTTACACCACGCGTGTGACTCCACTCGATGATCAGCAGCAGCAGCAGCAGCAGCAGCAGCAGCAGCCATAAAGAAAGGTGAAATGAGTTTATGAAAGTGAGAAGATGACCAAGTCGCACGACAGTCTTCTAGACCTTACGACGCTCTGCGACGTATTGATACGTC
**************************************************.((((((...(((((.((.((.((.((....)).)).)).)).))............)))...........................)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060668 160x9_males_carcasses_total |
SRR060674 9x140_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060683 160_testes_total |
SRR060684 140x9_0-2h_embryos_total |
M028 head |
M061 embryo |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ......................................................................................................................................................CAGGCTTCTAGGCCTTACTAC....................... | 21 | 3 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................GTGAAATGAGGTTGTGAAAGT................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................AGAAAGGTGAAATGAGTTT........................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................CCAGTCCCACGGCAGTCTTC.................................... | 20 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................CAGCAGCAGCAGCAGCAGCAG............................................................................................................. | 21 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................CAGCAGCAGCAGCAGCAGCAGCA.............................................................................................................. | 23 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................CAGCAGCAGCAGCAGCAG................................................................................................................... | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .............................................................CAGCAGCAGCAGCAGCAGCAG................................................................................................................ | 21 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................CCATCACGAAAGATGAAAT................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................................................................CAGCAGCAGCAGCAGCAG................................................................................................................ | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................AGCAGCAGCAGCAGCAGCAG............................................................................................................. | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................CAGCAGCAGCAGCAGCAGCAGCA........................................................................................................... | 23 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................AGCAGCAGCAGCAGCAGCAG................................................................................................................ | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ......................................................................CAGCAGCAGCAGCAGCAGCA........................................................................................................ | 20 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:11100583-11100776 - | dvi_13994 | GTCTCCATGGGCGAACTGGAGGTGCAGGCCAATGTGGTGCGCACACTGAGGTGAGCTACTAGTCGTCGTCGTCGTCGTCG----TCGTCGTCGTCGTCGGTATTTCT--------------TT--CCACT-----TT---ACT-CAA---------AT-ACTT------------------------------------T------------CACTCTTCTAC---TGGTTCAGCGTGCTGTCAGAAGATCTGGAATGCTGCGAGACGCTGCATAACTATGCAG |
| droMoj3 | scaffold_6473:10261336-10261500 + | GTCTCCATGGGCGAGCTGGAGGTGCAAGCGAATGTGGTGCGCACGCTGAGGTAAGTACAGA--------CT-----TCGATTG-----GCTT---------------TTCC---------------------------------------------------C-A----------ATTGG---ATTTCTAATATT----------------CCGCTC-TC-TT---CCTGGCAGCGTCTTGTCCGAGGATGTGGAGTGCTGCGATACGCTGCACAACTATGCCG | |
| droGri2 | scaffold_15203:3078419-3078585 - | ATCGCCATGGGTGAACTGGAGGTGCAGGCCAATGTGGTGCGCACCCTGAGGTAAGCGCAGT--------GA-----AGCA----GTGAGCAA---------------ACCA---------------------------------------------------C-A----------AATCA---AATTGTAATCAT----------------CAATTG--T-T-TTCATATTAAGTGTGCTGTCCGAGGATCCGGAATGCTGCGAATCCTTGTTGAACTATGCGG | |
| droWil2 | scf2_1100000004590:4652970-4653133 + | GTCTCCATGGGTGAGCTGGAAGTTCAGGCCAATGTTGTGCGTGCCCTGAGGTGAGTCACTC--------------------------------------------------------------AAAGGAC-----AAACTGAA-GA----------G-----TCCATC-----A-ATTAT---GCTGCAC------TTTA------------------TT-CT---CTGTGCAGTGTTCTATCCGAGGATTCAGATTGCTGTGAGATGCTATTGAAGTATACAG | |
| dp5 | XL_group1e:3375643-3375800 + | GTGTCCATGGGGGAGCTGGAGGTCCAGGCCAATGTTGTGCGGACTCTAAGGTAGCGGTAGT--------CA-----ATGA----CCATGGCT---------------GTCGCAGGACTTAA--TCTCTTC-----CTT------------------------------------------------------------------------------------C---TTTTGCAGCGTGCTCTCTGAGGACTCTGACTGCTGTGAGACCTTGCATAACTATGCCG | |
| droPer2 | scaffold_17:312899-313056 + | GTGTCCATGGGGGAGCTGGAGGTCCAGGCCAATGTTGTGCGGACTCTAAGGTAGCGGTAGT--------CA-----ATGA----CTATGGTT---------------GTCGCAGGACTTAA--TCTCTTC-----CTT------------------------------------------------------------------------------------C---TTTTGCAGCGTGCTCTCTGAGGACTCTGACTGCTGTGAGACCTTGCATAACTATGCCG | |
| droAna3 | scaffold_13248:2596573-2596731 - | GTATCCATGGGCGAGCTGGAGGTCCAGGCCAATGTGGTGCGCACTTTGAGGTGAGTTCTTG--------------------------------------------------------------------------------------GGATGAAGTT-----C--TTCGCCGGAA----ACTTGCTC------ATCTCTC------------------TT-CT---CCCTCCAGTGTCCTTTCGGAGGACACAGATTGCTGTGAAACCCTGCACAACTATGCGG | |
| droBip1 | scf7180000396429:380688-380856 + | GTTTCCATGGGCGAGCTGGAGGTCCAGGCCAATGTAGTGCGCACTTTGAGGTGAGTTTTTA------------------------------------------------------------TT---CATGCAAATCT---TTC-GAA---------GG-ATCTCC----------ACTAACTTGCTGGCC------TTTC------------------AA-CT---CCTTTTAGTGTCCTCTCGGAAGATACAGATTGCTGCGAAACCCTGCACAACTATGCGG | |
| droKik1 | scf7180000302393:73353-73504 + | GTGTCGATGGGCGAGCTGGAGGTCCAGGCGAACGTAGTTCGCACTCTGAGGTGGGTTTTGA--------TT-----TCTATTTAGTTTCGTT---------------GTTG-------------------------------------------------------------------CA---ATCTT------------------------------TA-AT---CCTTTTAGCGTGCTTTCCGAAGACTCCGAGTGCTGTGAAACGCTGCACAATTACGCTG | |
| droFic1 | scf7180000453882:1014886-1015043 - | GTCTCCATGGGCGAACTGGAGGTCCAGGCCAATGTGGTGCGCACTCTCAGGTGGGTGTGGC------------------------------------------------------------TT---TATG-----CCTCTTTA-AGG---------AG-CTCT-A----------ATTCG---GTTTGA--------AA----------------------TC---CCTTCCAGCGTTCTTTCCGAGGACTCCGATTGCTGTGAAACGCTGCACAACTACGCCG | |
| droEle1 | scf7180000491193:686594-686753 + | GTTTCCATGGGCGAGCTGGAAGTGCAGGCCAATGTGGTGCGCACTCTGAGGTAAATCGAAC------------------------------------------------------------TA---CATC-----CTCCATTC-GAA-------G-AG-ATAA-A----------ATTTA---GCCTTT--------AA--------------------T-CC---CTTTGCAGCGTGCTCTCCGAGGACTCGGATTGCTGTGAAACGCTGCACAACTATGCGG | |
| droRho1 | scf7180000779986:34436-34594 - | GTTTCCATGGGCGAACTGGAGGTGCAGGCCAATGTGGTGCGCACCCTGAGGTGGGTTTAAC------------------------------------------------------------AA---TACC-----CTTTCTTT-AGA---------AG-CTGT-G----------ATTTA---GCTGCT--------AA--------------------T-TC---CTTTCCAGCGTTCTCTCCGAGGACTCCGATTGCTGTGAAACGCTGCACAACTATGCTG | |
| droBia1 | scf7180000301345:792137-792297 + | GTTTCCATGGGCGAACTGGAGGTGCAGGCCAATGTGGTGCGCACGCTGAGGTGGGTTGTGC------------------------------------------------------------TC---TATATATT-GCTCTTTGATG----------AGTTTCT-G----------ACCAG---GCTTT-------------------------------G-CC---CCTCGCAGCGTTCTCTCCGAGGACTCCGATTGCTGCGAAACGCTGCACAACTATGCCG | |
| droTak1 | scf7180000415909:17533-17691 - | GTCTCCATGGGCGAACTGGAGGTGCAGGCCAATGTGGTGCGCACTCTGAGGTGGCTATATA--------------------------------------------------------------------------------------AGTTAAGGAA-----T--TCTTTCTGGC----TCTCATTA------ATTCATT------------------CT-CT---ATCCATAGCGTTCTTTCCGAGGACTCCGATTGCTGCGAAACGCTGCACAACTATGCGG | |
| droEug1 | scf7180000409631:183708-183863 - | GTTTCCATGGGCGAACTGGAGGTTCAAGCCAATGTGGTGCGCACTCTAAGGTGGGATTAAC------------------------------------------------------------TT---TAAC-----TTTCTATC--------------------------------------------------AT--TTCTTACTCAAAATCGA----TT-CC---CTTTCTAGTGTTCTTTCGGAGGACTCTGATTGCTGTGAAACGCTGCACAACTATGCTG | |
| dm3 | chrX:11052103-11052266 - | GTATCCATGGGCGAACTGGAGGTCCAAGCCAATGTGGTGCGCACACTCAGGTGGGTTTCCA--------------------------------------------------------------TCTCATC-----CTTTTAGC-GAA---------TG-ATTT-A----------ACTGA---GTTGTGA------TTA--------------------T-T-ACAATTTTTAGTGTGCTCTCGGAGGACTCTGATTGCTGTGAAACGCTGCACAACTATGCTG | |
| droSim2 | x:10501523-10501686 - | dsi_26714 | GTATCCATGGGCGAACTGGAGGTCCAGGCCAATGTGGTGCGCACACTCAGGTGGGTTTCCA--------------------------------------------------------------TCTCATC-----CTTCTGGG-GCA---------AG-ATCT-A----------ACTGA---GCTGTGA------TTA--------------------T-T-CCAATTTTTAGTGTGCTCTCGGAGGACTCTGATTGCTGTGAAACGCTGCACAACTATGCTG |
| droSec2 | scaffold_15:1756022-1756185 - | GTATCCATGGGCGAACTGGAGGTCCAGGCCAATGTGGTGCGCACACTCAGGTGGGTTTCCA--------------------------------------------------------------TCTCATT-----CTTCTGGG-GCA---------AG-ATCT-A----------ACTGA---GCTGTGA------TTA--------------------T-T-CCAATTTTTAGTGTGCTCTCGGAGGACTCTGATTGCTGTGAAACGCTGCACAACTATGCTG | |
| droYak3 | X:11074767-11074927 + | GTATCAATGGGCGAAATGGAGGTCCAGGCCAATGTGGTGCGCACTCTCAGGTGGGTTTTAC------------------------------------------------------------TT--CTATC-----CT---ATC-GAA---------AG-ATTT-A----------ACAGA---GCTATGA------TTC--------------------T-T-CCAATTTCTAGTGTGCTTTCGGAGGACTCGGATTGCTGTGAAACGCTGCACAACTATGCTG | |
| droEre2 | scaffold_4690:4078551-4078713 + | GTATCCATGGGCGAACTGGAGGTCCAGGCCAATGTGGTGCGCACTCTCAGGTGGGTTTCAC------------------------------------------------------------TT---TATC-----CTTCTATC-GAA---------CG-ATTT-A----------ACTGA---GCTTCGA------TAA--------------------T-C-GCCATTTTTAGTGTACTTTCGGAGGACTCCGATTGCTGTGAAACGCTGCACAACTATGCTG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/17/2015 at 04:16 AM