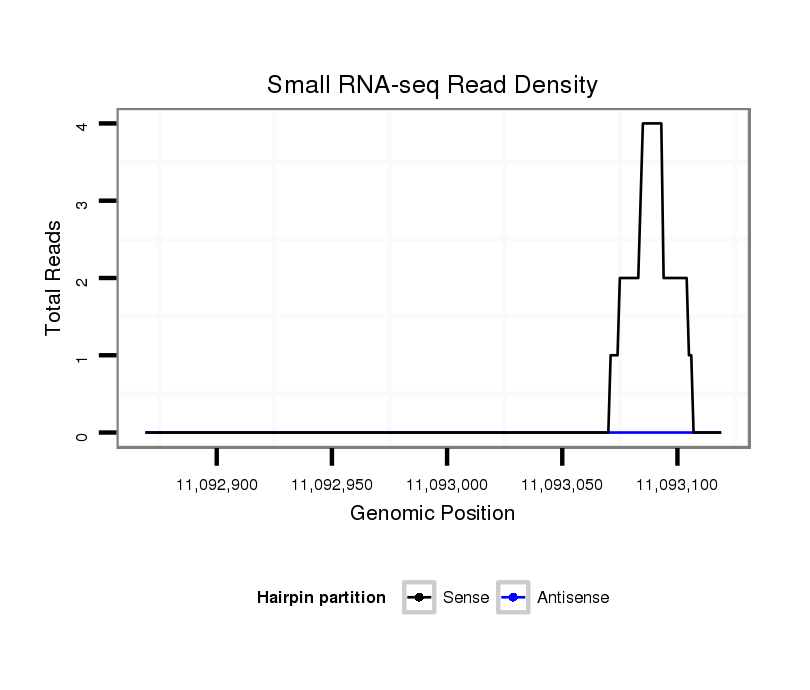
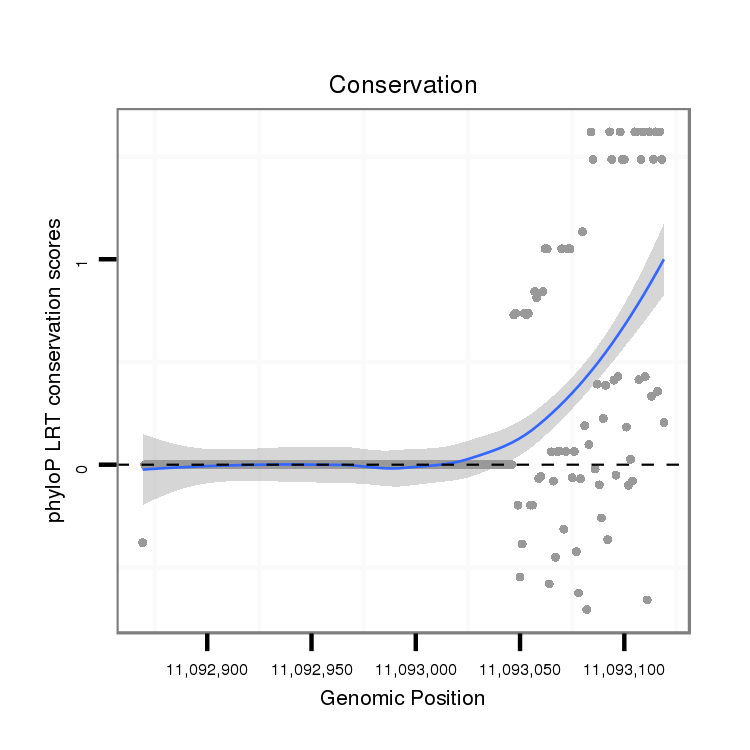
ID:dvi_13983 |
Coordinate:scaffold_12970:11092919-11093069 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_4412:4]; CDS [Dvir\GJ19523-cds]; intron [Dvir\GJ19523-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CGGTTGTGTGGGCGAATATTTTATATACTATGTCCTATCCTCTAAATTGATTCACATAAAATTTAGTGAGAATTGCTTCACTGCCAATGCTGGAAGTGGATTGAGAATTGAAAGTAAAATACGAGGCCCACGCAAGCCGAGTTCTATTAACATAGCGTAGTATAGGTACGTCTATTGACTTCGTCTTTTCTGTTTTTACAGACAATTTCCTGACCTGCCAGAGCTGTCATGGGCATTATCATCTGGATTGT **************************************************(((((..........)))))......((((((.(((....))).)))))).(((((((.(((((......((..(((........)))..)).........((((((((.......)))).)))).(((...)))))))).)))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060660 Argentina_ovaries_total |
SRR060670 9_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060673 9_ovaries_total |
V047 embryo |
SRR060681 Argx9_testes_total |
M027 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................................ATGGGCTTTATCATCTGGATT.. | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................TTCCTGACCTGCCAGAGCT.......................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................TGCCAGAGCTGTCATGGGCAT............... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................CAATTTCCTGACCTGCCAGAGCT.......................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GCCAGAGCTGTCATGGGCATTA............. | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................................ACCTGCCAGAGCTGTCATGGGCCTT.............. | 25 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................CGGAGCAGTCATGGGCATTC............. | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................ATCCTGTCCTGCCACAGCTGTC....................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................AGGATTGCTTCACCGCCA..................................................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................GCTTGAAGTGGATTGACA................................................................................................................................................. | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GCCAACACACCCGCTTATAAAATATATGATACAGGATAGGAGATTTAACTAAGTGTATTTTAAATCACTCTTAACGAAGTGACGGTTACGACCTTCACCTAACTCTTAACTTTCATTTTATGCTCCGGGTGCGTTCGGCTCAAGATAATTGTATCGCATCATATCCATGCAGATAACTGAAGCAGAAAAGACAAAAATGTCTGTTAAAGGACTGGACGGTCTCGACAGTACCCGTAATAGTAGACCTAACA
**************************************************(((((..........)))))......((((((.(((....))).)))))).(((((((.(((((......((..(((........)))..)).........((((((((.......)))).)))).(((...)))))))).)))))))...************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060666 160_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................TGCGTGCGGCCCAAGATAGT...................................................................................................... | 20 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| ............................................................................AAGTGAGGGTTAGGGCCTTC........................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................................................................................AGTACCCGAAGTAGTAGA....... | 18 | 2 | 15 | 0.07 | 1 | 0 | 0 | 1 | 0 |
| ...................................................................................................................TGGTATGCTTCGGGTGCGT..................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:11092869-11093119 + | dvi_13983 | CGGTTGTGTGGGCGAATATTTTATATACTATGTCCTATCCTCTAAATTGATTCACATAAAATTTAGTGAGAATTGCTTCACTGCCAATGCTGGAAGTGGATTGAGAATTGAAAGTAAAATACGAGGCCCACGCAAGCCGAGTTCTATTAACATAGCGTAGTATAGGTACGTCTATTGACTTCGTCTTT-TCTGTTTTTACAGACAATTTCCTGACCTGCCAGAGCTGTCATGGGCATTATCA-TCTGGATTGT |
| droMoj3 | scaffold_6473:10268648-10268706 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTGTGTAGAAAATCTGCAGAGCTGTCTGAGTTGCCATGGGGTTTATCA-TATGGATTGT | |
| droGri2 | scaffold_15203:3071301-3071342 + | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGATGCCCGTGCTGCCATGGCCAATATCAATCTGGATTGC | |
| droAna3 | scaffold_13248:2590619-2590682 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCATTTCAGAGAAAGATTCGATGGCCTGCCACTTTTGCGGTGGCTTCTATCA-CTTGGATTGC | |
| droBip1 | scf7180000396429:387826-387900 - | C---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCGTTCTCCTTCCATTTCAGAGAAAGATTCGGTGGCCTGTAAGTTTTGCGGTGGCTTCTATCA-CTTGGATTGC | |
| droTak1 | scf7180000415909:11276-11312 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCCATTTCTGCAGTGGCCTTTACCA-CCTCGACTGC | |
| droEug1 | scf7180000409631:177031-177067 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGCCATTTTTGCAGTGGCCTTTATCA-CATGGACTGC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/17/2015 at 04:07 AM