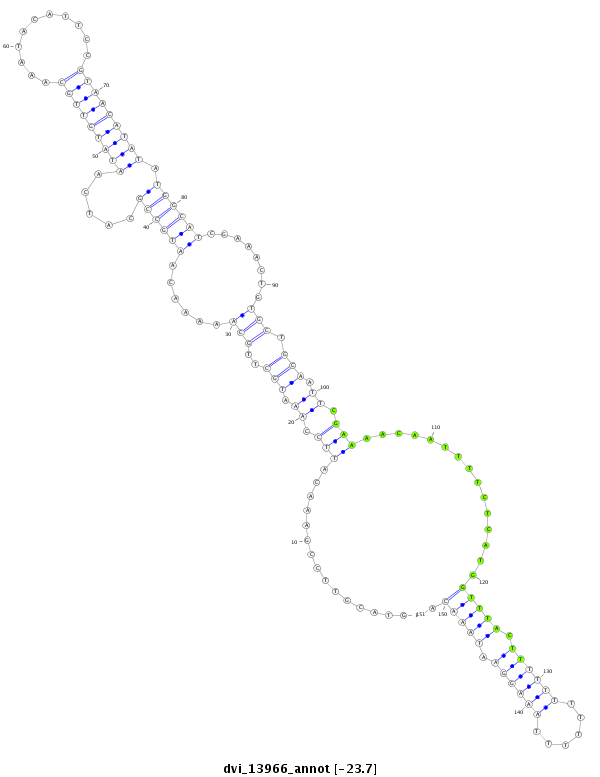


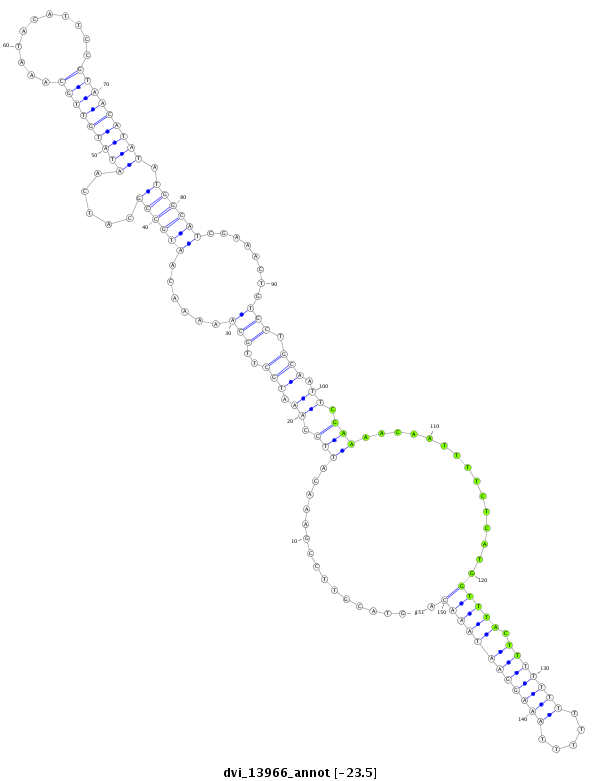
ID:dvi_13966 |
Coordinate:scaffold_12970:11003469-11003619 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -23.5 | -23.1 | -22.9 |
 |
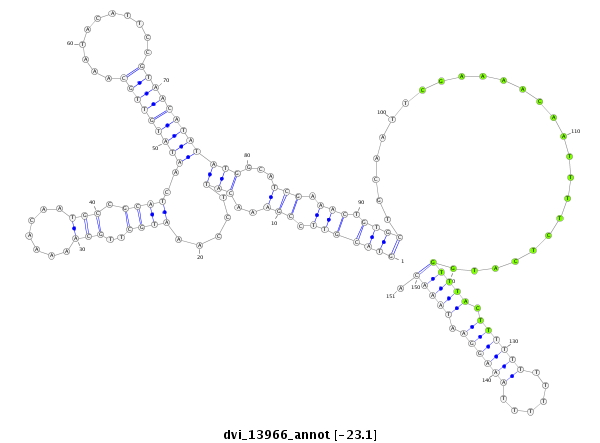 |
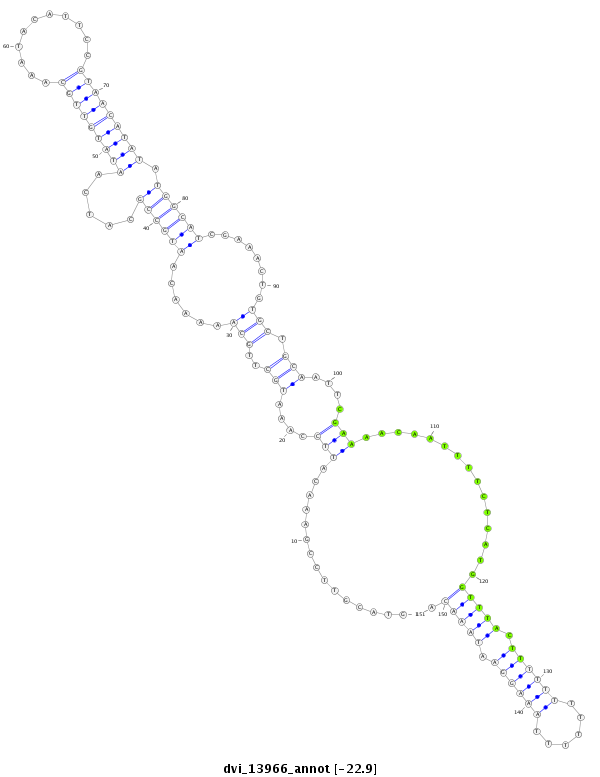 |
exon [dvir_GLEANR_3464:1]; CDS [Dvir\GJ18634-cds]; intron [Dvir\GJ18634-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- GCTGCTGAGCAGCGATGATATTACCAGCAGCGATGATGAGCGCGAGCCAGGTACGTTCCGAAACATTCCAAATGCTTGCAAAAACAATGCCGCATCAATATGTTGCAAATACATTCCGTAACATATATGGCATCGAAACTGTGCTGCAATTCGAAAACAATTTTCTCATGGTTTACTTTTTTTTTTTTAAAGGAATAAACACAAGTCTATTTCAAACTAAGCAACAATATTTTTCTCAATATAAGTATAAT **************************************************...............(((.((.(((..(((......((((((.....(((((((((...........))))))))).))))))........))).))).)).)))...............(((((.((((((......)))))).))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
M061 embryo |
SRR060666 160_males_carcasses_total |
SRR060681 Argx9_testes_total |
SRR060683 160_testes_total |
V047 embryo |
M047 female body |
V116 male body |
SRR060679 140x9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................AGCGATGATGAGCGCGAG............................................................................................................................................................................................................. | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....TGAGCAGCGATGATATTACCAGC............................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................TATTACCAGCAGCGATGATGAGCT................................................................................................................................................................................................................. | 24 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....CGGGTCAGCGATGATATTACC.................................................................................................................................................................................................................................. | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................ATATTACCAGCAGCGATGATG..................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................CGAAAACAATTTTCTCATGGTTTACTT......................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................AGTTTCACATGGTTAACTTTTT...................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....CGGGGCAGCGTTGATATTAC................................................................................................................................................................................................................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................TGATGAGGGCGAACCAGGCA...................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................GCTGGAATTCGAAAACGAT.......................................................................................... | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ....................................................................................................................................................................................................................................ATTTTTATCAATATAAGTC.... | 19 | 2 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 |
| ...................CTGACCAGCAGCGATGAT...................................................................................................................................................................................................................... | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CGACGACTCGTCGCTACTATAATGGTCGTCGCTACTACTCGCGCTCGGTCCATGCAAGGCTTTGTAAGGTTTACGAACGTTTTTGTTACGGCGTAGTTATACAACGTTTATGTAAGGCATTGTATATACCGTAGCTTTGACACGACGTTAAGCTTTTGTTAAAAGAGTACCAAATGAAAAAAAAAAAATTTCCTTATTTGTGTTCAGATAAAGTTTGATTCGTTGTTATAAAAAGAGTTATATTCATATTA
**************************************************...............(((.((.(((..(((......((((((.....(((((((((...........))))))))).))))))........))).))).)).)))...............(((((.((((((......)))))).))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V116 male body |
SRR060675 140x9_ovaries_total |
SRR060660 Argentina_ovaries_total |
SRR060688 160_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR060657 140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................................................TTAGTTGTGATCAGAAAAAGT..................................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................AAAAATTTGCGTATTTGTGT................................................ | 20 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................TCGGTCAATGGAAGCCTTT............................................................................................................................................................................................ | 19 | 3 | 14 | 0.14 | 2 | 0 | 0 | 1 | 1 | 0 | 0 |
| ...............................................................................................................................................................................................................AAAAAGTTTGAGTCGTTGT......................... | 19 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............................................TCGGTCAATGGAAGCCTT............................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:11003419-11003669 - | dvi_13966 | GCTGCTGAGCAGCGATGATAT---TACCAGC---------------AGCGATGATGAGCGCGAGCCAGGTACGTTCCGAAACA----------TTCCAAATGCTTGCAAA-AACAATGCC--------------------GCATCAATATGTT--GCAAATACAT--TCCGTAACATATATGGCATCGAAACTGTGCTGCAATTCGAAAACAATTTTCTCATGGTTTACTTTTTTTTTTTTAAAGGAATAAACACAAGTCTATTTCAAACTAAGCAACAATATTTTTCTCAATATAAGTATAAT |
| droMoj3 | scaffold_6473:10343259-10343388 + | GCTGCTGAATAGCGATGATGATAATGCCAGC---------------AGCGATGATGTGCGGAATGCAGGTATGTTCCAAAACATTCCAAAACATTCCAATTGCTTGCAGC-GAGCGCGCA--------------------GCATCCAGACGAG--AGAATCGCAT--TCC-------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15203:2995679-2995855 - | GCTGCTGGACAGTGACAATAATGCTGCCAACGATGTCAACGACGAGAGCGATGATTC------GGCAGGTACGTTTAA----G----------TCCCAAGTGATGGCAAGCGACAATGCCGAAAGCTCAAGGGATCATCTGCATCCAAATGGGTGGCAAATATATATTC----ATATGT---ATATTGAAAATGTGTTTCCACT---------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/17/2015 at 03:51 AM