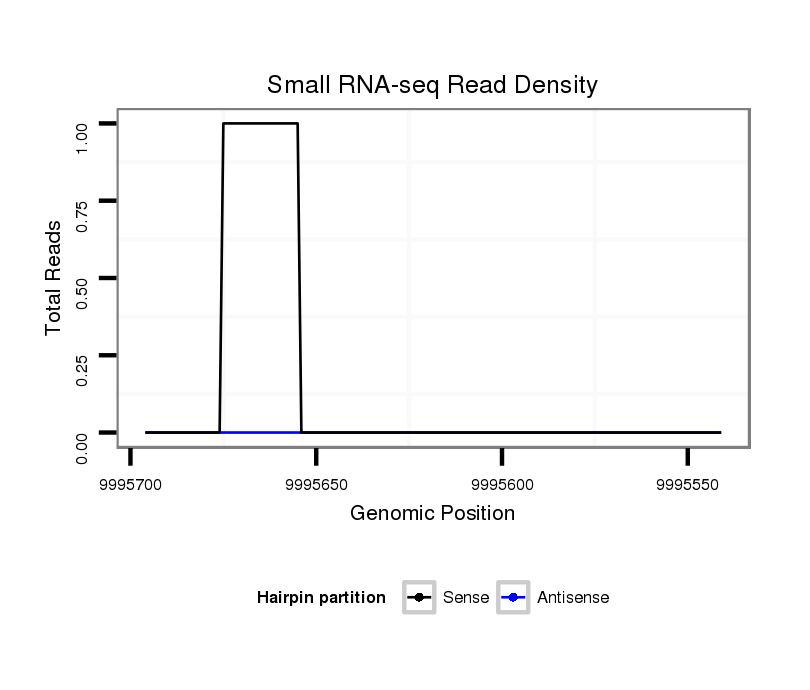
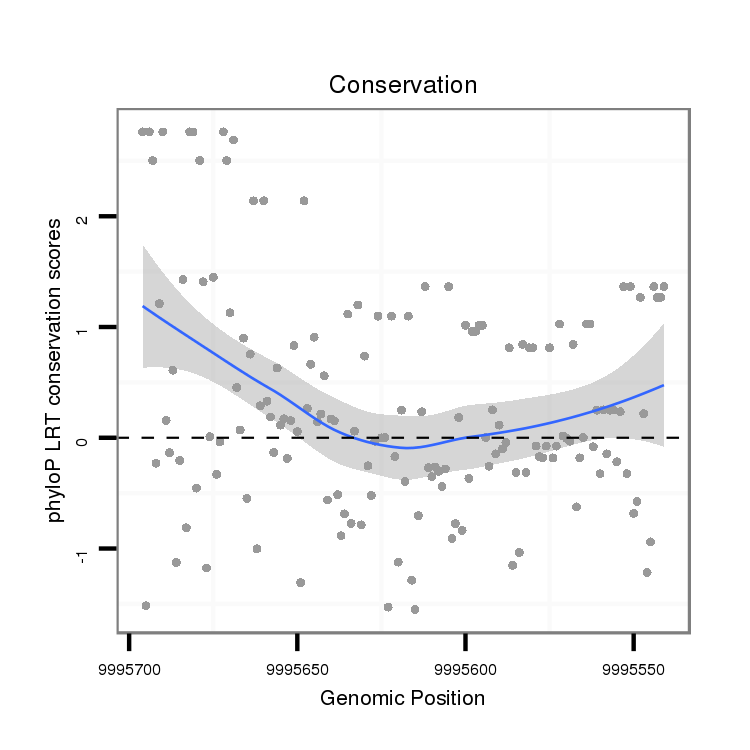
ID:dvi_13855 |
Coordinate:scaffold_12970:9995591-9995646 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_3494:2]; CDS [Dvir\GJ18667-cds]; CDS [Dvir\GJ18667-cds]; exon [dvir_GLEANR_3494:3]; intron [Dvir\GJ18667-in]; Antisense to intron [Dvir\GJ19479-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------################################################## AGAGTCAAGCGAAAATTGCTGAACTGGAAGGCTACGTAGCACACTTGCAGGTAAGAATAACGCTTCTTGCTATCAGGGATAGGAATACTCGTGGATTGCCTTTCAGGATGAGAAGCCTCTAGAAGCGCTCTTCGCCAAAGATCTGAAGCGTATGGA **************************************************...................((...(((..((.((....)).)).....)))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060678 9x140_testes_total |
SRR060689 160x9_testes_total |
V047 embryo |
SRR060662 9x160_0-2h_embryos_total |
SRR060674 9x140_ovaries_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060657 140_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........AAAATTGCTGAACTGGACGG............................................................................................................................. | 20 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................AACTGGAAGGCTACGTAGCAC.................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................GCGTAGCACACTGGCAGG......................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................CACTTCTTTCGATCAGGGA............................................................................. | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............AAATTGTTGAAATGGAAGG............................................................................................................................. | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................CGGAGCTGGAAGGCTAAGT....................................................................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................GGTAAGAAGAACGGGTCTT........................................................................................ | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................CCCGCTCTTCGTCAAAGAT.............. | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ...........AAAATTCCTGAACCGGGA............................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TCTCAGTTCGCTTTTAACGACTTGACCTTCCGATGCATCGTGTGAACGTCCATTCTTATTGCGAAGAACGATAGTCCCTATCCTTATGAGCACCTAACGGAAAGTCCTACTCTTCGGAGATCTTCGCGAGAAGCGGTTTCTAGACTTCGCATACCT
**************************************************...................((...(((..((.((....)).)).....)))...))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060670 9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
M061 embryo |
SRR060686 Argx9_0-2h_embryos_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|
| ...............................GATGCTCGGTGTGAACGTCCAT....................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................TCCTTATGAGCCCGTATCG......................................................... | 19 | 3 | 7 | 0.43 | 3 | 0 | 2 | 0 | 1 | 0 |
| ...................................................................................................................................AGCGGTTTCTAGGGTTCGCTT.... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 |
| ...........................................................................CCCGATCCTTATGTGCAAC.............................................................. | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:9995541-9995696 - | dvi_13855 | AGAGTCAAGCGAAAATTGCTGAACTGGAAGGCTACGTAGCACACTTGCAGGTAAGAATAACGCTTCTTGCTATCAGGGATAGGAATACTCGTGGAT---------------------------TGCCTTTCAGG------------ATGAGAAGCCTCTAGAAGCGCTCTTCGCCAAAGATCTGAAGCGTATGGA |
| droMoj3 | scaffold_6496:13670155-13670219 - | AAAGCCAAGCCCTCATTGAACAAATGCAACAGTACATTCAACAGCTGGAGGTAAGCATCATGATT---------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15112:3924125-3924177 + | AGAGCCATGCGATGATTGAGCAAATGCAGCAATATATACACCAGCTGGAGGTA---------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004382:918265-918370 + | AGAGTCAGGCTTTGATTGAGCAACTGCAGCAACACATACGCGACCACCAACAATC----------------------A--TCGGACACCAGTGCATCCCCAAT----------------------------GACCTG-------------------------------------TCGGAGATTTAAACGCATGGA | |
| dp5 | 3:8171685-8171710 + | AAAGCAATGCGTTGATCGATGAGCTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_4:2049751-2049924 + | AAAGCAATGCGTTGATCGATGAGCTGCAGCAGCATCTGCGCCACTACGAGGTGAGTATCTTGTATG--TCT-TGAGCAGTCATGACCTCTATTCCCTACTTGTGCATTTTAGGAAACTGGAGCTGCCTTTGAAGGAGAGTCCAGAGAA---------------GCAC---TCGATGAAAAGCTCAAACACTTGGA | |
| droAna3 | scaffold_13266:16634944-16635112 - | AAAGTCAGGCCATGATTGATCATCTGCAGGAACAAATACGCGGCTATCAGGTGAGATAAA--------CAT-AAACTATTAAAGATTAAAGTTCATTCCTTATTCCTTCTGA-----------------ACAGGCTGCATCTAGAGCTGGCAACCCAAGTGCTGTGATTGACGCTGAGAAGCTCAGGCGGCTGGA | |
| droBip1 | scf7180000396384:744018-744184 - | AGAGTCAGACCATGATCGAGCATCTGCAGCAACAAATACGCGGCTATGAGGTGGGATTACAGCATATACAT-ACACCATTTCGGACTAAAATGTAA---------------------------TCCCTTACAGGCTGCGGCCAGAGCTGTCCACCCAAGTGCTGCGATCGACGCTGAGAAGCTCATACGTTTGGA | |
| droKik1 | scf7180000302277:506054-506108 + | ACAGTCAGACCATAATAGATGGACTGCAGGAACAGATACGCGCATATGAAGTAAG-------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454039:140588-140617 - | AAAGCCAGATGTTAATCGAAGAGTTGCAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491282:562766-562791 - | ACAGCCAGGCGATAATTGATGATCTG------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000759836:33163-33192 + | ACAGCCAGGCGATAATTGATGACTTGCAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302291:842501-842530 + | ACAGCCAGACAATTATCGATGAGTTGCAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000415379:502704-502733 - | ACAGCCAGAGAATAATCGATGAGTTGCAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409758:395597-395626 + | AAAGTCAGACTATTATTGATGAGTTGCAGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | 2r:9995272-9995326 - | AAAGCCAGGCAATAATCGACGAGTTGCAAGAGCAAATAAACGCCTATCAGGTGAG-------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_1:6851394-6851448 - | AAAGCCAGGCAATAATCGACGAGTTGCAAGAGCAAATAAACGCCTATCAGGTGAG-------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2R:7626539-7626593 + | ACAGCCAGGCAATGATCGACGAGTTGCAAGAGCAGATACACGCCTATCAGGTGAG-------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4845:16485029-16485058 + | ACAGCCAGGCATTAATCGATGAGTTGCAAG--------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:42 PM