
ID:dvi_13854 |
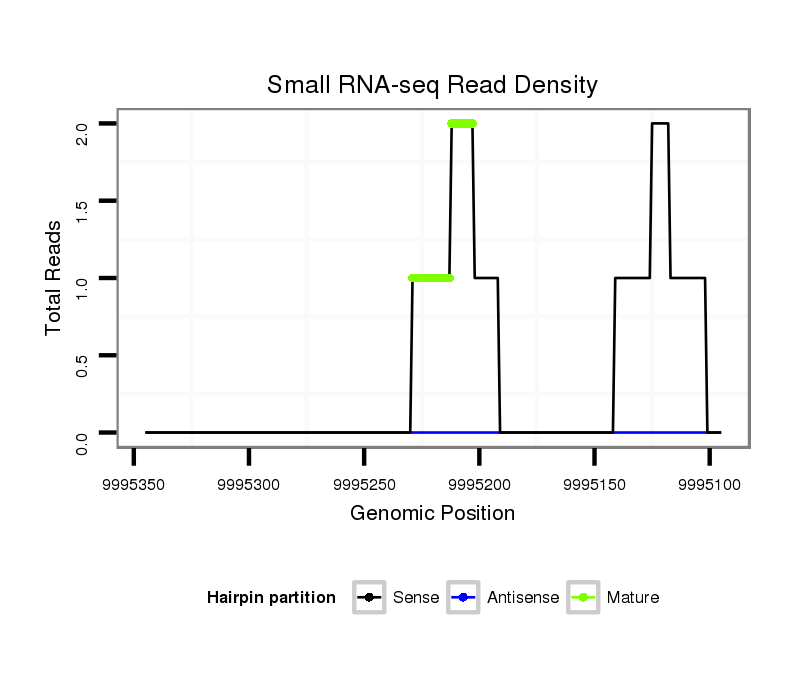
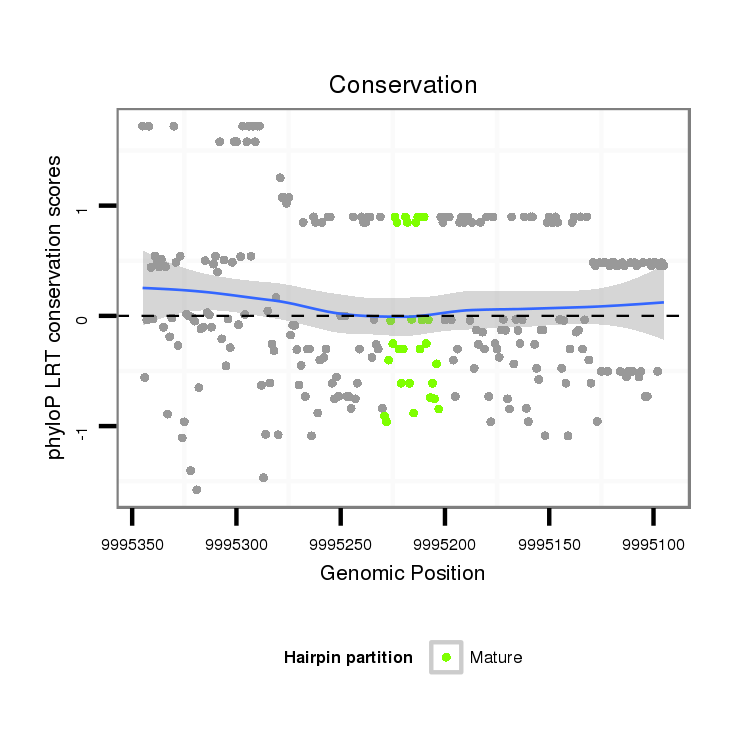
Coordinate:scaffold_12970:9995145-9995295 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
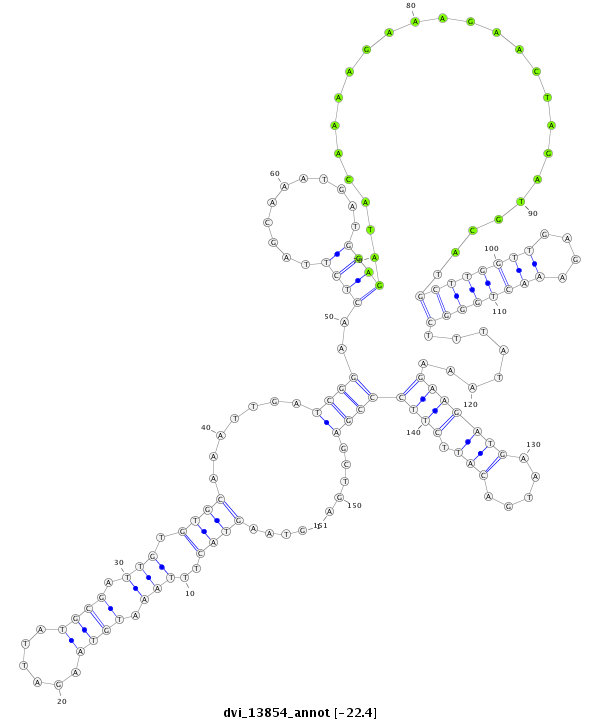
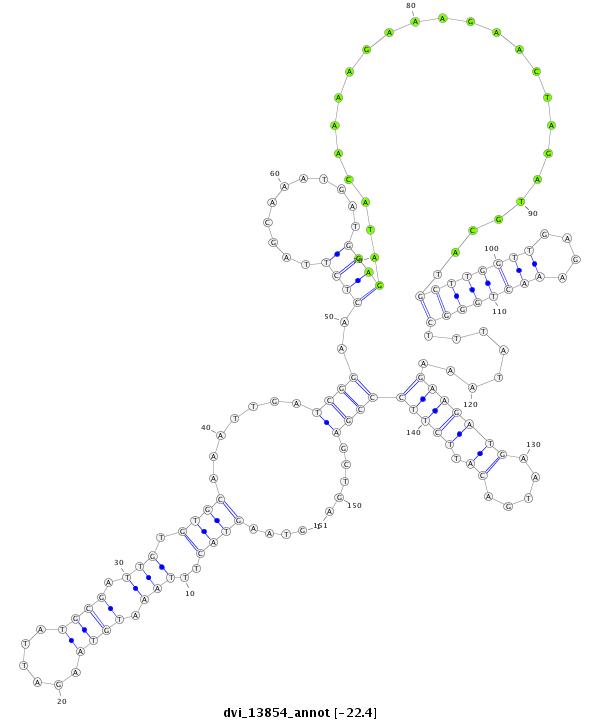
| -22.7 | -22.4 | -22.4 |
 |
 |
 |
CDS [Dvir\GJ18667-cds]; exon [dvir_GLEANR_3494:4]; intron [Dvir\GJ18667-in]; Antisense to intron [Dvir\GJ19479-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ACATGATGAACCTACTCCAACAGCATTACAGGTTCAGCCTCATGGCGAATGTAAGTACTTTAAATGTAAGATTATGCGATTGTGTGCAAATTGATCGGAACTCTTAGCAAATGATGGAGATACAAAAGAAAGAACTAGATGCATGCTTGGTTGAGAAACTGGGCTTTATAAAGAAGATGAATGACATTCTTCCCGAGCTGAATTGTCAGAATGAGTTGCTAAAAGAAGCGACAATAACTGTGCAGAACCTC **************************************************...(((.((((...((((.(((((((((......))))...)))))....((((...........)))).))))....)))).))).....((.((((((..(((((..((...(((((.......)))))...))))))).)))))))).************************************************** |
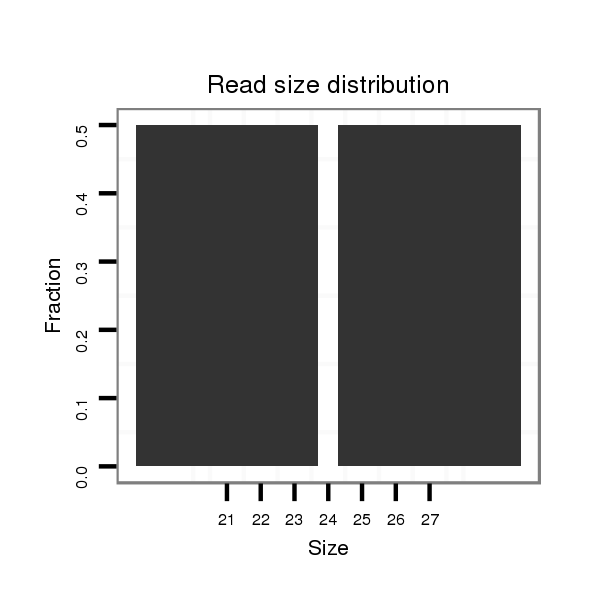
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060688 160_ovaries_total |
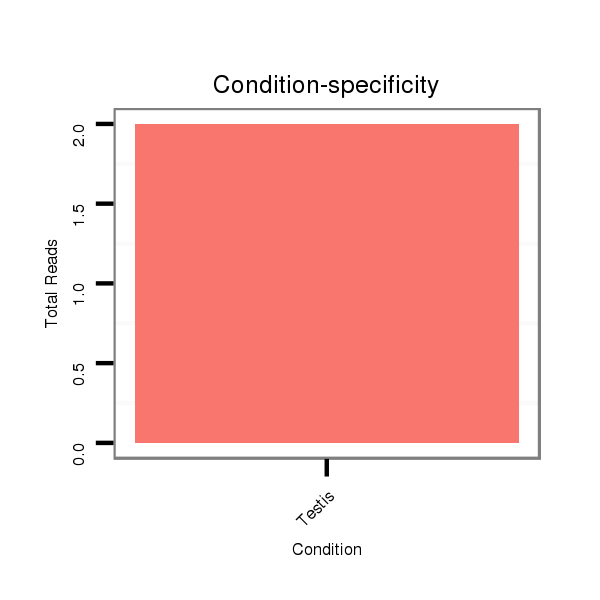
SRR060679 140x9_testes_total |
SRR060678 9x140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060660 Argentina_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060672 9x160_females_carcasses_total |
SRR060673 9_ovaries_total |
SRR060658 140_ovaries_total |
SRR060669 160x9_females_carcasses_total |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
SRR060668 160x9_males_carcasses_total |
SRR060671 9x160_males_carcasses_total |
V116 male body |
SRR060655 9x160_testes_total |
SRR060656 9x160_ovaries_total |
SRR060659 Argentina_testes_total |
SRR060667 160_females_carcasses_total |
SRR060670 9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................................................................................ATTCTCAGAATGACTTGGTAA............................. | 21 | 3 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................AAAAGAAGCGACAATAACTGTGCA....... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GAGATACAAAAGAAAGAACTAGATGCA............................................................................................................ | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................GTCAGAATGAGTTGCTAAAAGAAG....................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................ACTAGATGCATGCTTGGTTGA................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................ATAAGTTGCTAAACGAAGCGGC................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................ATTACAGGTTCAGCCTCATGCCGA........................................................................................................................................................................................................... | 24 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................ATTCTCAGAATGACTTGGTAAA............................ | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................AAAGAAGCGAAAATACCTGA.......... | 20 | 3 | 20 | 0.60 | 12 | 0 | 1 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 2 | 2 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...........................................................................................................................................................................AGAAGAGGAATCACATTCTGC........................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................ATTCTCAGAATGACTTGGT............................... | 19 | 3 | 20 | 0.35 | 7 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................AGAACGAGTTGCATGCTTG...................................................................................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................ATTCTCAGAATGACTTGGTA.............................. | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................GTTCGGATCTCTTAGCAA............................................................................................................................................. | 18 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TCTCAGAATGACTTGGTAAA............................ | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................TTCTCAGAATGACTTGGTAA............................. | 20 | 3 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................AGCTTAATGGTCAGAATG...................................... | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................AGCAGGCTTGGTTGGGAAAC............................................................................................ | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................................AAAGAAGCGAAAATACCT............ | 18 | 2 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................TTGGGTGAGCAACTGGGCGT..................................................................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TCTCAGAATGACTTGGTAA............................. | 19 | 3 | 14 | 0.07 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................GATTGTGTGCAATGTGATAG.......................................................................................................................................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................TAGCTTAATGGTCAGAATG...................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....GATGAACTTACTGCAACAGT................................................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ............GACTCCAACACCATTAGA............................................................................................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TGTACTACTTGGATGAGGTTGTCGTAATGTCCAAGTCGGAGTACCGCTTACATTCATGAAATTTACATTCTAATACGCTAACACACGTTTAACTAGCCTTGAGAATCGTTTACTACCTCTATGTTTTCTTTCTTGATCTACGTACGAACCAACTCTTTGACCCGAAATATTTCTTCTACTTACTGTAAGAAGGGCTCGACTTAACAGTCTTACTCAACGATTTTCTTCGCTGTTATTGACACGTCTTGGAG
**************************************************...(((.((((...((((.(((((((((......))))...)))))....((((...........)))).))))....)))).))).....((.((((((..(((((..((...(((((.......)))))...))))))).)))))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................CGCCTTCACAGTCTCACTCA................................... | 20 | 3 | 5 | 0.20 | 1 | 1 | 0 |
| ...............................................................................................................................................TCGAACCGACTCTCTGACC......................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:9995095-9995345 - | dvi_13854 | ACATGATGAACCTACTCCAACAGCATTACAGGTTCAGCCTCATGGCGAATGTAAGTACTTTAAA-------------TGTAAGATTA---TGCGATTGTGTGCAAATTGATCGGAACTCTTAGCAAATGATGGAGATACAAAAGAAAGAACTAGATGCATGCTTGGTTGAGAAACTGGGCTTTATAAAGAAGATGAATGACATTCTTCCCGAGCTGAATTGTCAGAATGAGTTGCTAAAAGAAGCGACAATAACTGTGCAGAACCTC |
| droMoj3 | scaffold_6473:11526997-11527247 + | AGCTGAAGACCAGTCTCCGTGAGCAGCGCAAGATCGCCAACAAGGCGAAGGTGAGTATGCAGAGATCTGTCTGTCTATCT-------------GGCCCTGCACTGATGATG---CCATTGCAGCAATTGGTTACAACACGCGAGCAGGAGCTGGTGAACTGCTTGACCGAGAAACAGGGTCTCCTGGTGAAACTTAACGAACTTCAGCCGGAGCTGATTGCCCAAAACGAAGTGGTGAAGGAAGCAACTGTGACAATTAAGAATCTC | |
| droWil2 | scf2_1100000004382:918596-918820 + | AACTTCTAGAGCAAATCGACCAGACTTCCGAATCCAGCAATCAGGCAAATGTAAGTATTAAGGATTTTTTTTTTTGATTTAAGAGCGTCCACAGTTTATGGACAAATTT-------TTATTAGCAAACCATACGATCACAAAAGAGCGAATTGAGTTCTTGTTTAAATGAGAATCATCGCTTAGTGAATGCTTTGGCCAATGTTGAGAATGAGCTGGTATATCAGCATAAAT----------------------------------- | |
| dp5 | 3:8172011-8172067 + | AGCTGGTGGAGCAAATCGAGAAGGCGCGGCAGTGCAGCGACGAGGCAAATGTAAGTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droPer2 | scaffold_4:2050077-2050133 + | AGCTGGTGGAGCAAATCGAGAAAGCGCGGCAGTGCAGCGACGAGGCAAATGTAAGTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droAna3 | scaffold_13266:16634732-16634791 - | AGCTGGTGGAACGCCTCCAAAAGGATACAGAATCCAGCGAAGCGGCCAATGTAAGTATCT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396384:743783-743865 - | AGCTGGTGGAACGCCTCCAGAGCCATTCAGAGTCTAGCGATGCGGCCCATGTAAGTAAACATTGTCC----TTTAACCCTAAGAGTA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droKik1 | scf7180000302277:506408-506467 + | AAATGGTGGAGCTCCTTGAGTTGAATGCCGAGGCCAGCGATGCAGCAAACGTAAGTATTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
Generated: 05/16/2015 at 10:41 PM