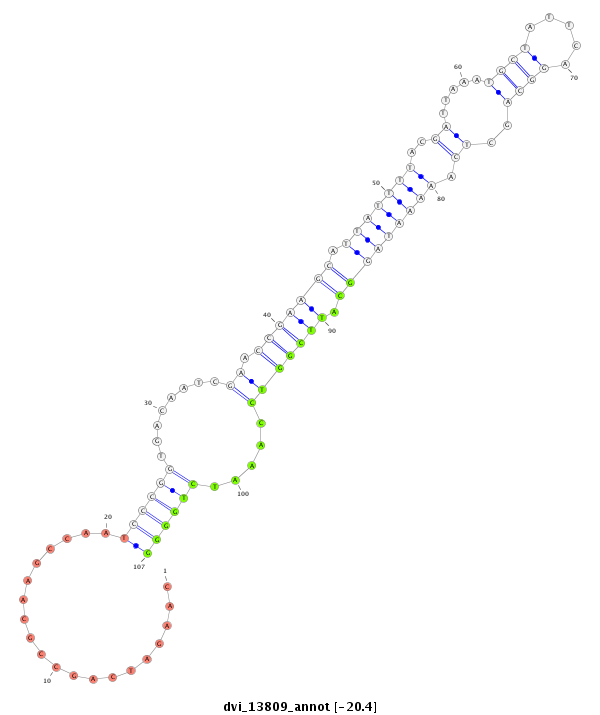
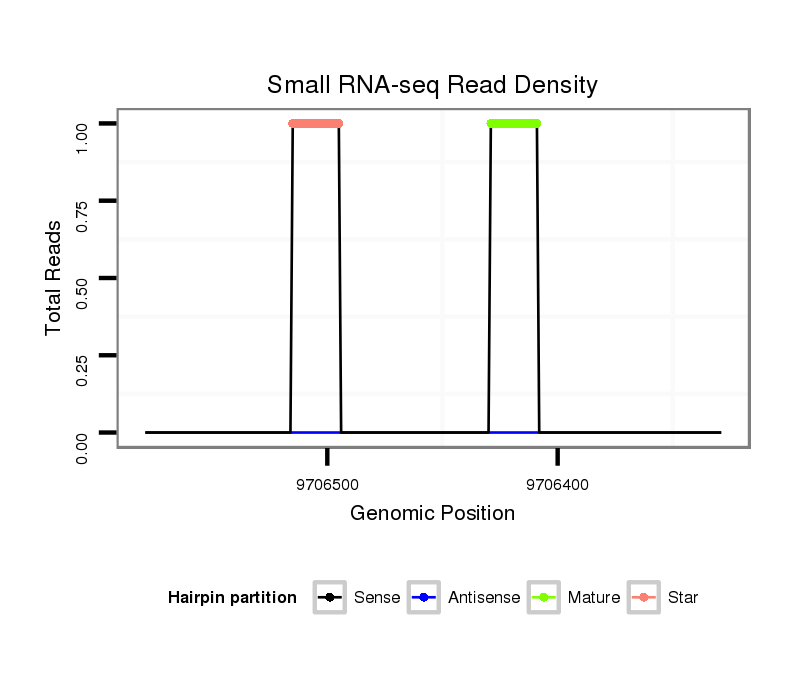
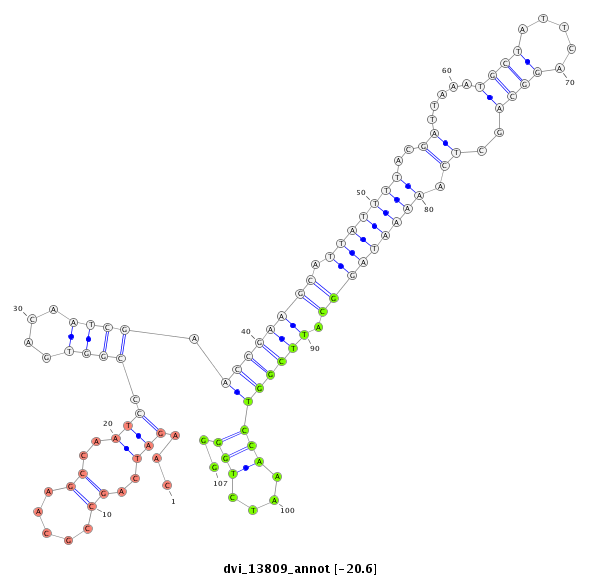
ID:dvi_13809 |
Coordinate:scaffold_12970:9706379-9706529 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
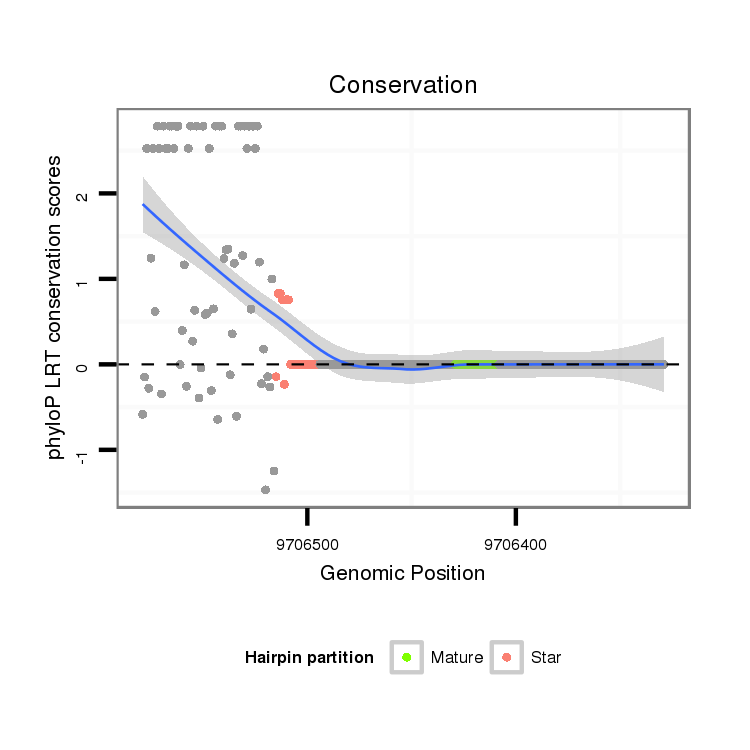
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
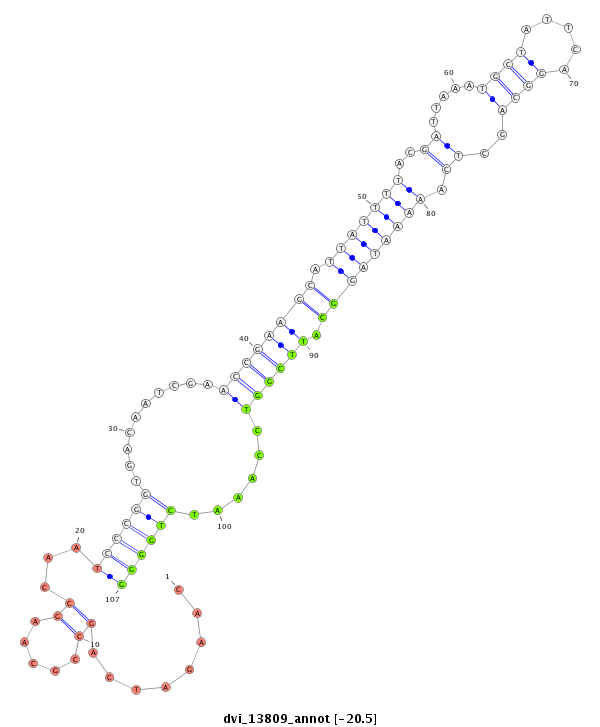
| -20.6 | -20.6 | -20.5 |
 |
 |
 |
CDS [Dvir\GJ18680-cds]; exon [dvir_GLEANR_3505:3]; intron [Dvir\GJ18680-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TTCGGCCACATGGATGATCAGCGAGCTGCTGCGACTTATGCGCACAAACAGTAAGTACTTCAATCAAGATCAGCCGCAAGCCAATCCCGGTGACAATCGAACCGAAGCATTATTTTACGATTAAATGCTATTCAGGCAGCTCAAAAATAGGCATTCGGTCCAAATCTGGGGTCTGGCTGTAGCCGAAGGTCCATCCAAGTTAAAATGAAGTCTGCAGCTACAGTAAAAACTCGCTTGGGCGGACAGCGAGC ****************************************************************....................((((((........((.(((((((.(((((((..((.....((((.....))))..)).))))))))).))))))).....))))))******************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
M047 female body |
SRR060679 140x9_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060658 140_ovaries_total |
SRR060666 160_males_carcasses_total |
SRR060659 Argentina_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................................................................GAAGTCTGGTGCTACAGTA.......................... | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................GCATTCGGTCCAAATCTGGGG................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................CAAGATCAGCCGCAAGCCAAT...................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GAAGTCTGTAGCTCCAGTA.......................... | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GAAGACTGGAGATACAGTAA......................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GCAGTCTGGTGCTACAGTA.......................... | 19 | 3 | 12 | 0.25 | 3 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................................CTTGGGCGTAAAGCGAGC | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................................CTACAGCAAAAACGCGCTCG.............. | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................................................................................GAAGTCTGGTGCTACAGTC.......................... | 19 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GAAGTGTGGTGCTACAGTA.......................... | 19 | 3 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GAAGTATGGAGCTCCAGTA.......................... | 19 | 3 | 17 | 0.06 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................GTAAATACTTCCATGAAGAT..................................................................................................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ......................................................................................................................................................................GGGGGTATGGGTGTAGCC................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AAGCCGGTGTACCTACTAGTCGCTCGACGACGCTGAATACGCGTGTTTGTCATTCATGAAGTTAGTTCTAGTCGGCGTTCGGTTAGGGCCACTGTTAGCTTGGCTTCGTAATAAAATGCTAATTTACGATAAGTCCGTCGAGTTTTTATCCGTAAGCCAGGTTTAGACCCCAGACCGACATCGGCTTCCAGGTAGGTTCAATTTTACTTCAGACGTCGATGTCATTTTTGAGCGAACCCGCCTGTCGCTCG
********************************************************************************....................((((((........((.(((((((.(((((((..((.....((((.....))))..)).))))))))).))))))).....))))))**************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M027 male body |
SRR060659 Argentina_testes_total |
SRR060660 Argentina_ovaries_total |
SRR060658 140_ovaries_total |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060661 160x9_0-2h_embryos_total |
V116 male body |
SRR060662 9x160_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................GAAGTTGGGTCTAGTCGG................................................................................................................................................................................ | 18 | 2 | 3 | 1.00 | 3 | 1 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................AAGTTGGGTCTAGTCGGGG.............................................................................................................................................................................. | 19 | 3 | 11 | 0.45 | 5 | 1 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................GAAGTTGGGTCTAGTCGGGG.............................................................................................................................................................................. | 20 | 3 | 5 | 0.40 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................CAGTGTTAGCGTGGCCTCG............................................................................................................................................... | 19 | 3 | 8 | 0.25 | 2 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................TAGGGTCACTGGTAGCTT...................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GGGCTACCAGGTGGGTTCA................................................... | 19 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................GAAGTTGGCCCTAGTCGG................................................................................................................................................................................ | 18 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................TGAAGTTAGTTCTCGACG................................................................................................................................................................................. | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................GAAGTTGGGCCTAGTCGGC............................................................................................................................................................................... | 19 | 3 | 12 | 0.08 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................TCCGTAGACCAGGTTTAGA.................................................................................... | 19 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ........................................................TGAAGTTGGGCCTAGTCGG................................................................................................................................................................................ | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................AGAAGTTGGGTCTAGTCGG................................................................................................................................................................................ | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................TAGGCGCTCGAAGACGGTGA....................................................................................................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................GGGTCACTGGTAGCTTCGC................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:9706329-9706579 - | dvi_13809 | TTCGGCCACATGGATGATCAGCGAGCTGCTGCGACTTATGCGCACAAACAGTAAGTACTT-CAATCAAGATCAGCCGCAAGCCAATCCCGGTGACAATCGAACCGAAGCATTATTTTACGATTAAATGCTATTCAGGCAGCTCAAAAATAGGCATTCGGTCCAAATCTGGGGTCTGGCTGTAGCCGAAGGTCCATCCAAGTTAAAATGAAGTCTGCAGCTACAGTAAAAACTCGCTTGGGCGGACAGCGAGC |
| droMoj3 | scaffold_6473:11863146-11863202 + | CACAGCCACCTGGATGATCAGCGAGCTGGTGCGCCTGATGCGCACGAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_15081:2655829-2655885 + | CTCGGCTACATGGATGATCAGCGAACTGTTGCGTTTGATGCGCACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004963:2467407-2467464 + | GGCAGCCACTTGGATGATCTCTGAGCTTTTACGCCTTATGAAATCGAACAGTGAGTAC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XL_group1e:1126612-1126668 - | GACGGCCACCTGGATGATCTCTGAGCTGCTGCGACTGATGAAAACCAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_30:68601-68657 + | GACGGCCACCTGGATGATCTCTGAGCTGCTGCGACTGATGAAAACCAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_13334:769023-769081 + | CACCTCCACGTGGATGATTGCCGAGATCCTGAGGCTCATAAAGAGTAATAGTGAGTATT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396733:400107-400163 + | CACCTCTACGTGGATGATTGCCGAGATCCTGAGGCTTATGAAGAGTAATAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droKik1 | scf7180000302610:1236544-1236599 - | CACGGCCACCTGGATGATAGCCGAGCTGCTCAGGCTGATGAAGACCAACAGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454077:1203552-1203607 + | GGCAGCCACTTGGATGATCGCGGAGCTCTTGCGATTAATGAAGTCAAACAGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000491013:1133810-1133875 - | CACAGCCACCTGGATGATAGCCGAGCTGCTGCGACTGATGAAGACAAACAGTGAGTGTTCTTTAAT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droRho1 | scf7180000779333:150282-150338 - | GACGGCCACCTGGATGATAGCAGAGCTCCTGCGACTGATGAAGACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302421:1224950-1225005 + | GACGGCCACCTGGATGATAGCCGAGCTGCTGCGACTGATGAAGACAAACAGTGAGT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droTak1 | scf7180000413135:29750-29806 - | GACGGCCACCTGGATGATAGCCGAGCTGCTGCGACTGATGAAGTCAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409271:35970-36026 - | GACGGCCACCTGGATGATAGCTGAGCTACTGCGACTGATGAAGACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dm3 | chrX:16977492-16977548 - | GGCGGCCACCTGGATGATTGCCGAACTGCTGCGACTAATGAAAACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSim2 | x:16095021-16095077 - | GACGGCCACCTGGATGATTGCCGAACTGCTGCGACTAATGAAAACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_17:810703-810759 - | GACGGCCACCTGGATGATTGCCGAACTGCTGCGACTAATGAAAACAAACAGTGAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | X:15588669-15588736 - | GACGGCCACCTGGATGATTGCCGAGCTGCTGCGACTAATGAAGACGAACAGTGAGTACAATCAACCAA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4690:7330332-7330403 - | GACGGCCACCTGGATGATTGCCGAATTGCTGCGACTAATGAAAACAAACAGTAAGTATTTCCAAACAAGTTC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:05 PM