
ID:dvi_13762 |
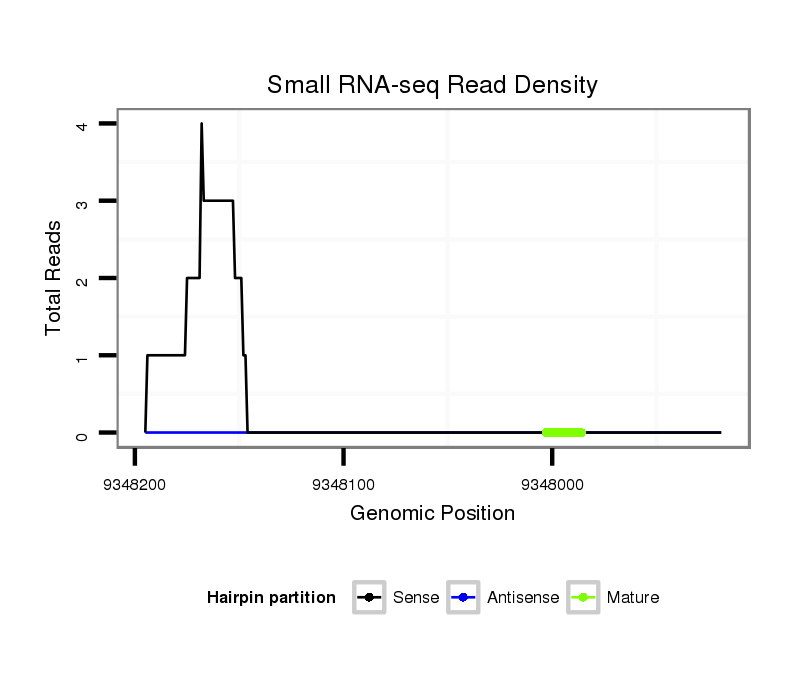
Coordinate:scaffold_12970:9347969-9348145 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -15.4 | -15.3 | -15.3 |
 |
 |
 |
exon [dvir_GLEANR_3521:4]; CDS [Dvir\GJ18697-cds]; CDS [Dvir\GJ18697-cds]; exon [dvir_GLEANR_3521:5]; intron [Dvir\GJ18697-in]
No Repeatable elements found
| mature | star |
| ##################################################---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## CCCGAGGTCACTGAAATCATAGATGATGATCACGGAAAGCAAGCGAATAAGTGAGTGTCCCTTGCCGACTAGTTCAGTCGAATGTTTCCGATTAAATTCATTCTTAATACTACTTATTCAAAATTCTTTGTCAGTTCATCAAAATTTCTTTAAATTTTTGTTTACACAATTTGCTTACAATTTAGGAAAGCTTTCGATATTATAATTAAGATTTTATACTTTTATAGAACACAAATGATGCCGCCACCACCCGGGCTGTTGATGTCCCGGACTAAAG **************************************************(((((((((....(((((((.....)))))...))....)))...))))))(((((((......((((.(((.((((((((.(((......(((((....(((((....)))))...)))))))).))))....))))...))).)))).....)))))))................************************************************** |
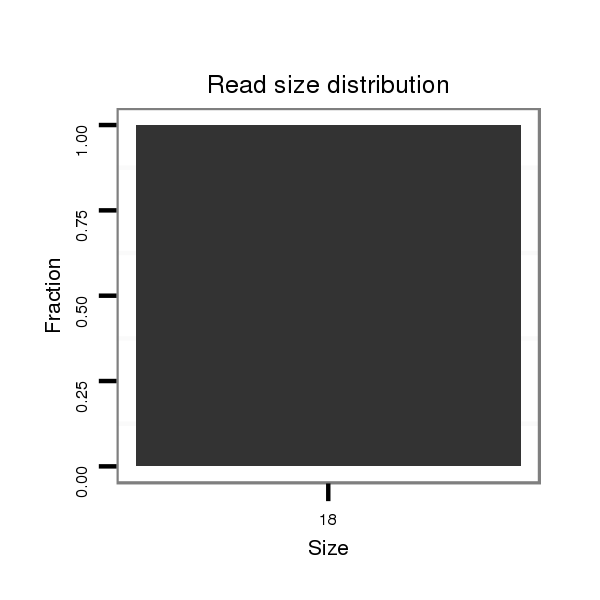
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060661 160x9_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
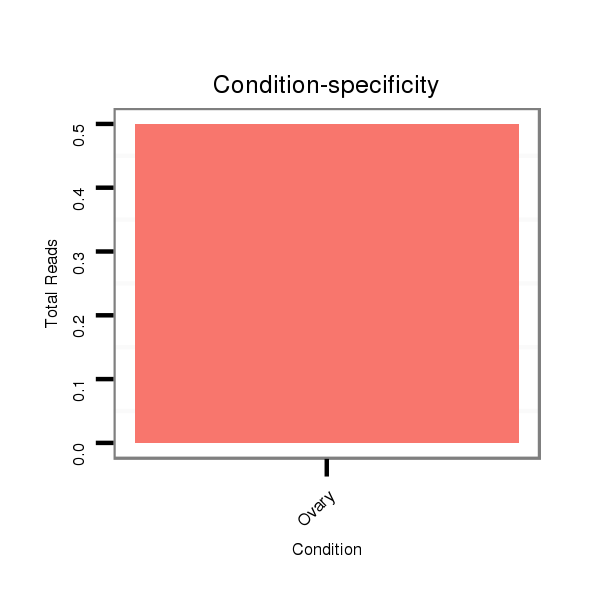
SRR060677 Argx9_ovaries_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060675 140x9_ovaries_total |
SRR060679 140x9_testes_total |
V047 embryo |
M027 male body |
SRR060666 160_males_carcasses_total |
SRR060689 160x9_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...........................GATCACGGAAAGCAAGCGAA...................................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .CCGAGGTCACTGAAATCATAGATGATG......................................................................................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................AGATGATGATCACGGAAAGCAAG.......................................................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................GATCACGGAAAGCAAGCGAATA.................................................................................................................................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................TAAGTGATTGTCTCTTGCCGA................................................................................................................................................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............GAAATTATAGATGATGATCAT.................................................................................................................................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................TTCGATATTATAATTAAG................................................................... | 18 | 0 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................TGAATTCATTCTTAATACTGAT................................................................................................................................................................... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................GAATTTTTACTTTTATAGAACC.............................................. | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................CGAAGGAGTGAGGGTCCC........................................................................................................................................................................................................................ | 18 | 3 | 15 | 0.13 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| .....................TATGATGATGACGGATAGC............................................................................................................................................................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
GGGCTCCAGTGACTTTAGTATCTACTACTAGTGCCTTTCGTTCGCTTATTCACTCACAGGGAACGGCTGATCAAGTCAGCTTACAAAGGCTAATTTAAGTAAGAATTATGATGAATAAGTTTTAAGAAACAGTCAAGTAGTTTTAAAGAAATTTAAAAACAAATGTGTTAAACGAATGTTAAATCCTTTCGAAAGCTATAATATTAATTCTAAAATATGAAAATATCTTGTGTTTACTACGGCGGTGGTGGGCCCGACAACTACAGGGCCTGATTTC
**************************************************(((((((((....(((((((.....)))))...))....)))...))))))(((((((......((((.(((.((((((((.(((......(((((....(((((....)))))...)))))))).))))....))))...))).)))).....)))))))................************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
M047 female body |
SRR060672 9x160_females_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060660 Argentina_ovaries_total |
SRR060664 9_males_carcasses_total |
M027 male body |
SRR060658 140_ovaries_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................................................................................................................................................GCGGCGGTGCGCCCGACA.................. | 18 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................................TGCGGTGGGCCCGACAACTA.............. | 20 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................AAGTTGTAAGAAACGGTCAA............................................................................................................................................. | 20 | 2 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................................................ACGATGATGGTGGGCCCG..................... | 18 | 3 | 20 | 0.25 | 5 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 2 | 0 |
| ....TCCAGTGACTTGAGTAACT.............................................................................................................................................................................................................................................................. | 19 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................................CTTGTGATTGCTACGGGGG................................ | 19 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................TTTAAAAACAAATGTGTTG........................................................................................................... | 19 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................CAGGTCACCTCACAAAGGC........................................................................................................................................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................................................................................................................................................................TGGCCACGACTACTACAGG.......... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
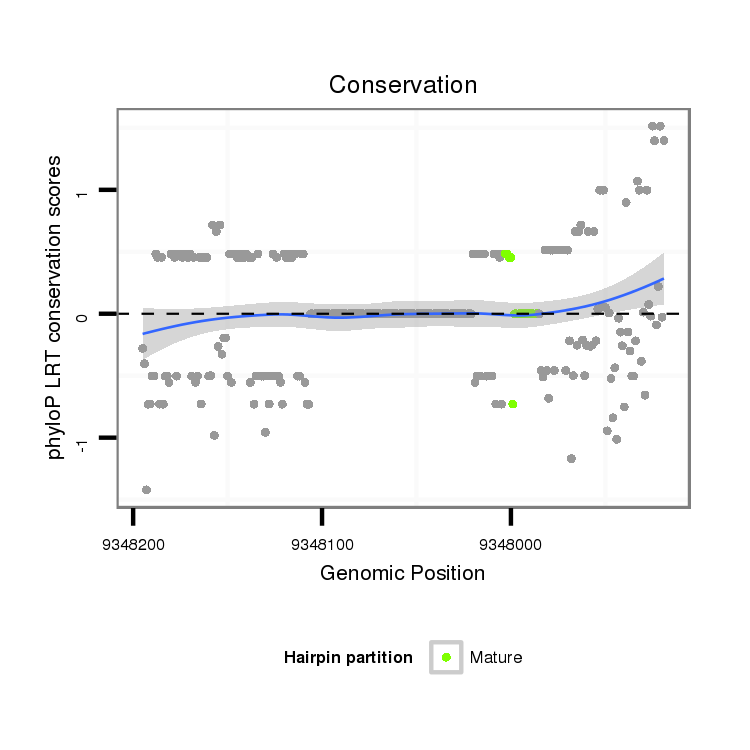
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:9347919-9348195 - | dvi_13762 | CCCGAGGTCACTGAAATCATAGATGATGATCACGGAAAGCAAGCGAATAAGTGAGTGTCCCTTGCCGA-----------CTAGTTCAGTCGAATGTTTC-----------CGATTAAATTCATTCTTAATACTACTTATTCAAAATTCTTTGTCAGTTCATCAAAATTTCTTTAAATTTTTGTTTACACAATTTGCTTACAATTTAGGAAAGCTTTCGATATTATAATTAAGATTTTATACTTTTATAGAACACAAATGATGCCGCCACCACCCGGGCTGTTGATGTCCCGGACTAAAG |
| droMoj3 | scaffold_6308:1385246-1385429 + | CCGTCAATCCCGAGTATCGTAGATGACGTCCCCGGGCAACTATCGGAAAAGTGAGTGACATTCATGACAGTGCTATCTTTTGGCAAAGTCGAACATTAATTCATTGTTTCA--------------------------------------------------------------------------------------TTTAGTTCGAAACAGATTTCGC-----------------------------AGCCGCAACCGTCGTCGTCGTCTAAAGAGTTACCAATGCCACGCACTAAAG | |
| droGri2 | scaffold_15203:2038099-2038164 + | TAA---------------------------------AATCAAGTA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CATCCTATTTTTACAGCACACAGATGATGCTGCCAAAT------------TTGTCCCGGACTAAAG | |
| droWil2 | scf2_1100000004909:10595278-10595313 + | TAT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CGCCATCATCGAGCATGT---TGCAACAGACGGAGG | |
| droFic1 | scf7180000451789:118730-118730 - | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000302187:1078-1085 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACCGATG | |
| droTak1 | scf7180000415281:100118-100125 - | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACCGATG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/16/2015 at 09:36 PM