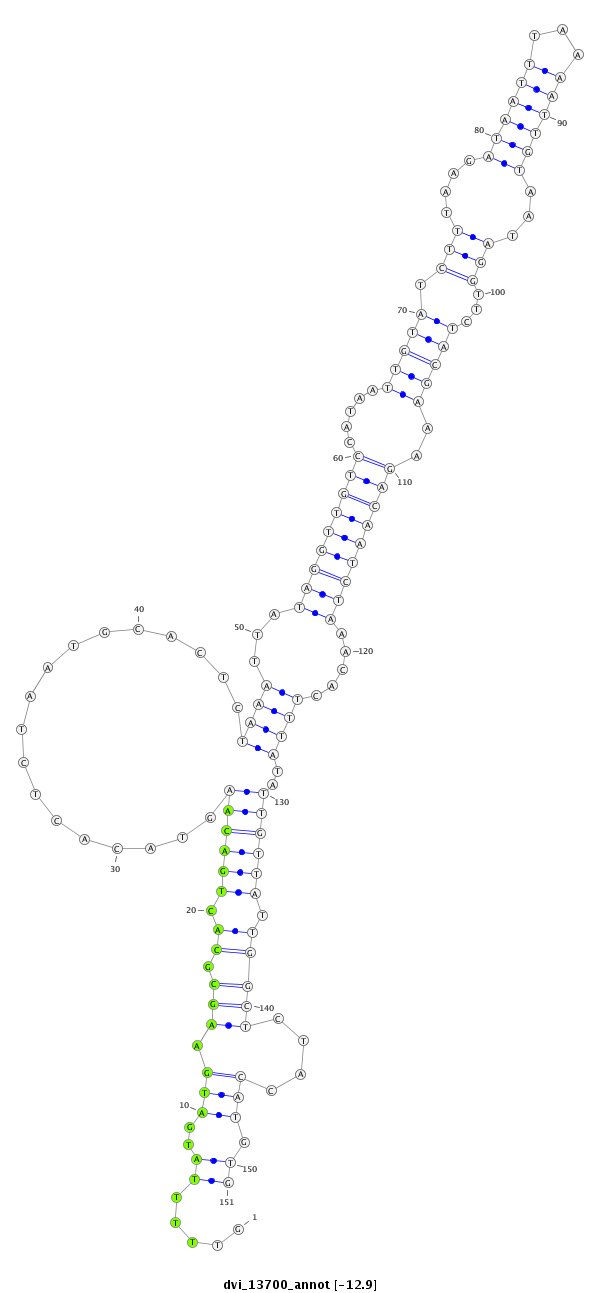
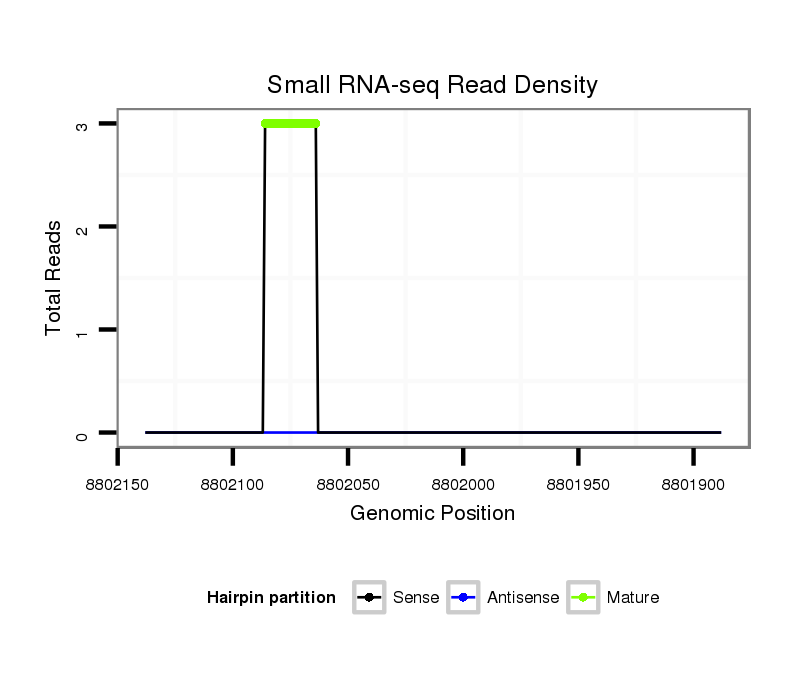
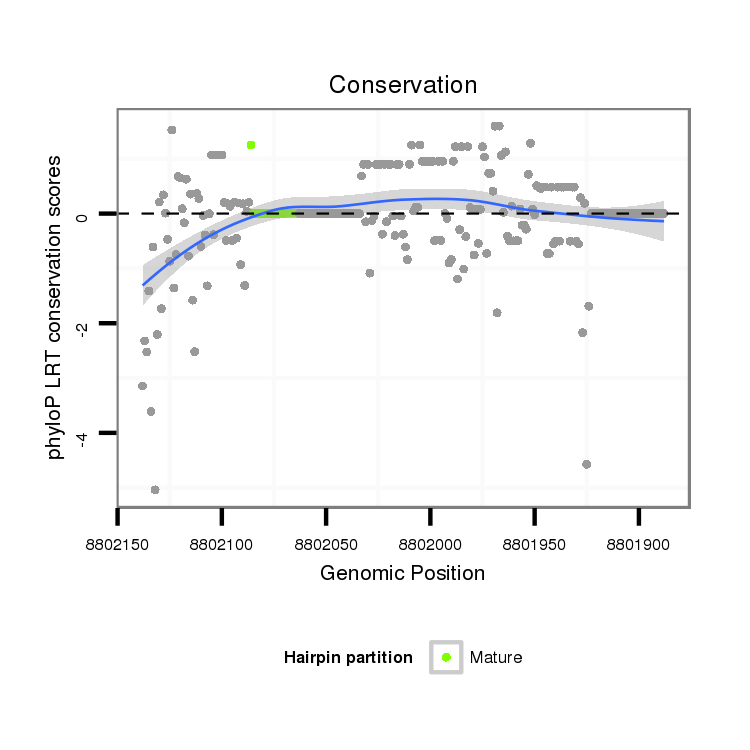
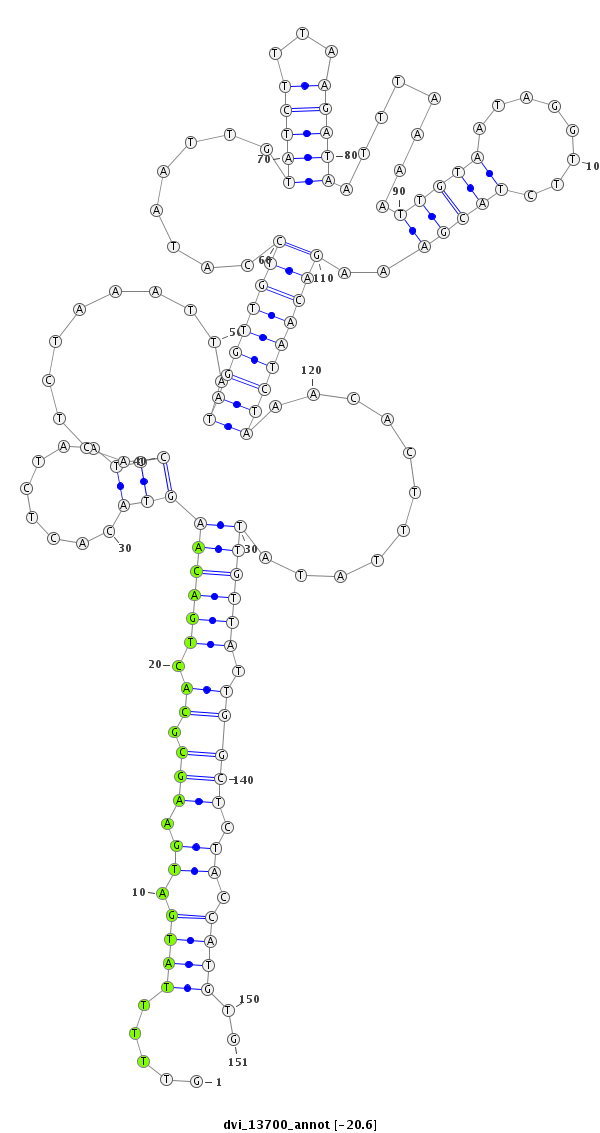
ID:dvi_13700 |
Coordinate:scaffold_12970:8801938-8802088 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.7 | -20.6 | -20.5 | -20.5 |
 |
 |
 |
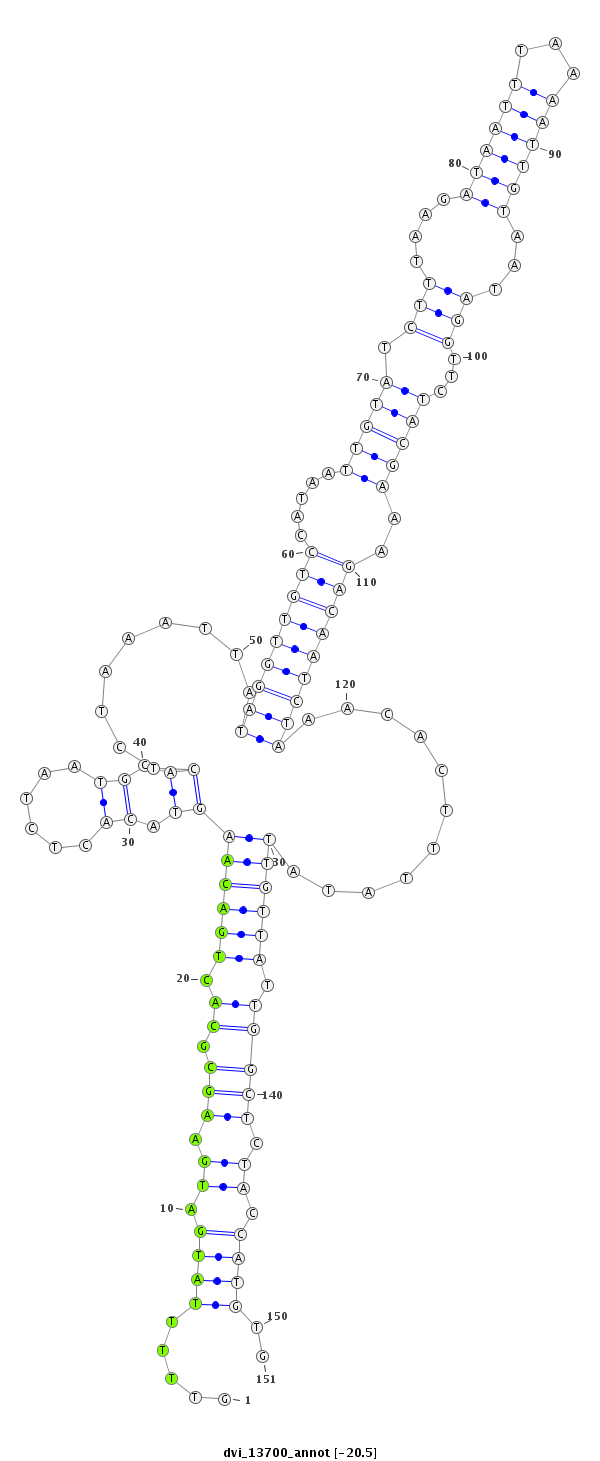 |
exon [dvir_GLEANR_3554:1]; CDS [Dvir\GJ18727-cds]; intron [Dvir\GJ18727-in]
No Repeatable elements found
| ----------------------------######################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TTAGCTGCTGTTAATGTATTAAATTAATATGAAACATTTTGAAGTTAAAGGTTTTTATGATGAAGCGCACTGACAAGTACACTCTAATGCACTCTAAATTATAGGTTGTCCATAATTGTATCTTTAAGATAATTTAAAATTGTAATAGGTTCTACGAAAGACAATCTAAACACTTTATATTGTTATTGGCTCTACCATGTGATTTGAAGAATTCAACTGGAGAACGAAAAGTGTTTTATTGAGTGTGGGAT **************************************************.....((..(((.(((.((.((((((..................((((...(((((((((.....(((((.(((....((((((...))))))...)))...)))))..))))))))).....))))..)))))).)))))....))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060658 140_ovaries_total |
SRR060677 Argx9_ovaries_total |
M028 head |
M027 male body |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
SRR060659 Argentina_testes_total |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060665 9_females_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
V116 male body |
M047 female body |
SRR060660 Argentina_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060679 140x9_testes_total |
SRR060681 Argx9_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................ATGTGAATTGCAGAATTC..................................... | 18 | 2 | 17 | 3.18 | 54 | 0 | 0 | 10 | 10 | 0 | 7 | 5 | 1 | 3 | 3 | 3 | 2 | 2 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 |
| ....................................................TTTTATGATGAAGCGCACTGACA................................................................................................................................................................................ | 23 | 0 | 1 | 3.00 | 3 | 2 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................CAAGTCCACTCTCATGTACT.............................................................................................................................................................. | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................ATCTGGTTTGAAGAATTC..................................... | 18 | 2 | 9 | 0.22 | 2 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................GTGAATTGAAGAAATCAAC.................................. | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................TGTGAATTGCAGAATTCA.................................... | 18 | 2 | 17 | 0.12 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................................................................................................................................................CTTGTGATTTGGAGCATTCA.................................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................CATGTGAATTGCAGAATTCC.................................... | 20 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AATCGACGACAATTACATAATTTAATTATACTTTGTAAAACTTCAATTTCCAAAAATACTACTTCGCGTGACTGTTCATGTGAGATTACGTGAGATTTAATATCCAACAGGTATTAACATAGAAATTCTATTAAATTTTAACATTATCCAAGATGCTTTCTGTTAGATTTGTGAAATATAACAATAACCGAGATGGTACACTAAACTTCTTAAGTTGACCTCTTGCTTTTCACAAAATAACTCACACCCTA
**************************************************.....((..(((.(((.((.((((((..................((((...(((((((((.....(((((.(((....((((((...))))))...)))...)))))..))))))))).....))))..)))))).)))))....))).))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060680 9xArg_testes_total |
SRR060687 9_0-2h_embryos_total |
M061 embryo |
V053 head |
SRR060659 Argentina_testes_total |
SRR060681 Argx9_testes_total |
SRR060675 140x9_ovaries_total |
SRR1106727 larvae |
SRR060685 9xArg_0-2h_embryos_total |
M028 head |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................TGTAAAGCTTCAATTTGCA....................................................................................................................................................................................................... | 19 | 2 | 3 | 0.67 | 2 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TATGTAAAGCTTCAATTT.......................................................................................................................................................................................................... | 18 | 2 | 20 | 0.60 | 12 | 11 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TATGTAAAGCTTCAATTTC......................................................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................TGTAAAGCTTCAATTTGCAT...................................................................................................................................................................................................... | 20 | 3 | 11 | 0.18 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...............................TATGTAAAGCTTCAAATTCCA....................................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................TGTAAAGCTTCAATTTGC........................................................................................................................................................................................................ | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................CTGTTCATCTGATGTTACGTG............................................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................TGTTTTCTGTTAGATTTG................................................................................ | 18 | 1 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................TCCGAGATGGTAGACT................................................. | 16 | 2 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 |
| .................................TGTAAAGCTTCAATTTGCCA...................................................................................................................................................................................................... | 20 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................................GACGACTTGCTTTTCACCA................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................TGTAAAGCTTCAATTTTC........................................................................................................................................................................................................ | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................GCTTTATGTTAGTTTTGTGAT............................................................................ | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................AGGTATTAACCGAGGAATT............................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................AGGGATTAACATAGAGAGT............................................................................................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:8801888-8802138 - | dvi_13700 | TTAGCTGCTGTTAATGTATTAAATTAATATGAAACATTTTGAAGTTAAAGGTTTTTATGATGAAGCGCACTGACAAGTACACTCTAATGCACTCTAAATTATAGGTTGTCCATAATTGTATCTTTAAGATAATTTAAAATTGTAA-TAGGTTCTACGAAAGACAATC-TA-AACACTTTATATTGTT----ATTGGCTCTAC--CATGTGATTTGAAGAATTCAACTGGAGAACGAAAAGTGTTTTATTGAGTGTGGGAT |
| droMoj3 | scaffold_6496:10048409-10048544 + | AATGTAT-TCTTGC-TCATAAAA-----------------------------------------------------------------------------------TATGCATAAATATATTTTTTCTATATTTTAAAATATTTAGTACATTGTAGAAAAAAAAA-AA--AAAGACTTTTTTAAATTGCGCGTTGACTCTAATGAAAATAATTTAAAATATTTA------------------------------------ | |
| droGri2 | scaffold_15245:10320400-10320445 - | AGCAAAC--------AAACAAAATTCAAA---------------------------------------------------------------------------------------------------------------------------------------------AAATACCTCTTTAAGCCAAGAGT-----------------------------TT------------------------------------ | |
| droWil2 | scf2_1100000004762:4107712-4107767 - | TTTAAACC-AAAAA-------------------------------------------------------------------------------------------TTGTTATTAATTGTAATTTAAAAATCAAT------------------------------------AAATA------------------TTGT----------------------ATTAG------------------------------------ | |
| droPer2 | scaffold_2:6417661-6417675 - | CTGATTGTTGTCAA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T------------------------------------ | |
| droAna3 | scaffold_13340:21844446-21844460 + | CATGCTGAT--TAAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GT------------------------------------ | |
| droBip1 | scf7180000396759:1282664-1282705 + | AATTTAA-CATTA--TCATAGGC--------------------------------------------------------------------------------------------------------------------------------TACACGATAATTAAT-----------------------------TT----------------------AAACA------------------------------------ | |
| droKik1 | scf7180000302388:1356427-1356477 - | TAAGCTAAAGTTTTTATATTGATTTGATG-----T----TTATTTAAAATGTT----------------------------------------------------------------------------------------------------------------------------------------------TT----------------------TATAA------------------------------------ | |
| droFic1 | scf7180000454050:635652-635713 - | TTTTATA--------------------------------------------------------------------------------------------------------------------------TAATTTAAAATAGTTA-CCCCTTCAATAATTTATTATA-TA-AACACTTAACATTG-------------------------------------TT------------------------------------ | |
| droEle1 | scf7180000491193:148545-148587 - | TGAAATAA-A-------------TAAATGAACAACATTTTGAAGTAGAAGATT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTA------------------------------------ | |
| droRho1 | scf7180000778995:15370-15394 + | GTAGTTGCTGTTGTTTCATAAAA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CT------------------------------------ | |
| droBia1 | scf7180000302126:39941-39999 - | AGCTACT-TATTGATGTATAAATTAGGTATAATATATTTAGAAGTTACATATT----------------------------------------------------------------------------------------------------------------------------------------------TT----------------------TAAGA------------------------------------ | |
| droSim2 | 3l:10850605-10850655 - | AATGTATATGTAAA---------TTGATATGGAACATTTTACAAATAGCAACT----------------------------------------------------------------------------------------------------------------------------------------------TT----------------------TGTGA------------------------------------ | |
| droSec2 | scaffold_0:10111585-10111627 - | TAAGCTCCTACAAATGTATAGAGACCATG---------------------------------------------------------------------------------------------------------------------------------------------GGAGA------------------TGTT----------------------TCTAA------------------------------------ | |
| droYak3 | 3R:6510616-6510635 + | TTTGTTGCTGTTGGTGTGT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------T------------------------------------ |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
Generated: 05/16/2015 at 08:58 PM