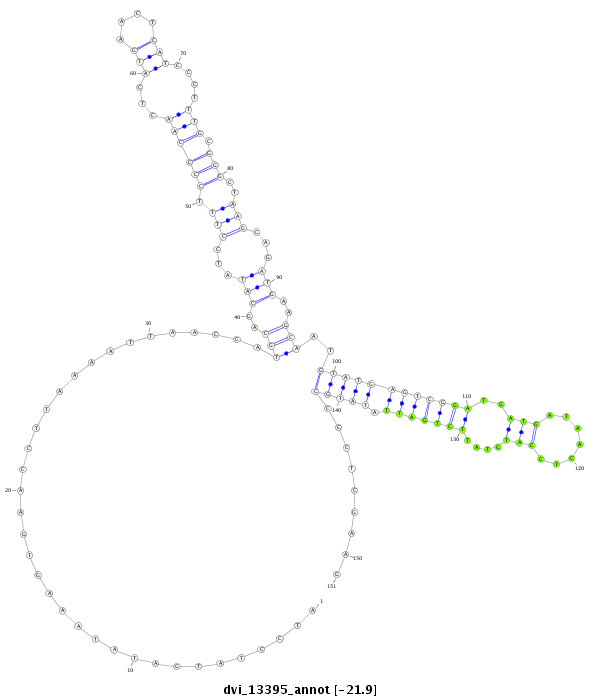
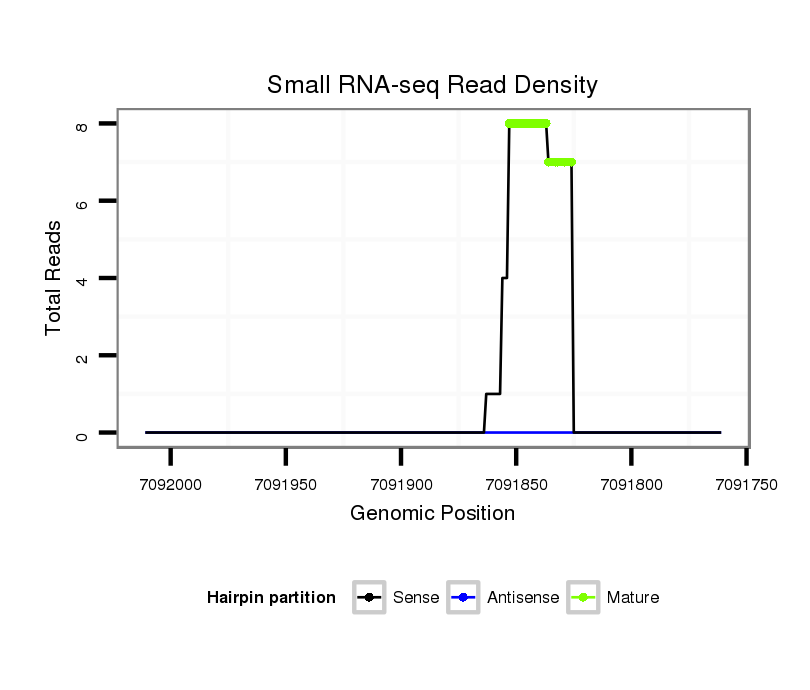

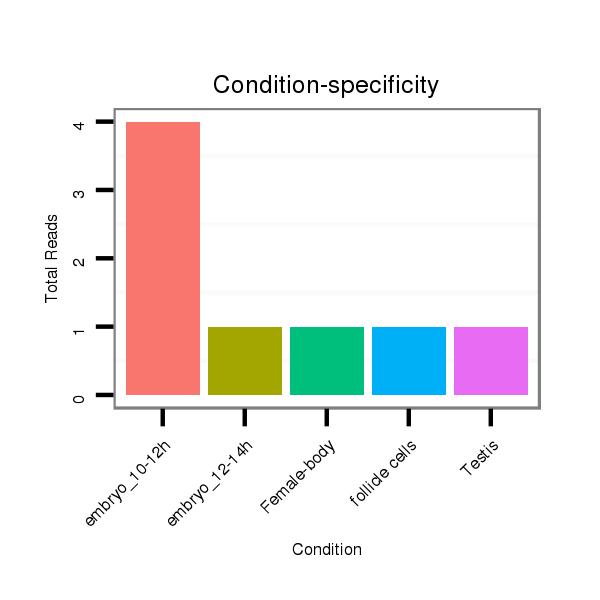
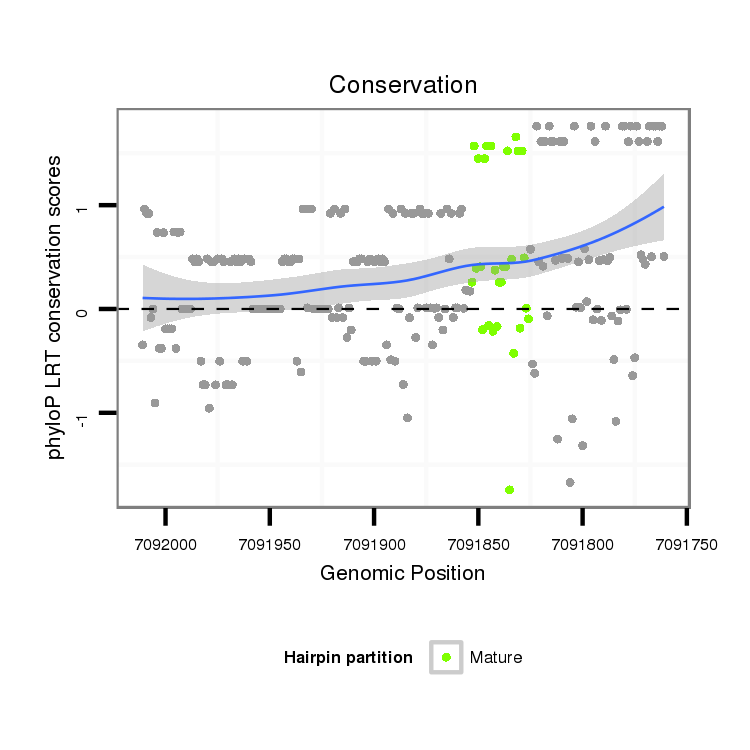
ID:dvi_13395 |
Coordinate:scaffold_12970:7091811-7091961 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.9 |
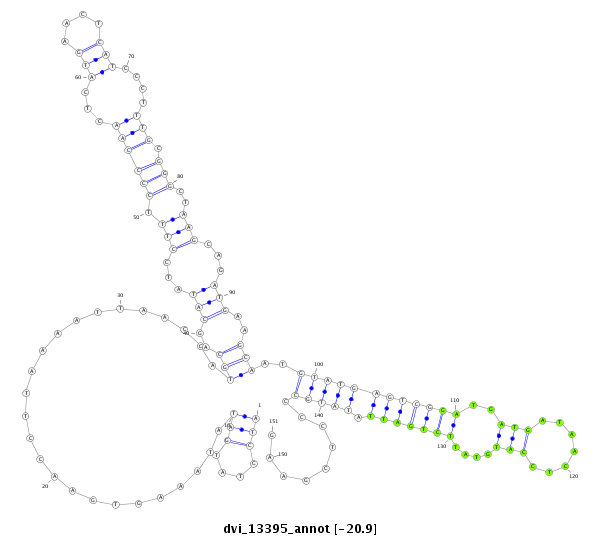 |
CDS [Dvir\GJ18821-cds]; exon [dvir_GLEANR_3640:2]; intron [Dvir\GJ18821-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AACCCAGCCCCTTTATACAAGTCTTGACTCCAGCGGTCAGAACAACACAAATCCTATGATATAAAGTGAACCTTAAAATTAACCATGCAGCATATCCTTTCCCCAACTCATGAACTCATCCCTTTGCGGGCTAAGCAGATGAAGCAATGTATGAGTCGGATGATGATAACTCCATGTATTCTGATTATATGCCCCTCGAAGAGCAAGTCCAATTGAGCGCGAAGACTACCAAGCTGCTGCAAGAGATCAAG **************************************************...................................(((..(((...(((.((((((...(((....)))....))).)))..)))...)))..)))..((((((((((((..(((.......)))....))))))).))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106722 embryo_10-12h |
GSM1528803 follicle cells |
M047 female body |
SRR060679 140x9_testes_total |
SRR1106723 embryo_12-14h |
M027 male body |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................................................................................................................................GATGATGATAACTCCATGTATTCTGATT................................................................. | 28 | 0 | 1 | 4.00 | 4 | 2 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................TCGGATGATGATAACTCCATGTATTCTGATT................................................................. | 31 | 0 | 1 | 3.00 | 3 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................GTATGAGTCGGATGATGATAACTCCAT............................................................................ | 27 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................TGAAGAGCAAGTCCAATTGAGCGCGAAGACTA....................... | 32 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................GGTGATGATACCTCCATGTT......................................................................... | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .......................................................................................................CAACTGATGAACTGATCCC................................................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................ATTCTTTCCCCAACT............................................................................................................................................... | 15 | 1 | 19 | 0.16 | 3 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................CACAAATCCAACGGTATAAAG......................................................................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........................................................................................................................................................GTGTCGGATGATGATCAGT................................................................................ | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...................................................................................................TACCCTACTGATGAACTCA..................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TTGGGTCGGGGAAATATGTTCAGAACTGAGGTCGCCAGTCTTGTTGTGTTTAGGATACTATATTTCACTTGGAATTTTAATTGGTACGTCGTATAGGAAAGGGGTTGAGTACTTGAGTAGGGAAACGCCCGATTCGTCTACTTCGTTACATACTCAGCCTACTACTATTGAGGTACATAAGACTAATATACGGGGAGCTTCTCGTTCAGGTTAACTCGCGCTTCTGATGGTTCGACGACGTTCTCTAGTTC
**************************************************...................................(((..(((...(((.((((((...(((....)))....))).)))..)))...)))..)))..((((((((((((..(((.......)))....))))))).))))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060678 9x140_testes_total |
M061 embryo |
SRR060681 Argx9_testes_total |
M028 head |
SRR060672 9x160_females_carcasses_total |
SRR060679 140x9_testes_total |
V116 male body |
V047 embryo |
M027 male body |
SRR060658 140_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................GTCGTGTAGGGAAGGGGTTGA............................................................................................................................................... | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................GGTTAACTGGCGCTTCTTAT....................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........CGAAATATGCTCAGAGCTGA.............................................................................................................................................................................................................................. | 20 | 3 | 6 | 0.33 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................TAGGGAGACACCCGCTTCGT.................................................................................................................. | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................GATTGGTCTGCTTCGTTA....................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................AGACTAGTTTACGGGGAG...................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................GGATCTTCTCGTTCGGGG........................................ | 18 | 3 | 20 | 0.20 | 4 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | 1 |
| ............................................................................................................................................................................................TACGGGGACCTTATGGTTC............................................ | 19 | 3 | 14 | 0.14 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................CCGGACTTTTTGTGTTTAGGA.................................................................................................................................................................................................... | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................GGAGTTTTTCGGTCAGGTTA...................................... | 20 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................GGATCGTCTCGTTCAGGGT....................................... | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ................................................................................................................................................................................................CGGAGCTTCTCGTTCAGTG........................................ | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..................................................................................................................................................................................CAGACTAGTTTACGGGGAG...................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:7091761-7092011 - | dvi_13395 | AACCCAGCCCCTTTATACAAGTCTTGACTCCAGCGGTCAGAACAACACAAATCCTATGATATAAAGTGAAC---------CTTAA---------------AATTAACCATGCAGCATATCCTTTCC-CCAACTCATGAACTCATCCCT--TTGCGGGCTAAGC--AGATGAAGCAATGTATGAGTCGGATGATGATAACTCC----------ATGTATTCTGATTATATGCCCCTCGAAGAGCAAGTCCAATTGAGCGCGAAGACTACCAAGCTGCTGCAAGAGATCAAG |
| droMoj3 | scaffold_6473:12585383-12585623 - | GACCCAC-----------------TGACCAAACCGTTTAGCCCCACACGAGTC--------------GAACAATCACTTGCTTGAAATTCAGTCTCCTTACATTAA--------CATATCCTCTCCCCCAATCCACGGACTCGTACTTACTTTCCGGCCAAGCACAGACGAAGCAATGTATGAGTCAGATGAAGATAGCGCC----------ATGTACTCAGATTATATGCCCCTCGAAGAGCAAGTTCAATTGAGCGCCAAGACCTCGAAGCTCCTGCAAGAGATCAAG | |
| droWil2 | scf2_1100000004909:7796442-7796534 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATGATGAAAATACC----------ATGAATTCTGGCTACTTTCCCTTCGAGGAGCTGCTCACTACGAGCGCCAAGAATGTGAATCTATTGCAAGAGATCAAA | |
| droAna3 | scaffold_13334:556227-556322 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGGACGACGACAGCTCC----------ATGAACTCGGACTACTTGCCCTTCGATGAGCTTCTGCACTCCAGCACCAAGAACGTGAACCTCCTGCAGGAGATCAAG | |
| droBip1 | scf7180000396431:997982-998077 - | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGGATGACGACAGCTCC----------ATGGACTCGGACCTGGTGCCCTTCGACGAGATCATCCGTTCGAACACCAAGAGCATGAACCTCGTGCAGGAGATCAAG | |
| droRho1 | scf7180000771612:77666-77730 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATCTGCGCATCGAGGAGCTGCTCCACTTGACCCACCATAATTTGAACCTGGTGTCGGATATCAAG | |
| droBia1 | scf7180000302421:1369163-1369341 + | AACCATCCAACCCCGTCCA--------------------------------------------------------------------------------ACATCAA--------CCTCCCATCCG-----------------ATCC-C--ATTCATGCCGAGC--GGGTAGCGGTA-GGGCGAACTGCATGGCGATAACTGGCCACCAAATATCGGGATCGGATAACCTGCGCATCGGCGTGCTGTTCCACTTGACCCATCATCATGCAAACCTGGTGCCGGAGATCAAG | |
| droTak1 | scf7180000415568:93963-94038 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAATCGGATAACCTGCGCATCGGCGTGCTCTTCCAGTTGACCCATCATCACGCGAACCTGGTGCCGGAGATCAAG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/17/2015 at 04:02 AM