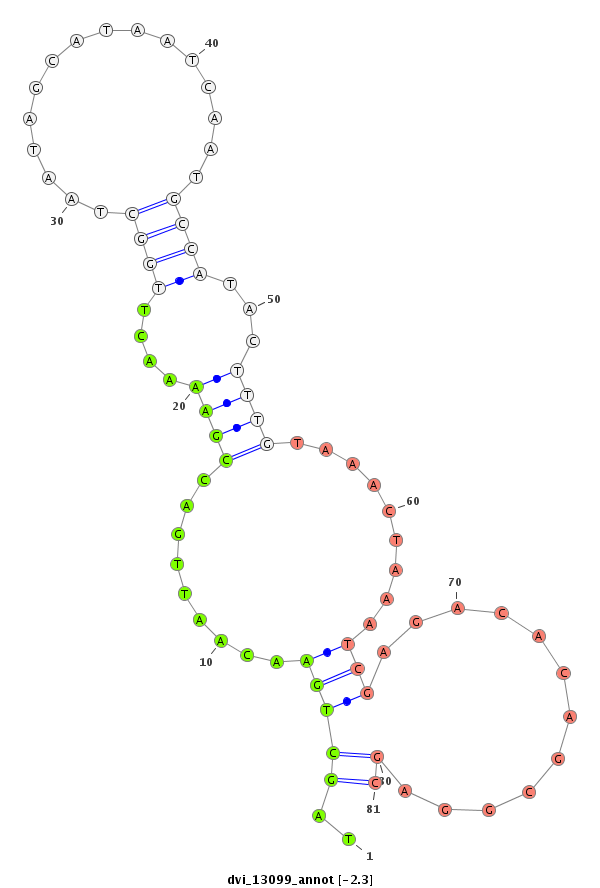
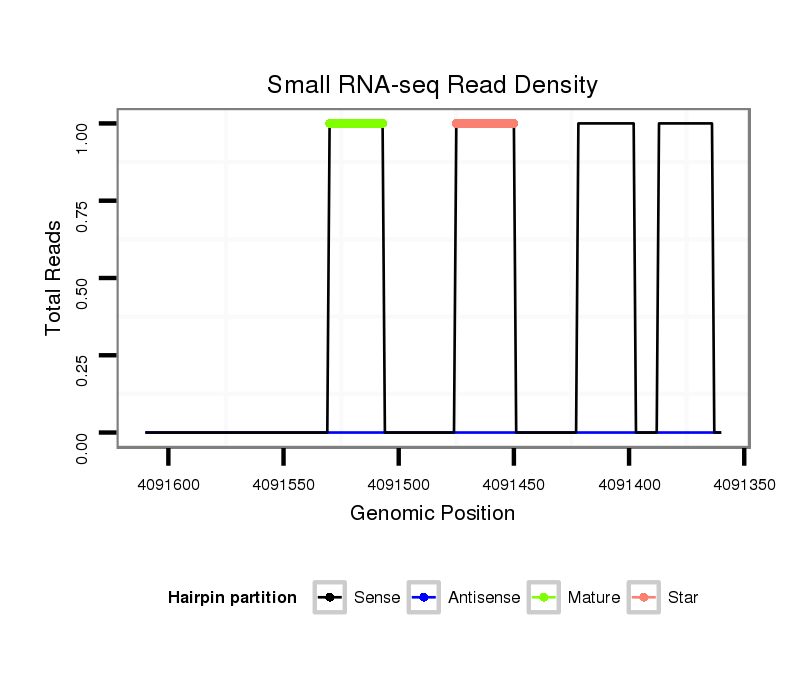
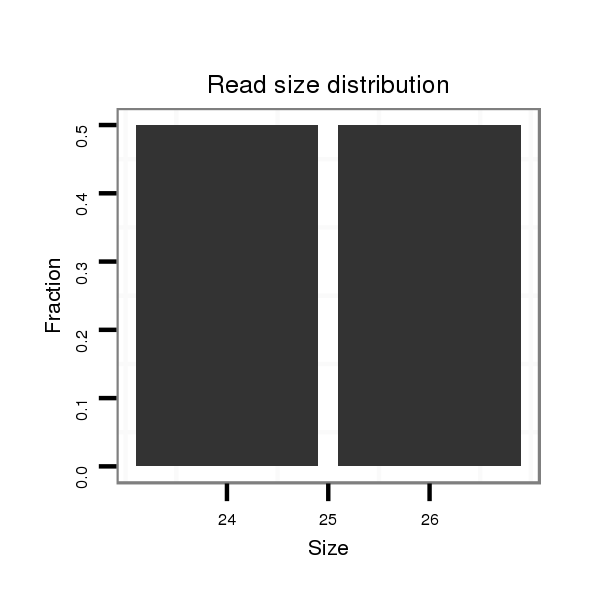
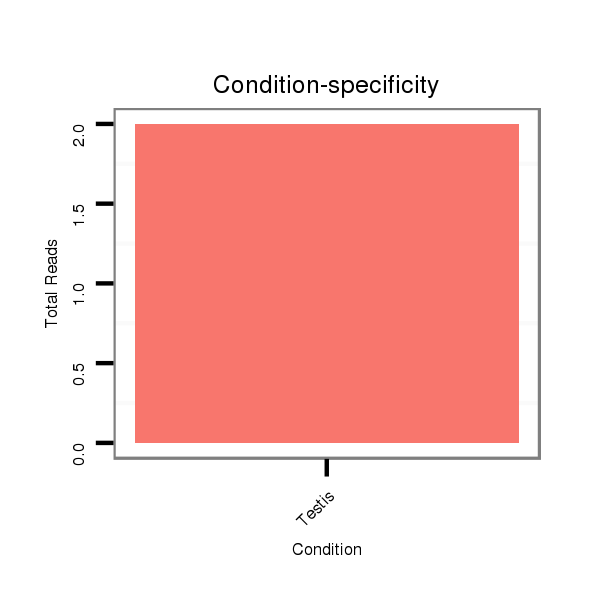
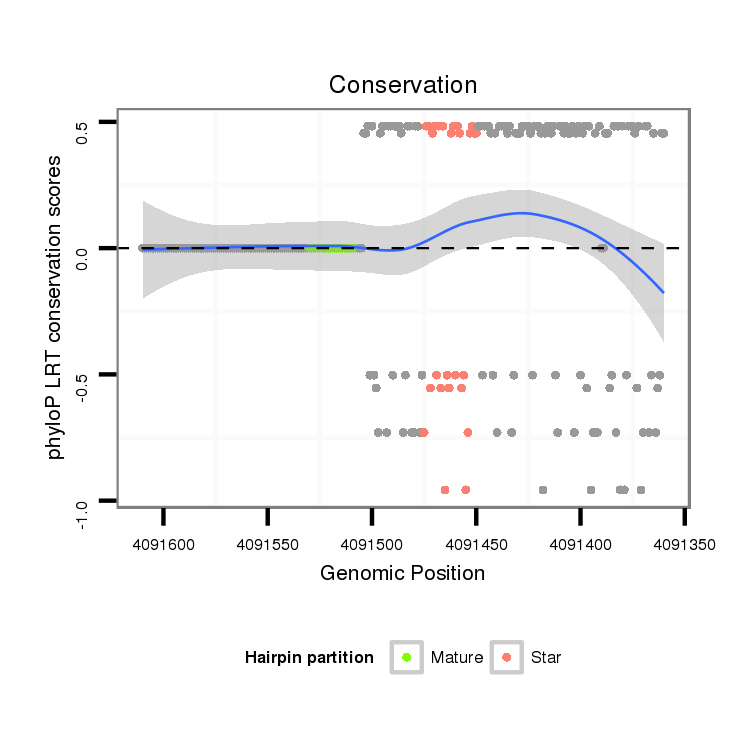
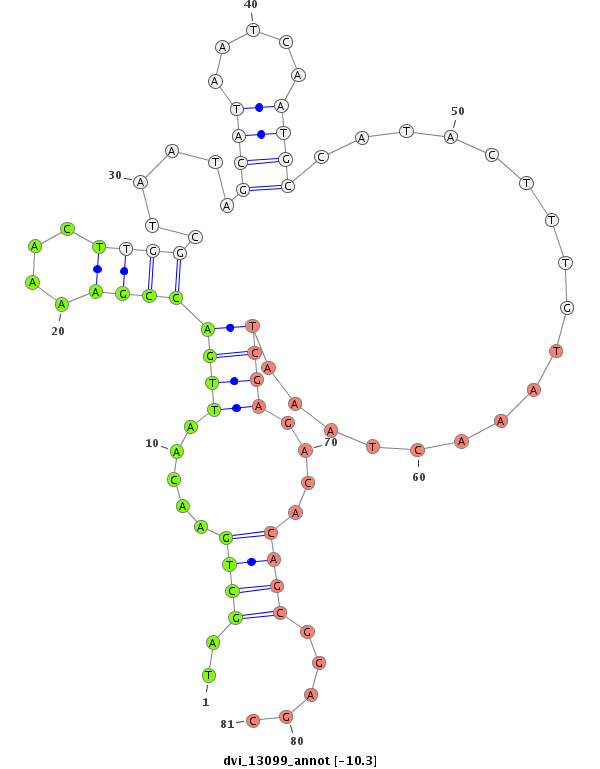
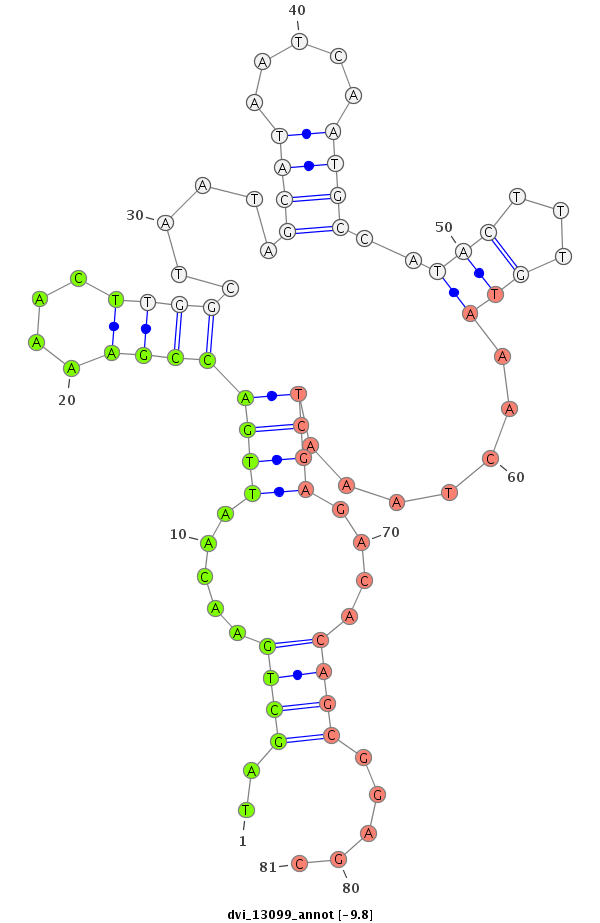
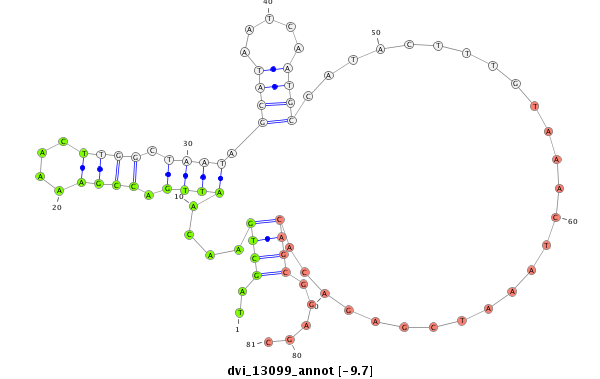
ID:dvi_13099 |
Coordinate:scaffold_12970:4091410-4091560 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
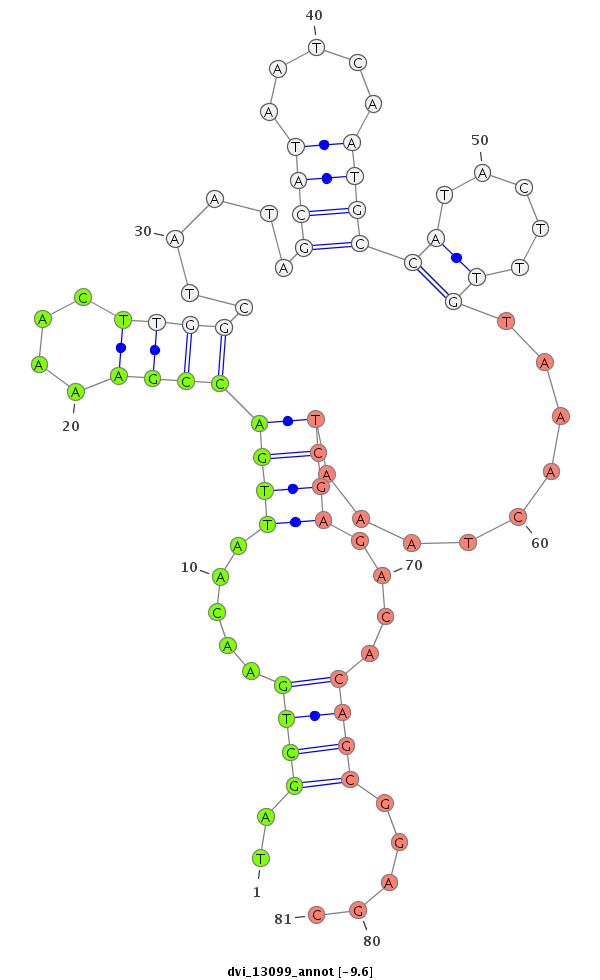
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -10.3 | -9.8 | -9.7 | -9.6 |
 |
 |
 |
 |
exon [dvir_GLEANR_3757:1]; CDS [Dvir\GJ18946-cds]; intron [Dvir\GJ18946-in]
No Repeatable elements found
| --------------------------------##################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TGACTATTTGACTAATGAGTTTATTCAAAAACATGCCAAAGGCAAAACCGGTAAGAAATCTCGGGATTTGATTGAGCCCTTAGCTGAACAATTGACCGAAAACTTGGCTAATAGCATAATCAATGCCATACTTTGTAAACTAAATCGAGACACAGCGGAGCATCTAACTGATTATGTAACCGAATACGTAACCGAAGACCTTCTCGAAGAACTTTGTGAAGAGCTCAGTGACGATACATCCAAGTGCTCGG ********************************************************************************..(((((.........((((....((((................))))...)))).........)))............))****************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060655 9x160_testes_total |
SRR060670 9_testes_total |
SRR060678 9x140_testes_total |
SRR060680 9xArg_testes_total |
SRR060683 160_testes_total |
SRR060689 160x9_testes_total |
SRR1106729 mixed whole adult body |
SRR060687 9_0-2h_embryos_total |
V116 male body |
SRR060671 9x160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................................................................CTCAGTGACGATACATCCAAGTGC.... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................TAAGAAATCTCGGGATATGATTGAGC.............................................................................................................................................................................. | 26 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................TAACCGAAGACCTTCTCGAAGAACT...................................... | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................GAAATCTCGGGATATGATTGAGCCCT........................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................TAGCTGAACAATTGACCGAAAACT................................................................................................................................................... | 24 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................TAAACTAAATCGAGACACAGCGGAGC.......................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................ACTGATTATGGAACC...................................................................... | 15 | 1 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .....................................................................................................................................................................................GATTACGTTACCTAAGACC................................................... | 19 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .GACTACTTGCGTAATGAGTT...................................................................................................................................................................................................................................... | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ................................................................................TAGGTGCACAATCGACCGA........................................................................................................................................................ | 19 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACTGATAAACTGATTACTCAAATAAGTTTTTGTACGGTTTCCGTTTTGGCCATTCTTTAGAGCCCTAAACTAACTCGGGAATCGACTTGTTAACTGGCTTTTGAACCGATTATCGTATTAGTTACGGTATGAAACATTTGATTTAGCTCTGTGTCGCCTCGTAGATTGACTAATACATTGGCTTATGCATTGGCTTCTGGAAGAGCTTCTTGAAACACTTCTCGAGTCACTGCTATGTAGGTTCACGAGCC
******************************************************************************************..(((((.........((((....((((................))))...)))).........)))............))******************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|
| ............................TATGTACGGTTTCTGTTTCGGC......................................................................................................................................................................................................... | 22 | 3 | 8 | 0.13 | 1 | 1 | 0 |
| .............................................................................................................................................................................................TAGGCTTCTGGAAGA............................................... | 15 | 1 | 11 | 0.09 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:4091360-4091610 - | dvi_13099 | TGACTATTTGACTAATGAGTTTATTCAAAAACATGCCAAAGGCAAAACCGGTAAGAAATCTCGGGATTTGATTGAGCCCTTAGCTGAACAATTGACCGAAAACTTGGCTAATAGCATAATCAATGCCATACTTTGTAAACTAAATCGAGACACAGCGGAGCATCTAACTGATTATGTAACCGAATACGTAACCGAAGACCTTCTCGAAGAACTTTGTGAAGAGCTCAGTGACGATACATCC---AAGTGCTCGG |
| droMoj3 | scaffold_6473:8648689-8648834 - | ----------------------------------------------------------------------------------------------------------GCTGACTTCATCATTAATGATATCATTGAGAATCTGATTGATGATACTAGTGAGCATTTAACCGCTTATGTCGCCGAATACATAACGGAAGATATTCTCGACGAGCTATCGGCA--GCATATTCAGAATACTTGAAGAAATCGAATGG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
Generated: 05/19/2015 at 05:55 PM