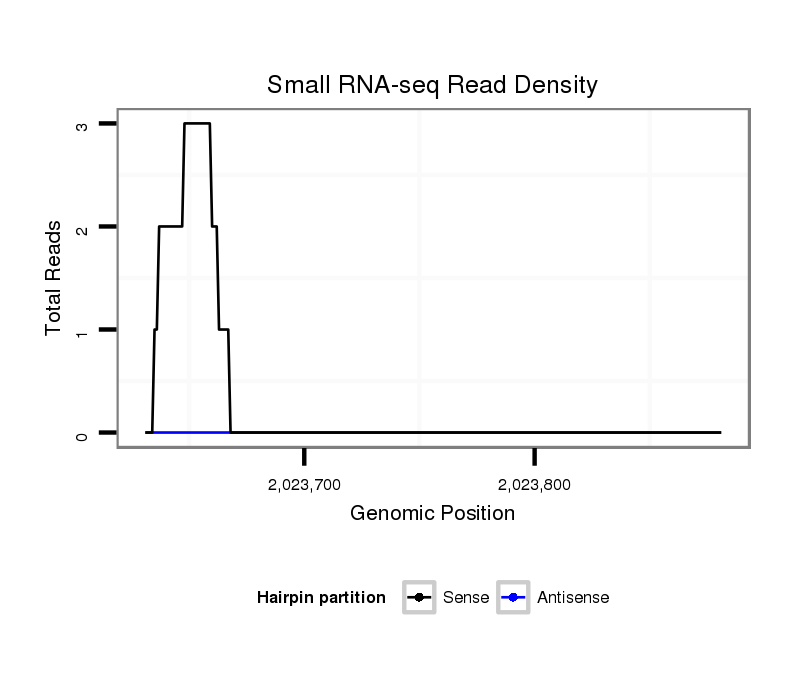
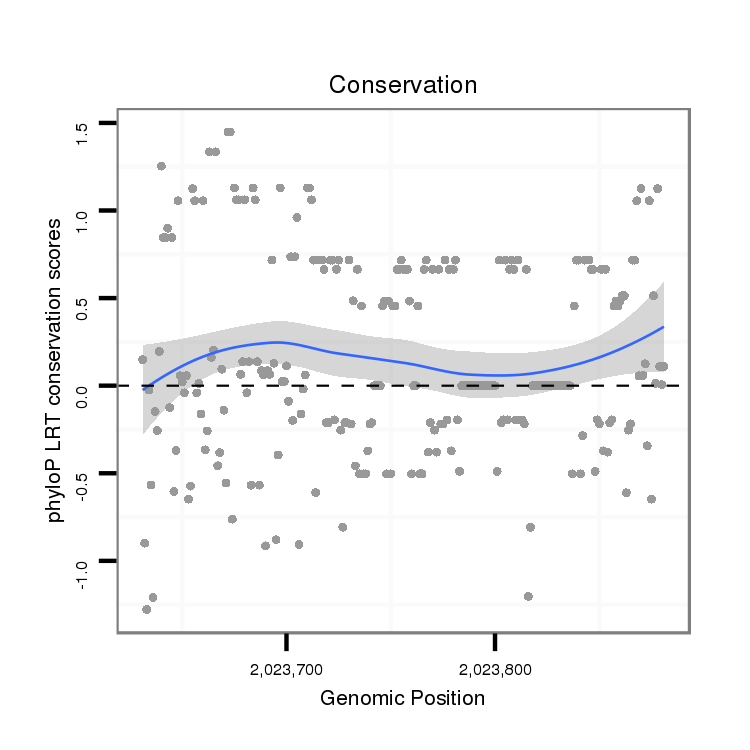
ID:dvi_12860 |
Coordinate:scaffold_12970:2023681-2023831 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_4025:2]; CDS [Dvir\GJ19140-cds]; intron [Dvir\GJ19140-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AAAAGTTAAGCGAAGGCGGCGGCGACGCTGATGGCGACACCAAATCGTTGGTGAGTTGACCAAACTTCTGCAAAAACAAAACACTTTGATTAGCATCTATGGTGGGCGCGATGCCCAATGGGCGTGGCAACAGTAGTTACATGAATCGGCAGCAGCTAAAATCGAAACCCAAACTGCAGCTGCTGCCACCGCCACCGCCACCGCCACGAATATTTCCGGTGCCATGGACCATGTGAACCATACGCTCAGCC **************************************************..(((((.....))))).(((.....((((....))))....)))....((((((..(((..((((...))))(((((.......))))).......((((((((((....................))))))))))..)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060663 160_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
V116 male body |
SRR060687 9_0-2h_embryos_total |
V053 head |
V047 embryo |
GSM1528803 follicle cells |
M027 male body |
SRR060668 160x9_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....GTTAAGCGAAGGCGGCGGCGACGCT.............................................................................................................................................................................................................................. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......TAAGCGAAGGCGGCGGCGACGCTGAT........................................................................................................................................................................................................................... | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................GGCGGCGACGCTGATGGCGA...................................................................................................................................................................................................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................CACCAAATCGATGGTCAGTTG................................................................................................................................................................................................. | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................CTCTGCAAGAACAAACCACT...................................................................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................TTGAATCGGCAGCAGCCAAAAA......................................................................................... | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................GGCCGCGAGGCTGATGGCGGC..................................................................................................................................................................................................................... | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...................................................TGAGTTGTCCATAATTCTGCA................................................................................................................................................................................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................TATTTCTGGTGGCACGGAC...................... | 19 | 3 | 14 | 0.14 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 |
| ......................................................GTTGACTTAACTTCTACAAAA................................................................................................................................................................................ | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .........GCGAAGGCCGCGGCGAAG................................................................................................................................................................................................................................ | 18 | 2 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................ATCCAATCGTTGGTGAGTA.................................................................................................................................................................................................. | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTTTCAATTCGCTTCCGCCGCCGCTGCGACTACCGCTGTGGTTTAGCAACCACTCAACTGGTTTGAAGACGTTTTTGTTTTGTGAAACTAATCGTAGATACCACCCGCGCTACGGGTTACCCGCACCGTTGTCATCAATGTACTTAGCCGTCGTCGATTTTAGCTTTGGGTTTGACGTCGACGACGGTGGCGGTGGCGGTGGCGGTGCTTATAAAGGCCACGGTACCTGGTACACTTGGTATGCGAGTCGG
**************************************************..(((((.....))))).(((.....((((....))))....)))....((((((..(((..((((...))))(((((.......))))).......((((((((((....................))))))))))..)))..)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060662 9x160_0-2h_embryos_total |
SRR060671 9x160_males_carcasses_total |
V047 embryo |
V116 male body |
SRR060687 9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
M047 female body |
SRR060672 9x160_females_carcasses_total |
M028 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................................................CCCGAAGCGTTGGCATCAA................................................................................................................. | 19 | 3 | 6 | 1.83 | 11 | 0 | 11 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...TCGATTCGCTTCCACCGCCACT.................................................................................................................................................................................................................................. | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................CTTATAAGGGCCACGGTTC......................... | 19 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................CCCGCAGCGTTGGCATCAA................................................................................................................. | 19 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................ACCTCTGAACTGGTTTGGAGAC..................................................................................................................................................................................... | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................GGGTACTTAAAAAGGCCACGGTAC......................... | 24 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................CCTTATAAAGGCCACGGTAA......................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................GCGCTTATGAAGGCCACGGTAA......................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................CTTATGAAGGCCACGGTAA......................... | 19 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................................GCTTATCACGGCCACGGTAT......................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................TTGTTTTTAAAGGCCACGGTA.......................... | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................GTTTTGGGTTTCTGAAACTAA................................................................................................................................................................ | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................................TAGATACCACCAGCATTAC.......................................................................................................................................... | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................................................................................................................GGTGGCGGTGGCGGTGGCGGTG............................................ | 22 | 0 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................................................................................CCTTATGAAGGTCACGGTAC......................... | 20 | 3 | 10 | 0.10 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................CGGTGCCGGTGGCGCTCGCGGTG............................................ | 23 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................CGGCACCTGGGACACTTC............. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................................................................................TGGCGGTGGCGGTGGCGGTA............................................ | 20 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................................CGGTGGCGGTGGCGGAGCG.......................................... | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:2023631-2023881 + | dvi_12860 | AAAAGTTA----AGCGAAGG------CGGCGGCGACGCTGATGGCGACAC---CAAATCGTTGGTGAGTTGACCAAACTTCTGCAAAAACAAAACACTTTGATTAGCATCTA-------TGGTGGGCGCGATGCCCAATGGGCGTGGCAACAGTAGTTACATGAATCGGCAGCAGCTAAAATCGAAACCCAAACTGCAGCTGCTGCCACCGCCACCGCCACCGCCACG-AATATTTCCGGTGC---------------------CATGGAC--------------------------------------------CATGTGAACCATACGCTCAGCC |
| droMoj3 | scaffold_6359:3231295-3231533 + | AGAAAATT----AGCGAAGCTGGCGCTGGCGGTGACGTTGATGGCGCCACAAGAAAATCGCTGGTGAGTCAAGTGAATTTCTGC------AAAACAGTTTGATTAGCACGTAGAGCTTATA--GAGTATAA----CAGTAGGCGTGGCAG--GCGGTTACGTGAATCGACAGC-----------------CAACTGCAGCTGCTGTG-------------------TGCAACGTTTCCGGTGTGTAACGAACCCATAGCCTCTTCATGGACCATG-------------------------------------------CCAAACCATATGT-TAGGC | |
| droGri2 | scaffold_14853:6976205-6976445 - | AAGCAG--AGCTAG------------TGAAGACAACGTCGGCGGCGAGGC---AAAGTCGTTGGTAAGTCGAGCAAAAATACG----ACTCAAACACTTTGGCTAACATGCG-------TGGCG----TGG-----------CGTGGC-------GTGGCAGGTTTTGACAAG-----------------CAGTTACAGTTATAGGG---------------------CAA-GTTTCCCAAGTCCTGCAAGTCTGCAGCAACTCCCCA-------CATTACCGGTTAGACAGTCAGTCGTCCCACGCCGCTGGACCATGTGAACTATACGCTTAGGC | |
| droWil2 | scf2_1100000004511:8734084-8734086 + | GCC---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | XL_group1a:948412-948427 + | AACAAATT----GGCGACGG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_26:1062263-1062278 + | AACAAATT----GGCGACGG----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droAna3 | scaffold_12929:2924437-2924493 - | AAGTCG----------------------------------ATGGAGCCGT---CAAGTCGTCGCCCAGCAGCACA-TCCTCTAAAGAAGCACAAC-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000391693:5344-5362 - | AGAACGAA----------------------------------GGAGCCGT---CAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000777097:98338-98392 - | GGAATCCAAGCCAG------------CGGCCGGCACCTCGTCGCCGAAGG---AAAC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCAGTTGG-TAAGT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
Generated: 05/16/2015 at 10:08 PM