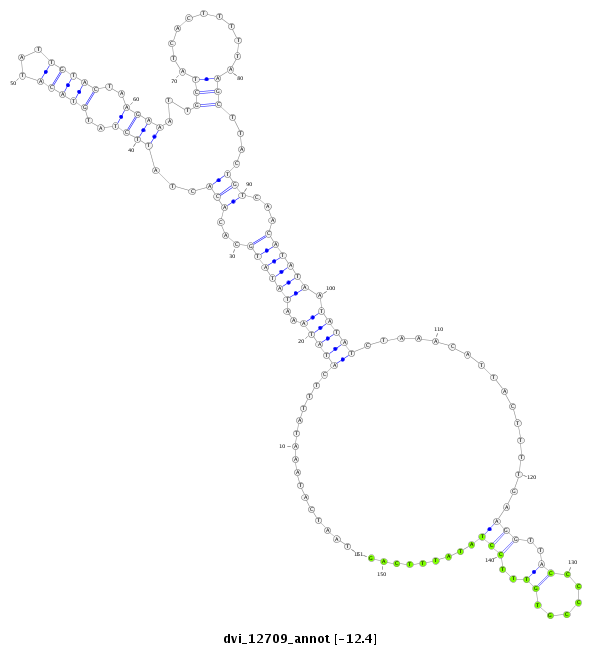



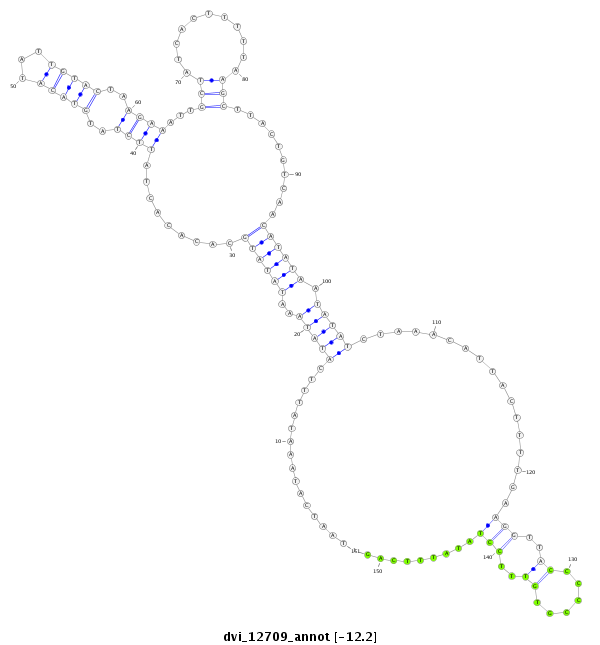
ID:dvi_12709 |
Coordinate:scaffold_12970:58867-59017 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -12.2 | -12.1 | -12.0 |
 |
 |
 |
CDS [Dvir\GJ19080-cds]; exon [dvir_GLEANR_3924:2]; intron [Dvir\GJ19080-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TGTTGTGAATTTAACGCAAACATAAATAGGAAGAGCAATATCAAACCAAATAATCATAAATATTTCATATAAATATATGCACACACTATTCTATGTACATATTGTACTAAGAAATTGCTATCACTTTTTAAGCTTACTGTCAACATATAATATATCTAAACATTACTTTTGAAGGTTACCCCCGTGTTTCCTATATTTCAGAATCGCCGGCAGCGTGTTCGAAGTAAGCCTGTTAAAAAGCGCGGCGAATT **************************************************................(((((..((((((...(((...((((..(((((...)))))..))))...(((...........)))....)))...)))))).))))).................(((..((......))..))).........************************************************** |
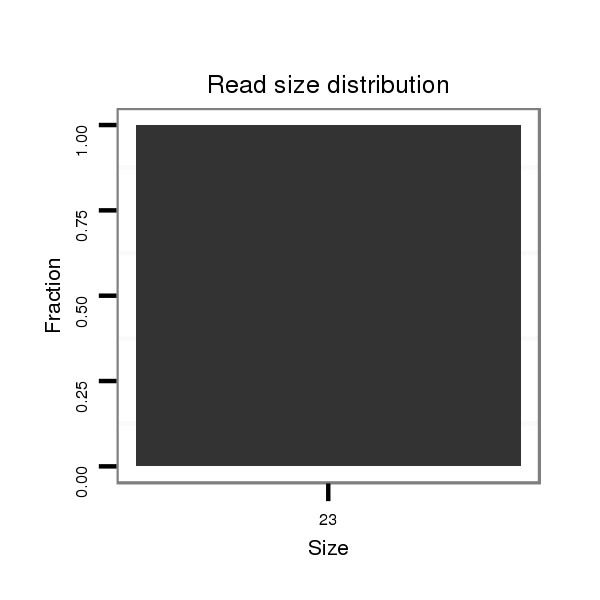
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060678 9x140_testes_total |
SRR060669 160x9_females_carcasses_total |
V053 head |
SRR060684 140x9_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060670 9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............GCATACATAAATAGGAAGA......................................................................................................................................................................................................................... | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................CCCCCGTGTTTCCTATATTTCAG.................................................. | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGATATCATTTTGTAAGCTTAC.................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCATTTTTAAAGCTTATT................................................................................................................. | 23 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................CCCGTGTTACCTAGATTT..................................................... | 18 | 2 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCACTTCATAAGCTTAAT................................................................................................................. | 23 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................GTTAAAAAGCACGGCGAT.. | 18 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCACTTCATAAGCTTAT.................................................................................................................. | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCACTATATAAGCTTAT.................................................................................................................. | 22 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............GCATACATAAATAGGAAGAT........................................................................................................................................................................................................................ | 20 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................TAACTTTTTCAGCTTACTGTCT............................................................................................................. | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCATTTTTCAAGCTTAT.................................................................................................................. | 22 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................TGCTATCATTTTTAAAGCTTAT.................................................................................................................. | 22 | 3 | 8 | 0.13 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................CTTTTTCCGCTTACTGTCA............................................................................................................. | 19 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................GTTAAAAAGCACGGCGATC. | 19 | 3 | 18 | 0.11 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ...................................................................................................................TGCTATCATTTTTAAAGCTGA................................................................................................................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................ACATCACTTTGTAAGCTTA................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ACAACACTTAAATTGCGTTTGTATTTATCCTTCTCGTTATAGTTTGGTTTATTAGTATTTATAAAGTATATTTATATACGTGTGTGATAAGATACATGTATAACATGATTCTTTAACGATAGTGAAAAATTCGAATGACAGTTGTATATTATATAGATTTGTAATGAAAACTTCCAATGGGGGCACAAAGGATATAAAGTCTTAGCGGCCGTCGCACAAGCTTCATTCGGACAATTTTTCGCGCCGCTTAA
**************************************************................(((((..((((((...(((...((((..(((((...)))))..))))...(((...........)))....)))...)))))).))))).................(((..((......))..))).........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060676 9xArg_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
M061 embryo |
SRR060659 Argentina_testes_total |
SRR060677 Argx9_ovaries_total |
V053 head |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
SRR060680 9xArg_testes_total |
V116 male body |
SRR060669 160x9_females_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................................................TCCAATGGGTGCACAATG............................................................. | 18 | 2 | 1 | 3.00 | 3 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................GAAAAATTCGAATGACAGTTGTATAT...................................................................................................... | 26 | 0 | 1 | 2.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................CGTCCAATGGGTGCACAATG............................................................. | 20 | 3 | 2 | 2.00 | 4 | 0 | 2 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..................................................................................TGTGATAAGATACATGTATAACATGATT............................................................................................................................................. | 28 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................AACGATAGTGAAAAATTCGAATGACAGT............................................................................................................. | 28 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................................CAATGGGGGCACAAAGAGT.......................................................... | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................CGGCCGTCGCACAAGCTTCA.......................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................TCCAAAGGGTGCACAATG............................................................. | 18 | 3 | 20 | 0.40 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 5 | 3 | 0 | 0 |
| ..........................................................................................................................................................................CGTCCAATGGGTGCACAA............................................................... | 18 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................CCGTCCAATGGGTGCACAA............................................................... | 19 | 3 | 18 | 0.28 | 5 | 0 | 1 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
| .............................................................................................................................AAAAACCGAATGGCAGTTGTA......................................................................................................... | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................CGTCCAATGGGTGCACAAT.............................................................. | 19 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................TCCAATAGGTGCACAATG............................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12970:58817-59067 + | dvi_12709 | TGTT-----------GTGAATTTAACGCAAA--CATAAATAGGAAGAGCAATATCAAACCAAATAATCATAAATATTTCATATAAATATATGCACACACTATTCTATGTACATATTGTACTAAGAAATTGCTATCACTTTTTAAGCTTACTGTCAACATATAATATATCTAAACATTACTTTTGAAGGTTACCCCCGTGT-----------TTCC-----TATATTTCAGAATCGCCGGCAGCGTGTTCGAAGTAAGCCTGTTAAAAAGCGCGGCGAATT |
| droMoj3 | scaffold_6498:2979588-2979773 - | TT-------------GTAGAATAAATACAAAACGATAAATTAACATAGAAATATTTAACTCA-----------------------------------------------------------------------------TTTAAGCGCCATGCCATCACATCAAAAATTGAAATACC-CTTCTACAATATAC---CGTATTCTAATTATACATTTTTTCGTTTGTTTTAGAACCGCCGACAGCGTATCCGAAACAAGCCGCTTAAGAAGCGTGGCGAAGT | |
| droGri2 | scaffold_15203:8952767-8952864 + | ATATTTAAAATATTTGTAAAATAT-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGAAATCCTTTCTCTTG-TTTAGAATCGGCGTCAACGGGTTCGCAGTAAGCCACTGAAGAAGCGTGGCGAAGT | |
| droWil2 | scf2_1100000004909:7662552-7662631 + | TA-------------T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CCACATCA-----------TTCTTATTATTAATTACAGAATCGTCGCATTCGTGTGCGCAGTAAGTCATCAAAGAAACGAGGGGATCT | |
| dp5 | XL_group1e:1252525-1252580 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACAGACGCGTCGCACTCGTGTGCGCAACAAGGCGGTCAAGAAGCGCGGCGAGGT | |
| droPer2 | scaffold_18:99290-99345 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TACAGACGCGTCGCACTCGTGTGCGCAACAAGGCGGTCAAGAAGCGCGGCGAGGT | |
| droAna3 | scaffold_13248:2831791-2831843 + | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGGCGCGCCGCACCCGGGTGAGGAACAAGCCGCCCAAGAAACGCGGCGAGGT | |
| droBip1 | scf7180000396429:176442-176494 - | C-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGACGCGCCGCACCCGGGTGAGGAACAAGCCGCCCAAGAAACGCGGCGAAGT | |
| droKik1 | scf7180000302230:595438-595468 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGCAAGCAGCCAAAGAAGCGCGGCGAGGT | |
| droFic1 | scf7180000454073:1360157-1360187 - | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGCAAGCCGCCCAAGAAACGCGGCGAGGT | |
| droEle1 | scf7180000491044:115924-115954 - | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCAAGCCGCCAAAGAAACGTGGCGAGGT | |
| droRho1 | scf7180000768457:28514-28544 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAGCAAGCCTCCGAAGAAACGTGGCGAGGT | |
| droBia1 | scf7180000302041:628813-628843 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAACAAGCCGCCGAAGAAGCGCGGCGAGGT | |
| droTak1 | scf7180000414562:555050-555084 - | G-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTCGGAGCAAGCCGCCCAAGAAGCGCGGCGAGGT | |
| droEug1 | scf7180000409008:28348-28382 + | G-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAGGGGCAAGACGCCAAAGAAACGCGGCGAGGT | |
| dm3 | chrX:13609815-13609845 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAATAAGCCTTCAAAGAAACGTGGCGAGGT | |
| droSim2 | x:12945568-12945602 + | G-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAAGAATAAGCCTACAAAGAAACGTGGCGAGGT | |
| droSec2 | scaffold_20:248171-248205 + | G-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTAAGAATAAGCCTACAAAGAAACGTGGCGAGGT | |
| droYak3 | X:7904068-7904098 + | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATAAGCCGCAGAAGAAACGTGGCGAGGT | |
| droEre2 | scaffold_4690:13273970-13274000 - | G---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATAAGCCGCAGAAGAAACGTGGCGAGGT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 08:32 PM