

ID:dvi_12375 |
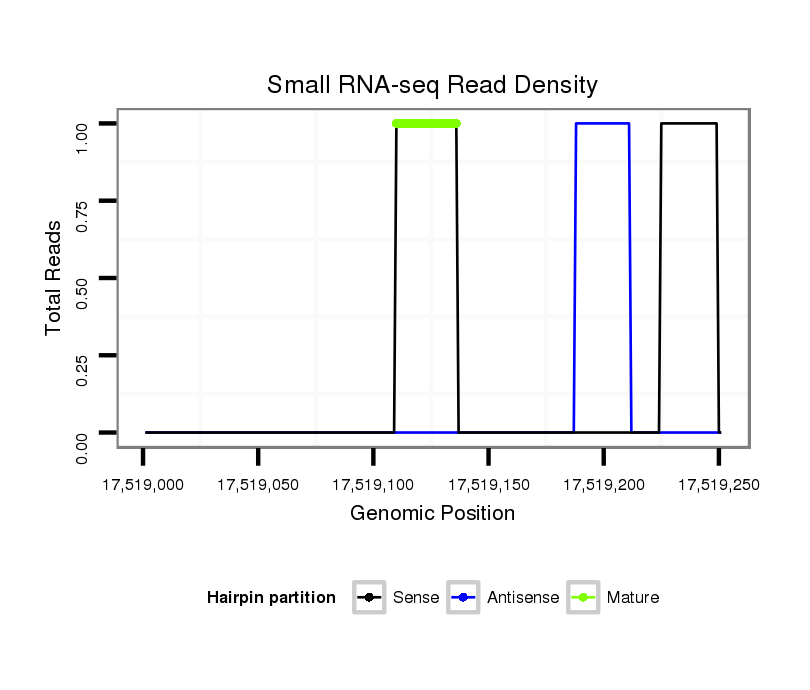
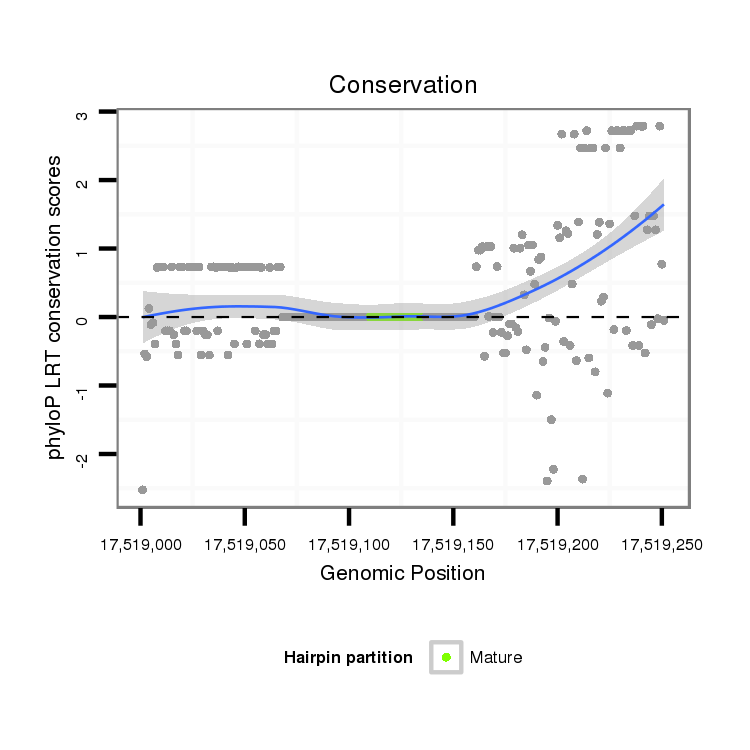
Coordinate:scaffold_12963:17519051-17519201 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -22.0 | -21.9 | -21.9 |
 |
 |
 |
exon [dvir_GLEANR_2290:2]; CDS [Dvir\GJ17720-cds]; intron [Dvir\GJ17720-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GTGGCACGTATAAAATCGTTGAAAATGAGCTTGATGGAAATCGGGCAAAACTTTAAACTTAGCCGAAAAATCTTTGAATTGCATTTTCTACCGTGGAATCTTGGATTCTGCGAAACCCCCATTTCCACCAAGGTGCAGTGGATTTTCCAAAGTGTGATAAACCTCTCAAGTGAAACCCTTTAACGTCAACTTTCATTACAGTCACACTCTCTGTTCGGCACTCGTATATGTTTTATTTGAACGATACAATC **************************************************................(((((((....((((((((((.....((((((....((.(((...))).)).....)))))).)))))))))))))))))......((((((............((((....))))............)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
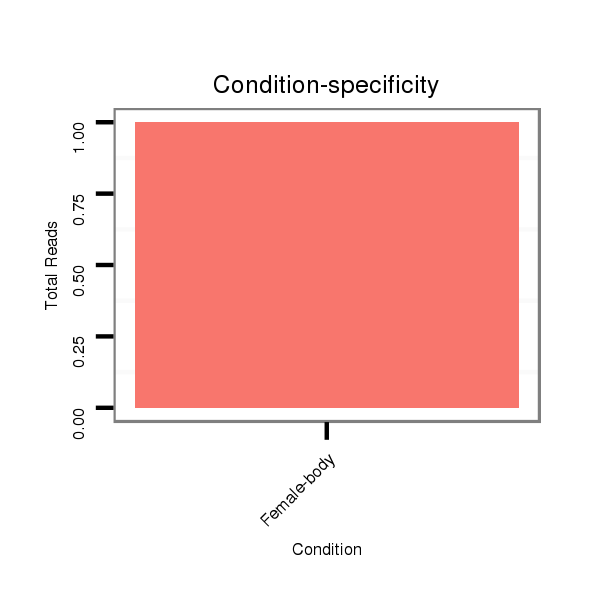
M047 female body |
SRR060681 Argx9_testes_total |
SRR1106730 embryo_16-30h |
SRR1106715 embryo_4-6h |
V047 embryo |
V053 head |
M028 head |
SRR060667 160_females_carcasses_total |
SRR1106712 embryo_0-2h |
SRR060656 9x160_ovaries_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060668 160x9_males_carcasses_total |
SRR1106728 larvae |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................................................................................TATATGTTTTATTTGAACGATACAA.. | 25 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................GCGAAACCCCCATTTCCACCAAGGTGC................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................CACACTCTCTGTTCGGCACTCGTATATGC.................... | 29 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................TTCCACCAAGTTGCGGTGG.............................................................................................................. | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................ATCTTTGAGTTGCATTTTCTTG................................................................................................................................................................ | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................TCCACCACGGTCCCGTGG.............................................................................................................. | 18 | 3 | 20 | 0.45 | 9 | 0 | 0 | 0 | 8 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..............................................................................................................................................................................................TTGCATTACAGTCGCATTCTCT....................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................TCCACCAAGGTCCCGTGG.............................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................CGAACCCCGCATCTCCACCA......................................................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................TCCACCAAGTTGCCGTGG.............................................................................................................. | 18 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................CTTTTACGTCAGCTTTCAT....................................................... | 19 | 2 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................TTGGATCTTGGATTTTGCGAAA........................................................................................................................................ | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................TTCCACCACGGTCCCGTGG.............................................................................................................. | 19 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................TTTTGTTTGAACGATAGA... | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................AATGAGTATGATGGAAAT.................................................................................................................................................................................................................. | 18 | 2 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..........................................................................................................................TTCCACCAAGTTCCCGTGG.............................................................................................................. | 19 | 3 | 17 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................TTCCACCAAGTTACAGGGG.............................................................................................................. | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........ATAAAATCGTTGAAAT.................................................................................................................................................................................................................................. | 16 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
CACCGTGCATATTTTAGCAACTTTTACTCGAACTACCTTTAGCCCGTTTTGAAATTTGAATCGGCTTTTTAGAAACTTAACGTAAAAGATGGCACCTTAGAACCTAAGACGCTTTGGGGGTAAAGGTGGTTCCACGTCACCTAAAAGGTTTCACACTATTTGGAGAGTTCACTTTGGGAAATTGCAGTTGAAAGTAATGTCAGTGTGAGAGACAAGCCGTGAGCATATACAAAATAAACTTGCTATGTTAG
**************************************************................(((((((....((((((((((.....((((((....((.(((...))).)).....)))))).)))))))))))))))))......((((((............((((....))))............)))))).************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060659 Argentina_testes_total |
SRR060666 160_males_carcasses_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|
| ...........................................................................................................................................................................................TTGAAAGTAATGTCAGTGTGAGAG........................................ | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................ATCTACTAAAAGGTTTCACACT.............................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 |
| ..................................................GAAATTTGAATCGGGTTTTT..................................................................................................................................................................................... | 20 | 1 | 5 | 0.40 | 2 | 0 | 2 | 0 | 0 |
| ...........TTTTCGCAACTTTTACTCGAGT.......................................................................................................................................................................................................................... | 22 | 3 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 |
| ...............................................................................................................CTTTGGTGGTCAAGGTCGTT........................................................................................................................ | 20 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 |
| ...............................................................................................................................................................TTGTAGAGTTAACTTTGGTAA....................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:17519001-17519251 + | dvi_12375 | GTGGCACGT-ATAAAATCGTTGAAAATGAGCTTGATGGAAATCGGGCAAAACTTTAAACTTAGCCGAAAAATCTTTGAATTGCATTTTCTACCGTGGAATCTTGGATTCTGCGAAACCCCCATTTCCACCAAGGTGCAGTGGATTTTCCAAAGTGTGATAAACCTCTCAAGTGAAACCCTTTAACGTCAACTTTC---------------ATTACAGTCACACTCTCTGTTCGGCACTCGTATATGTTTTATTTGAACGATACAATC |
| droMoj3 | scaffold_6500:22558325-22558390 + | TCAA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TATCC---------------ATTGCAGTCACGCTCTCAGTTCGTCATCCCTACATGTTCTATTTTAATGATACGATA | |
| droGri2 | scaffold_15252:14948902-14948960 + | TTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCACAGTCACACTCTCGGTGCGTCACTCGTATATGTTTTACTTTAACGACACAATG | |
| droWil2 | scf2_1100000004521:10140427-10140500 - | CCTTTA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTCAACTTCC---------------TTTCTAGTTACTCTTTCGGTGCGTCATTCGTATATGTTTTACTTCAATGACACAATG | |
| dp5 | XL_group1e:12473995-12474058 + | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTTT---------------GTTGCAGTAACTCTATCTGTGCGTCACCCTCACATGTTCTACTTTAACGATACTATA | |
| droPer2 | scaffold_81:47921-47983 + | CTTACA------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCGCTGATCACTCTCACCGTGCGGAGCCCGTACATGTTTTACTTCAACGATACAACA | |
| droAna3 | scaffold_12916:12998446-12998543 - | ATAA-------------------------------------------------------------------------------------------------------------------------------------------------------------ACCTATTAT-TATAAACCTCTAATG---ACTC--------ATTTTTAATCCTCTAGTGACCCTGTCTGTGCGCCACTCGTACATGTTCTATTTCAATGACACAATG | |
| droBip1 | scf7180000396580:520079-520135 + | AT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTAGTCACTCTATCTGTGCGCCACTCGTACATGTTCTACTTCAATGATACAATG | |
| droKik1 | scf7180000301476:496699-496783 + | TTAAGT----------------------------------------------------------------------------------------------------------------------------------------------------------------------AACCCTTTTCAACAT-ATTTTCT---------------ATACTAGTCACCCTGTCGGTGCGCCACTCGTACATGTTCTACTTCAACGACACAATG | |
| droFic1 | scf7180000454015:280063-280152 + | AA----------------------------------------------------------------------------------------------------------------------------------------------------------------CCTTTTAA-------------ACA---AATTTATTTACCACCTGTAA-CCCACAGTCGTTCTGTCCGTCCGTCACTCGTACATGTTCTACTTCAACGACACGATG | |
| droEle1 | scf7180000491028:2720439-2720493 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACAGTCGTTTTGTCGGTTCGTCACTCATACATGTTCTATTTCAACGACACGATG | |
| droRho1 | scf7180000757856:793-875 + | TTTACAAGTCATGGGAAACTTAGAAATAACTAACATGAAAATGGGTCAAAAATTTGACCAACGATAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATGAAAATTTTGAAT | |
| droBia1 | scf7180000302422:582775-582828 - | A---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGTCGTTTTGTCTGTCCGCCACTCATATATGTTCTACTTTAACGACACGATG | |
| droTak1 | scf7180000415865:201224-201292 - | TTAACG---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AATTTC---------------TCTACAGTTGTATTGTCTGTACGTCACTCTTACATGTTCTATTTCAATGACACGATG | |
| droEug1 | scf7180000409122:785167-785230 + | AA--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTTAT---------------TCCACAGTCGTTCTCTCCGTCCGTCACTCGTATATGTTTTACTTCAACGACACAATG | |
| dm3 | chr2L:12399204-12399255 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTTGTTTTGTCCGTTCGGCATACATATATGTTTTATTTCAATGACACGATG | |
| droSim2 | 2l:12055758-12055809 + | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTTGTTTTGTCCGTTCGGCATACGTATATGTTTTATTTCAATGACACGATG | |
| droSec2 | scaffold_16:589951-589992 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCGTTCGGCATACGTATATGTTTTATTTCAATGACACGATG | |
| droYak3 | 2L:8835157-8835198 + | ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TCCGTTCGGCATACGTATATGTTTTATTTCAACGACACGATG | |
| droEre2 | scaffold_4929:13017115-13017166 - | -----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGTTGTTTTGTCCGTTCGGCATACTTATATGTTTTATTTCAACGACACGATG |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 11:23 PM