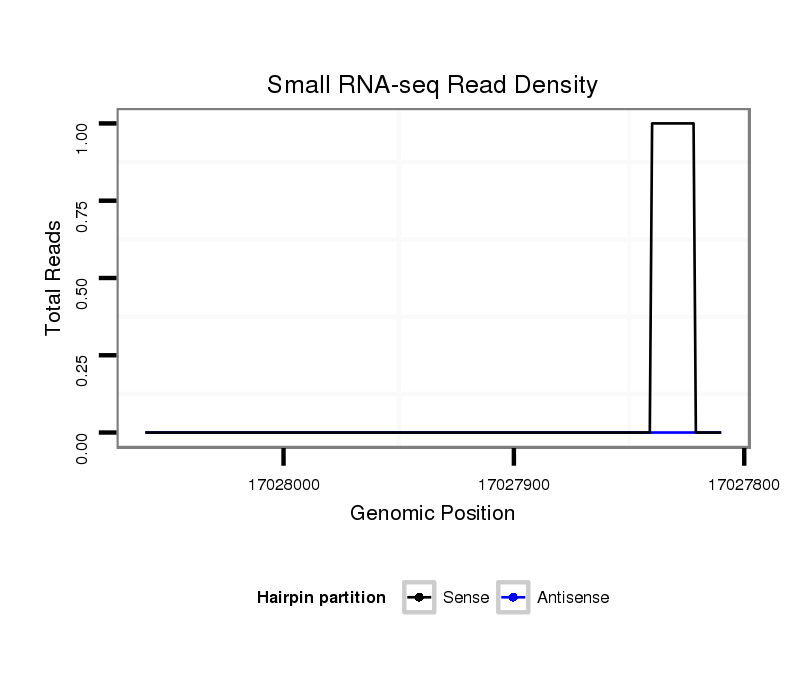
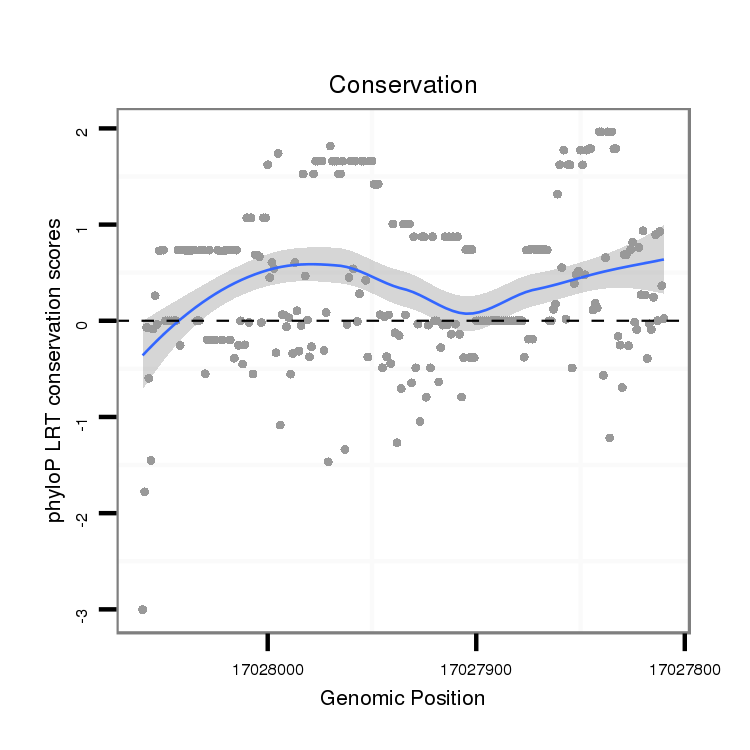
ID:dvi_12318 |
Coordinate:scaffold_12963:17027860-17028010 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_566:5]; CDS [Dvir\GJ20209-cds]; intron [Dvir\GJ20209-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TAATGTCCGAAAGAGTATTTAGAGTTCGTTGCAGTAACCCTTAACTTGCCACAGATGTTAAAATATGATCAGAATGTGACTTCTATAGTTTATAGCTGACATAGCAACAATCGGGCCTACATTTTAGAAGACCTTAGTCTAGTCATATGTCTGCCAAGAGTATTTCCTGTTCGTTGTAATATCCCGTGTAACTTCCCACAGGTGCCGCATAGCTGGCAGAAGCTGTGGCCCGGTCCGAGCAGCAACAGCGC **************************************************...(((((((....((((.(((......((((((((.(((.......))))))).........(((..((((......((((....))))......))))..)))..)))).....))).))))..)))))))..(((........)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
M047 female body |
V053 head |
M061 embryo |
M027 male body |
V116 male body |
V047 embryo |
SRR060670 9_testes_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060676 9xArg_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060663 160_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060683 160_testes_total |
SRR060688 160_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCTGTC.................................. | 21 | 3 | 9 | 12.11 | 109 | 45 | 12 | 15 | 9 | 5 | 8 | 6 | 0 | 2 | 0 | 1 | 3 | 1 | 1 | 1 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGTGCTGCATGGCTGTC.................................. | 20 | 3 | 15 | 9.80 | 147 | 52 | 23 | 16 | 31 | 3 | 3 | 6 | 0 | 7 | 0 | 2 | 0 | 1 | 1 | 0 | 1 | 1 |
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCT..................................... | 18 | 2 | 20 | 5.95 | 119 | 79 | 23 | 0 | 3 | 10 | 0 | 1 | 0 | 2 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCTGG................................... | 20 | 2 | 2 | 5.00 | 10 | 8 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCTG.................................... | 19 | 2 | 5 | 2.40 | 12 | 9 | 1 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGTGCTGCATGGCTGG................................... | 19 | 2 | 2 | 2.00 | 4 | 2 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCTGGCG................................. | 22 | 3 | 2 | 1.00 | 2 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................AGCTGTGGCCCGGTCCGAG............ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................ACACAGGTGCTGCATGGCTGG................................... | 21 | 3 | 3 | 0.67 | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................TGTCCTGTTCGTTGTAAAA...................................................................... | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................ACACAGGTGCTGCATGGCTG.................................... | 20 | 3 | 12 | 0.42 | 5 | 3 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................ACACAGGTGCCGCATGGCT..................................... | 19 | 2 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................GCACAGGTGCTGCATGGCTGG................................... | 21 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................CACAGGTGCTGCATGGCTGAC.................................. | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGTGCTGCATGGCTGGCG................................. | 21 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CAGGTGCTGCATGGCTGGCGG................................ | 21 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGTGCTGCATGGCTGAC.................................. | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................ACAGGTGCTGCATGGCTG.................................... | 18 | 2 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................CAGGTGCTGCATGGCTGG................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................CGCACAGGTGCTGCATGGCT..................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
|
ATTACAGGCTTTCTCATAAATCTCAAGCAACGTCATTGGGAATTGAACGGTGTCTACAATTTTATACTAGTCTTACACTGAAGATATCAAATATCGACTGTATCGTTGTTAGCCCGGATGTAAAATCTTCTGGAATCAGATCAGTATACAGACGGTTCTCATAAAGGACAAGCAACATTATAGGGCACATTGAAGGGTGTCCACGGCGTATCGACCGTCTTCGACACCGGGCCAGGCTCGTCGTTGTCGCG
**************************************************...(((((((....((((.(((......((((((((.(((.......))))))).........(((..((((......((((....))))......))))..)))..)))).....))).))))..)))))))..(((........)))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M027 male body |
M047 female body |
SRR060666 160_males_carcasses_total |
SRR1106727 larvae |
V116 male body |
SRR060673 9_ovaries_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................................................................CTGGATGCCAAATCTTCTGG...................................................................................................................... | 20 | 3 | 3 | 0.67 | 2 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................CCAGGCTCCTCTTTGTCG.. | 18 | 2 | 4 | 0.25 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................GCCAGGCTCCTCTTTGT.... | 17 | 2 | 5 | 0.20 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| .....................................................................................................................................................AGACGGTTCTGGTAAGGGA................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 1 | 0 | 0 | 1 | 0 |
| ............................................................................................................................................................TCTCTGAAGGGACAAGCAA............................................................................ | 19 | 3 | 15 | 0.07 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................................................................................................................CCCAGTGAAGTGTGTCCA................................................ | 18 | 3 | 19 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................................................CCAGGCTCCTCTTTGTCGA. | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................................................................................................................................................................................................................ACCGCGCCAGGCTCCTCTT....... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:17027810-17028060 - | dvi_12318 | TAATGTCCGAAAGAGTATTTAGAGTTCGTTGCAGTAACCCTTAACTTGCCACAGATGTTA-----------AAATA------TGATCAGAATGTGACTTCTATAGTTTATAGCTGACATAGCAACAATCGGGCCTA------------CATTTTAGAAGACCTTAGTCTAGTCATATGTCTGCCAAGAGTATTTCCTGTTCGTTGTAATATCCCGTGTAACTTCCCACAGGTGCCGCATAGCTGGCAGAAGCTGTGGCCCGGTCCGAGCAGCAACAGCGC |
| droMoj3 | scaffold_6500:22045425-22045484 - | CTTCA--C--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GCAGGTGCCCCACAGCTGGCAGAAGCTGTGGCCCGGTCCGAGCAGTAGCAGCGC | |
| droGri2 | scaffold_15252:14461759-14461846 - | AATATCCC---------------------------------------------------------------ATAACCATAATTTTCCCCTTCT-----------CCAT----------------------------------------------------------------------------------------------------------------------GCAGGTGCCGCACAGCTGGGCGAAATTGTGGCCAGGGCCAAGCAGCAAAAGCGT | |
| droWil2 | scf2_1100000004585:1440003-1440087 + | TTAGTTTA----------------------------------------ATATATATGTTAATGCCTGTGCCAAATT------TGATAAGGATCGGGTAGCTATATCATATAGCTCCCATAGGAACGATCGG----------------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 4_group3:10419037-10419074 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTGAAGCTCTGGCCAGGTCCCAGCAGCAACAGTGC | |
| droPer2 | scaffold_8:1632165-1632202 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGCTGAAGCTCTGGCCAGGTCCCAGCAGCAACAGTGC | |
| droAna3 | scaffold_12916:2934115-2934167 + | CA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATTCCTGCCAGCTGGCTGAAGCTTTGGTGCGGTCCTGGCAGCTCCGGTGC | |
| droBip1 | scf7180000396737:1826869-1826921 - | CA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATTCCTGCCAGCTGGCTGAAACTTTGGTGCGGTCCTGGTAGTTCCAGTGC | |
| droKik1 | scf7180000301431:7875-7933 - | GTTA-------------------------------------------------------------------AAATT------TTATGAAAATCGGACGACTATATCTTATAGCTGCCATAAGAACGATCGGG---------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droFic1 | scf7180000454108:306047-306143 + | GATTTTTGGAA------TATAGAGTT--TTCTAACGACTCTTGAATA---ACAA---ATA-----------AAATT------TCATCAAAATCGGACGACTATATCATATAGCTGTCATATGAACAAT-------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEle1 | scf7180000487999:1132-1211 + | GTTA-------------------------------------------------------------------AAATT------TCATCAAAATCGGACGACTATAGCATATAGCTCCAAAAGGAACAATCGGAAAAA------------TAATTGAAAAA-------------------------------------------------------------------------------------------------------------------AAATTA | |
| droRho1 | scf7180000772546:935-1017 + | CGCAGATC---------------------------------------------------------------AAGTTTGAAAATCGCCCCGATCGGATGTCTATATCCTATAGCTCCCATAGGAACAATCGGATATA------------T-------------------------------------------------------------------------------------------------------------------CTTGCAA-------GA | |
| droBia1 | scf7180000297051:12754-12873 - | AA-----------------------------------------------------------------------ATG------TTATCAAAATCGGACAACTATATTATATAGCTGCCATAGGAACGATCGGAAAATTAGTGGTTAAATAATACGAAAATATTATATCTT--TGGTGTTTTTTA-----------------------------ACATATAACTTC----------------------------------------------------CTAC | |
| droTak1 | scf7180000414220:24420-24528 - | GG----------------------------------------------------------------------------------------ACTTGACAACTATATCTTATAGCTCCCATAGGAAGGATCGGAAAAA------------AATGTAAAAAAACTCTAGCCT--CGGTGTTTTTTCAAATAT---------------------------------------------------------------------TACCTTTTACTCTTGGCAATGT | |
| droEug1 | scf7180000409066:89068-89119 + | A------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GATCCCAGCCAGTTGGTTGAAGCTATGGAGCGGACCTGGCAGTAACAGTGC | |
| dm3 | chr2L:17964400-17964454 - | CAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATTCCTGCCAGTTGGCTTAAACTATGGCCTGGACCTGGCAGCAACAGTGC | |
| droSim2 | 2l:17510770-17510824 - | CAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATTCCTGCCAGTTGGCTTAAACTGTGGCCTGGACCTGGCAGCAACAGTGC | |
| droSec2 | scaffold_7:1599319-1599373 - | CAT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGATTCCTGCCAGTTGGCTTAAACTATGGCCTGGACCTGGCAGCCACAGTGC | |
| droYak3 | 2R:3131804-3131865 + | TGTAATTC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAGATTCCTGCCAGTTGGCTTAAGCTGTGGTCTGGCCCTGGCAGCAACAGTGC | |
| droEre2 | scaffold_4845:6302022-6302083 + | TGTAATTC--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTAGATTCCTGCCAGTTGGCTAAAACTGTGGCCTGGACCTGGCAGCAACAGTGC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 10:01 PM