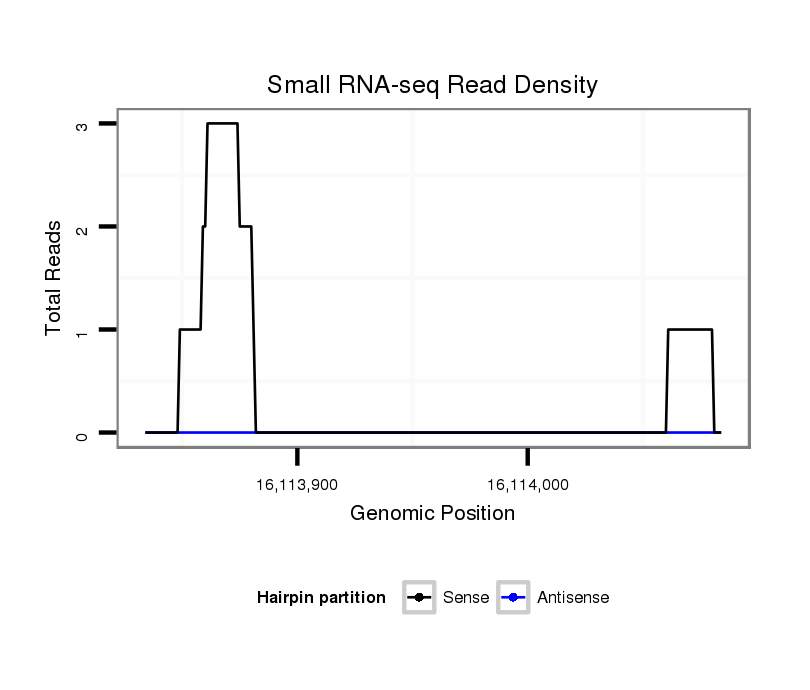
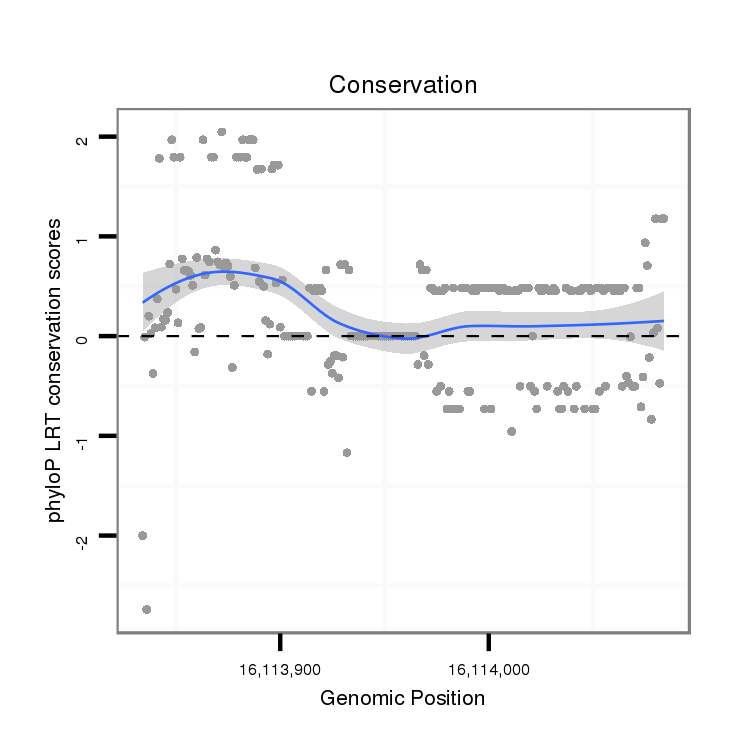
ID:dvi_12196 |
Coordinate:scaffold_12963:16113884-16114034 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_2243:1]; CDS [Dvir\GJ17668-cds]; intron [Dvir\GJ17668-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- TAATAATTTAATTCTGTTCAGCGCCCTCATTTGGCTGCTCGTCGCGCCAGGTAAGAGTTCAGGTGTTTCGGTGCCCCAAAATGTGTCAGAACCGTTATTCGATATTCGAGTGATATCTCAAAAGTCAGATATTAGCCTAAGTCCGAGTGTCCAAAATTGTGATACAAGATTGATGCGCCCGTATTAAAAGTGGATCGTGTCGGATCGCTGGAATTCGATTTGTTAAGCACCAACAGCAAAGTCGGAACGAA **************************************************......(((((..((...((((((..(((..(((((((.((.........((((((((..((((((((........)))))))).........)))))))).....)).)))))))...)))..))).)))...))....)))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
M027 male body |
SRR060659 Argentina_testes_total |
SRR060680 9xArg_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060657 140_testes_total |
SRR060666 160_males_carcasses_total |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............GTTCAGCGCCCTCATTTGGCTGCTCG.................................................................................................................................................................................................................. | 26 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................GAGATACAAGATTGATGCGCC........................................................................ | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................................CACCAACAGCAAAGTCGGAA.... | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................CTCATTTGGCTGCTCGTCGCGCC........................................................................................................................................................................................................... | 23 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................CATTTGGCTGCTCGTCGCGC............................................................................................................................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................TGTCGCGCCAGGTAAGAG.................................................................................................................................................................................................. | 18 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .................................GCTGCTCGTCGCGCCAGC........................................................................................................................................................................................................ | 18 | 1 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................AAAGATCTGGTGTTTCGGT................................................................................................................................................................................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 |
| ..........................................CGCGCCAGGGGAGAGTTCT.............................................................................................................................................................................................. | 19 | 3 | 14 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................TTGGCTCATCGTCCCGCCA.......................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................................................AGAGGTGTCAGAACCGTT........................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
ATTATTAAATTAAGACAAGTCGCGGGAGTAAACCGACGAGCAGCGCGGTCCATTCTCAAGTCCACAAAGCCACGGGGTTTTACACAGTCTTGGCAATAAGCTATAAGCTCACTATAGAGTTTTCAGTCTATAATCGGATTCAGGCTCACAGGTTTTAACACTATGTTCTAACTACGCGGGCATAATTTTCACCTAGCACAGCCTAGCGACCTTAAGCTAAACAATTCGTGGTTGTCGTTTCAGCCTTGCTT
**************************************************......(((((..((...((((((..(((..(((((((.((.........((((((((..((((((((........)))))))).........)))))))).....)).)))))))...)))..))).)))...))....)))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060671 9x160_males_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060669 160x9_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060672 9x160_females_carcasses_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060679 140x9_testes_total |
V116 male body |
SRR060685 9xArg_0-2h_embryos_total |
M027 male body |
SRR060667 160_females_carcasses_total |
V053 head |
SRR060664 9_males_carcasses_total |
SRR060676 9xArg_ovaries_total |
SRR060674 9x140_ovaries_total |
V047 embryo |
M047 female body |
SRR060665 9_females_carcasses_total |
SRR060675 140x9_ovaries_total |
SRR060681 Argx9_testes_total |
SRR060662 9x160_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .................................................................AAACCCACGGTTTTTTACACA..................................................................................................................................................................... | 21 | 3 | 16 | 19.81 | 317 | 69 | 43 | 33 | 29 | 30 | 25 | 18 | 15 | 11 | 9 | 6 | 6 | 5 | 5 | 0 | 3 | 3 | 1 | 2 | 1 | 1 | 1 | 1 | 0 |
| .................................................................................................................................................................................................................................TCGTGGTTCTCGCTACAGCCT..... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................................TGAAGCTAGAGAATTCGTG..................... | 19 | 3 | 20 | 0.10 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................................................................................................................................TACAGACCTCAAGCTAAACAA........................... | 21 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................AGTCGCGAGAATAAACCAAC...................................................................................................................................................................................................................... | 20 | 3 | 13 | 0.08 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................AGTCTTGGCAGTAAGGTGT................................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:16113834-16114084 + | dvi_12196 | TA----ATAATTTAATTCTGTTCAGCGCCCTCATTTGGCTGCTCGTCGCGCCAGGTAAGA------------------------------------------------------GTTC-AGGTGTTTCGGTGCCCCAAAATGTGTCAG---------------------------------------------------------------------AACCGTTATTCGATATTCGAGTGATATCTCAAAAGTCAGATATTAGCCT-AAGTCCGAGTGTCCAAAATTGTGATAC-AAGATTGATGCGCCCGTATTAAAAGTGGATCGTGTCGGATCGCTGGAAT--TCGATTTGTTAAGCACCAACAGCAAAGTCGGAACGAA |
| droMoj3 | scaffold_6500:5575715-5575977 + | TT----TTCCCTTAACTTTGTACGGCGCCTTTTTTCTGCTGCTCGTCGCGCCAGGTAAGAAAACCCCCCCAAAAAAAAAAAAAAAAAAACAAAAAATCACAAAATCCAAAATGTGTGTTGAGTGTCA------------AAGTGTCTG---------------------------------------------------------------------GGTCGATATCC--------------------------------CAGCCCAAAGACTGATATTAACAAAAAGTGATAAAAATATTGATGCGGCCGCATTAG-TTTGGACCGTGAATAAACGACGGACTACTCTCTATGCTAAGCACTATAAATAATAT--TGATAAA | |
| droGri2 | scaffold_15252:13543887-13544045 + | AA----ATAGTATGAAGTTGGCCAATGCTTTTATTTTGCTGCTCGTCTTGCCAGGTAAGAA--------------------------------------------------AGAGTTC-AAGTGTCT--------------------GTCCGTCCGTCCGTCCGTCCCACAAACGGCAGGACTCCCAAGTGTAGTGTGATAAAAAATAAAGCGCCCCGATTAATGTGC--------------------------------CAGTCC--------------------------------------------------------------------------------------------------------------------- | |
| dp5 | Unknown_singleton_2387:670-724 + | AA----GTCACATCTTTTTGGCCAGCTCTCTGCTGTTGCTACTCGCGTTGCCAGGTAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droPer2 | scaffold_1:9383508-9383566 + | CAAAAGTCCACATCTTTTTGGCCAGCTCTCTGCTGTTGCTACTCGCGTTGCCAGGTAAG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| droKik1 | scf7180000302405:466058-466087 - | AT----TT--------G----------------------------------------------------------------------------------------------------TAGGGTATTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ACG----GATCAAGACGAA | |
| droFic1 | scf7180000454051:9852-9922 - | AA----GTCACATCGTTTTGGCCAGCTGTTCGCTGTTGCTAATCGCCCTGCCAGGTAAAA---------------------------------------------------AGCATTT-GGGTATTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000780102:166416-166485 - | AA----GTCACATCGTTTTGGCCAGCTCTTTGCTGTTGCTAATCGCCCTGCCAGGTAAA-----------------------------------------------------GCATTTTGGGTATTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBia1 | scf7180000297014:5491-5509 + | AG----C--------------------------------AAATGACCT----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GGACAAA | |
| droTak1 | scf7180000415629:43474-43536 + | T------------CGTTTTGGCCAGCTCTCTGCTGTTGCTAATCGCCCTGCCAGGTAAAA---------------------------------------------------AGCATTT-GGGTATTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000409461:362558-362628 + | TA----GCCCCATCGTTTTGGCCAGCTCTTTGCTGTTGCTAATCGCCCTGCCAGGTAAAA---------------------------------------------------AGCATTT-GGGTATTT---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_3:3842271-3842326 + | AA----GTCACATCGTTTTGGCCAGCTCTTTGCTGTTGCTAATCGCCCTGCCAGGTAAAA----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droYak3 | 2L:10985125-10985174 + | TA----A-----------TGGCCAGCTCTTTGCTGTTGCTAATCGCCCTGCCAGGTAAAAT--------------------------------------------------TACA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEre2 | scaffold_4929:17256443-17256505 + | T------------CGTTTTGGCCAGCTCTTTGCTGTTGCTAATCGCCCTGCCAGGTAAAAAAAGA----------------------------------------------AGCATTT-TGG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 05:25 PM