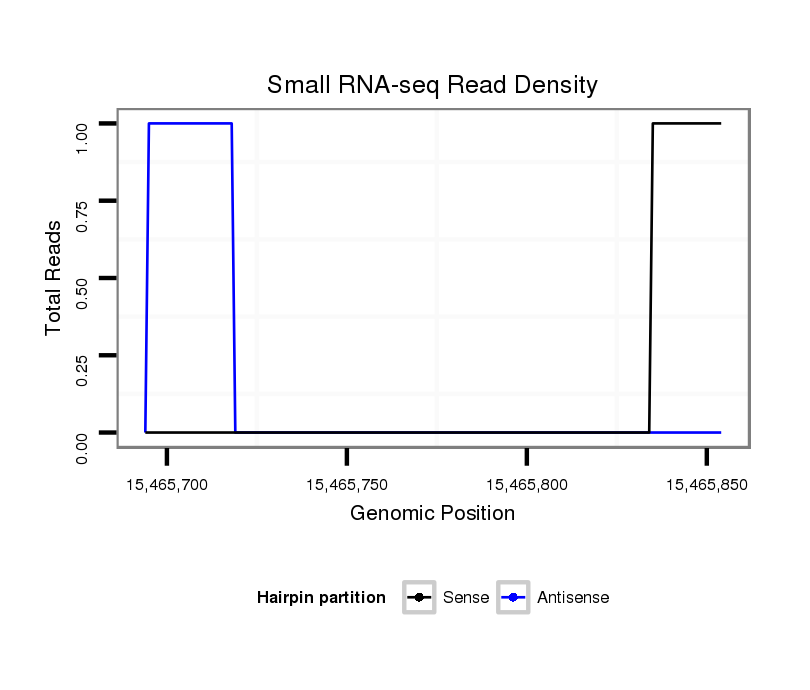
ID:dvi_12051 |
Coordinate:scaffold_12963:15465744-15465804 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
CDS [Dvir\GJ17614-cds]; CDS [Dvir\GJ17614-cds]; exon [dvir_GLEANR_2194:2]; exon [dvir_GLEANR_2194:3]; intron [Dvir\GJ17614-in]
No Repeatable elements found
| ##################################################-------------------------------------------------------------################################################## ACAAAGAGTCGGTTCAGTTTGATATACTGGATGCCACCGGGCAGCTGCACGTGAGCTGGATTGAGAGAATCTTGAGAGGATTCATAATGAAATCTAACTGAACTCTTGCAGGAACTAGATGGCGTTACACTGGAGTCCAATATACGCTGGGCCGACGCATT **************************************************...((.((((((....(((((((...))))))).......)))))).))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060662 9x160_0-2h_embryos_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
M047 female body |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
GSM1528803 follicle cells |
V053 head |
M028 head |
SRR060659 Argentina_testes_total |
SRR060685 9xArg_0-2h_embryos_total |
M027 male body |
SRR060689 160x9_testes_total |
V047 embryo |
SRR060680 9xArg_testes_total |
M061 embryo |
SRR060676 9xArg_ovaries_total |
SRR060683 160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ..............................................GCACGCGAGCTGGGTTCAGA............................................................................................... | 20 | 3 | 13 | 4.23 | 55 | 9 | 13 | 4 | 17 | 0 | 5 | 5 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................CACGCGAGCTGGGTTCAGA............................................................................................... | 19 | 3 | 18 | 2.61 | 47 | 8 | 2 | 10 | 11 | 0 | 7 | 7 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGCGGCACGCGAGCTGGGTT................................................................................................... | 20 | 3 | 5 | 2.40 | 12 | 3 | 4 | 1 | 1 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGCGGCACGCGAGCTGGGT.................................................................................................... | 19 | 3 | 20 | 1.40 | 28 | 9 | 2 | 4 | 1 | 0 | 5 | 6 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................ATACGCTGGGCCGACGCATT | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGCGGCACGTGAGCTGGGTTCAG................................................................................................ | 23 | 3 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................GCGGCACGCGAGCTGGGTT................................................................................................... | 19 | 3 | 11 | 0.73 | 8 | 4 | 2 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................CGGGGAGCTGTACGTAAGCT........................................................................................................ | 20 | 3 | 20 | 0.70 | 14 | 0 | 0 | 0 | 0 | 9 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................................AGAGACTTGAGAGGATT............................................................................... | 17 | 2 | 20 | 0.65 | 13 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 13 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................CAGGAACTTGATGCCGTTCC................................. | 20 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................GGGGAGCTGCACGTAAGCT........................................................................................................ | 19 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................CGTGAGCTGGGTTTAGGGAA............................................................................................ | 20 | 3 | 6 | 0.50 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................CTGCACGTGAGCTGGGTTT.................................................................................................. | 19 | 2 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................ACGTGAGCTGGATTTAGA............................................................................................... | 18 | 1 | 8 | 0.25 | 2 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................................................GACGATTCGTAATGAAAT.................................................................... | 18 | 2 | 9 | 0.22 | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................CGATAGCTGGTTTGAGAG.............................................................................................. | 18 | 3 | 20 | 0.20 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 1 |
| ...........................................................................................................................................AGAAACGCTGGGCGGACGC... | 19 | 3 | 12 | 0.17 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................CGTGAGCTGGGTTGAGACA............................................................................................. | 19 | 2 | 7 | 0.14 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................CGGCACGCGAGCTGGATTCA................................................................................................. | 20 | 3 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................TGCACGTGAGCTGGGATTAG................................................................................................ | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................CGTGAGCTGGGTTGAG................................................................................................ | 16 | 1 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................ACCGGGCAGCTGCATGCG............................................................................................................ | 18 | 2 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................AGCGGTACGTGAGCTGGGTT................................................................................................... | 20 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................ACTGACCTCTTGCAGGAAATT............................................ | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
TGTTTCTCAGCCAAGTCAAACTATATGACCTACGGTGGCCCGTCGACGTGCACTCGACCTAACTCTCTTAGAACTCTCCTAAGTATTACTTTAGATTGACTTGAGAACGTCCTTGATCTACCGCAATGTGACCTCAGGTTATATGCGACCCGGCTGCGTAA
**************************************************...((.((((((....(((((((...))))))).......)))))).))............************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|
| .GTTTCTCAGCCAAGTCAAACTATA........................................................................................................................................ | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 |
| ..........................................TCGCCGTGCGCTCGACC...................................................................................................... | 17 | 2 | 19 | 0.05 | 1 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:15465694-15465854 + | dvi_12051 | ACAAAGAGTCGGTTCAGTTTGATATACTGGATGCCACCGGGCAGCTGCACGTGAGCTGG-----ATTG-AGA------------------GAATC----TTG---AGAGGATTCATAATGAAA-------TCTA-AC----TGAACT---------CTTGCAGGAACTAGATGGCGTTACACTGGAGTCCAATATACGCTGGGCCGACGCATT |
| droMoj3 | scaffold_6500:18451902-18452063 + | ACAAGGAGTCGGTACAGTTTGACATCCTGGATGCCCACGGGCAGCTGCATGTGAGTGCTG---TCACTCTAACA---TT-----------CTCAATAGAATCATC-------T------GAAGGATA---A-----T----TCAAAT---------CCTGCAGGAACTGGACGGCGTCACCCTGGAGTCGAATATACGCTGGGCCGATGCATT | |
| droGri2 | scaffold_15126:4964908-4965058 - | ACAATGAGTTGATTCAGTTTGACATACTGGATGCCACCGGGCAGTTGCATGTGAGTG------AATTGCATTAG-------------------TTCTTCGAGTCG----------------AA-------TTCGAT-----AGAGTT---------CTTGTAGGAGCTGGACGGCGTTACACTGGAGTCCAACATACGCTGGGCCGATGCGTT | |
| droWil2 | scf2_1100000004585:5306980-5307135 + | ATAAAGAACAAGTTCAATTTGATATACTAGACGCTACGGGGCAATTGCATGTAAGTTTTT---TCTGAGAAAGT-----------------A--GTCGCAAGACT--------------------T----AGAGAGCCTT--ACTTC---------TTGCTAGGAATTGGATGGCGTTAGTCTGGAATCCAATATCAGATGGGCAGATGCTTT | |
| dp5 | 4_group1:4139891-4140052 - | ACAAAGAGCAGGTGCAGTTCGATATACTAGATGCCACAGGGCAGTTACAGGTGAGTCTTT---AATTTC---------------------GAATT----ATT---GGACTACTGATTATTA-------CATTTGGGG----TTCCTT---------ATTTTAGGAACTGGATGGTGTTACTTTGGAGTCGAATATACGCTGGGCGGATGCCTT | |
| droPer2 | scaffold_5:1124097-1124258 + | ACAAAGAGCAGGTGCAGTTCGATATACTAGATGCCACAGGGCAGTTACAGGTGAGTCTTT---AATTTC---------------------GAATT----ATG---GGACTACTGATTATTA-------CATCTGGGG----TTCCTT---------CCTTTAGGAACTGGATGGTGTTACTTTGGAGTCGAATATACGCTGGGCGGATGCCTT | |
| droAna3 | scaffold_12916:15297367-15297529 - | ACAAGGAGCAAATTCAATTTGACATTCTCGACGCTACCGGCCATCTACAAGTGGGTCTCA---AGTATT---------------------CCTTT----TAA---ATACTCTT------TAAATATAAAATAAGTGG----CTTTAT---------TTAACAGGAACTAGATGGTGTCACTCTCGAGTCCAACATCCGGTGGGCGGATGCTTT | |
| droBip1 | scf7180000396572:1722948-1723106 + | ACAAGGAACAAATTCAGTTTGACATTCTGGATGCTACCGGCCATTTACAAGTGGGTGTCA---AGTATT---------------------CCTTT----TAA---ATACTTTT------GAAATATA---A-ATTGG----CTTTCT---------TTATCAGGAACTAGATGGAGTCACTCTGGAGTCCAACATCCGGTGGGCGGATGCTTT | |
| droKik1 | scf7180000302384:97518-97676 + | ACAAGGAGCAGATTCAGTTTGACATTCTGGATGCCACCGGTCAGCTGCAAGTGAGTGGAA---TGACTTTTTAAGTAGGTTTAAAGTT-TAT--------------------------------------TCT-GGGTTA--TGTTC---------TTTAAAGGAATTAGATGGCGTGAGTCTGGAGTCCAATATACGCTGGGCGGATGCCTT | |
| droFic1 | scf7180000454115:81893-82049 - | ACAAGGAGCAAATTCAGTTTGACATTCTGGATGCCACCGGCCACCTGCAAGTGGGATTTA---AATCTCTTTTT----------------CACATTAGAAAAATT--------------TT---------ATT-AAA----TCTTTT---------TTTATAGGAACTAGATGGCGTTAGTCTAGAATCGAACATTCGCTGGGCCGATGCTTT | |
| droEle1 | scf7180000491028:1664512-1664671 + | ACAAGGAGCAAATTCAGTTTGACATTCTGGATGCCACCGGTCGGCTGCAAGTGGGTTTTA---AATTTGGCTTA----------------TAAATT-GAAGAA-------------AGTTT---------GTAAAGACTC--CATTC---------TTTATAGGAACTAGATGGCGTCAGTCTGGAGTCCAACATTCGCTGGGCCGACGCATT | |
| droRho1 | scf7180000767146:1-156 - | ACAAGGAGCAAATTCAGTTTGACATTCTGGATGCCACCGGTCGGCTGCAAGTTGGTTTAA---A-TTATAC--ATAAATT-AAAAAGTTT--------------A-------------------------GTAAAGACTC--TTTTC---------ATGATAGGAACTAGATGGCGTAAGTCTGGAGTCCAATATTCGCTGGGCCGACGCTTT | |
| droBia1 | scf7180000302422:9087015-9087178 - | ACAAGGAGCCCATTCAGTTTGACATTCTGGATGCCACCGGCCACCTGCAAGTAGGTTTTA---AAGTAAATTTA----------------TTCTTCTTTAAAATA-------------------------TTTACTAAAA--T---TGTTGTCTTATTTTTAGGAACTAGATGGCGTTAGTTTGGAGTCCAACATCCGCTGGGCCGACGCCTT | |
| droTak1 | scf7180000415604:180388-180546 + | ACAAGGAGCAAATCCAGTTTGACATTCTGGATGCCACCGGTCAGCTGCAAGTGGGTTTGA---AATTTCACTAGAAATTCTTAAGAAT-TTA--------------------------------------TTT-AAAATA--TTTAC---------TTTTTAGGAACTAGATGGTGTTAGTCTGGAGTCCAATATCCGCTGGGCCGATGCCTT | |
| droEug1 | scf7180000409463:569238-569397 - | ACAAGGAGCAAATCCAGTTTGACATTCTGGATGCCACTGGACAGCTGCAAGTAAGGTTTT---GGTTAGAAATA----------------TAATAT-AA-------------AGAAAGTTA---------CTAGT-ACTA-TCTTTC---------TATAAAGGAACTAGATGGCGTTAGTCTGGAGTCCAATATCCGCTGGGCCGACGCCTT | |
| dm3 | chr2L:7391970-7392125 - | ACAAGGAGCAGATTCAGTTTGACATTCTGGACGCCACCGGTCAGCTACAAGTGGGTTTTA---AATTGCATTTT----------------TCATTTTGCAAGACA-------------------------TTAAAGA----CCATTT---------CATGTAGGAACTAGATGGAGTTAGTCTGGAGTCCAACATCCGCTGGGCCGACGCATT | |
| droSim2 | 2l:7144880-7145039 - | ACAAGGAGCAGATTCAGTTTGACATTCTGGACGCCACCGGTCAGCTGCAAGTGGGTTTTA---AATGGGATTTT----------------CAATTTTGCATGACA-------------------------TTAAAGACCATTTTTTT---------TTTTTAGGAACTAGATGGAGTTAGTCTGGAGTCCAACATCCGCTGGGCCGACGCATT | |
| droSec2 | scaffold_3:2906897-2907054 - | ACAAGGAGCAGATTCAGTTTGACATTCTGGACGCCACCGGTCAGCTGCAAGTGGGTTTTA---AATGGGATTTT----------------CCATTTTGCAAGACA-------------------------TTAAAGACCA--TTTTT---------TTTTTAGGAACTAGATGGAGTTAGTCTGGAGTCCAACATCCGCTGGGCCGACGCATT | |
| droYak3 | 2L:5511126-5511284 + | ACAAGGAGCAGATTCAGTTTGACATTCTGGACGCCACCGGTCAGCTGCAAGTGGGTTTTTTTTAATTGAATGTA----------------CAATTTTGGAAGACT-------------------------TTAAGGA----CCTGTT---------GTTGTAGGAACTAGATGGAGTCAGTCTGGAGTCCAATATCCGCTGGGCCGACGCATT | |
| droEre2 | scaffold_4929:16308415-16308570 - | ACAAGGAGCAGATTCAGTTTGACATTCTGGACGCCACCGGCCAGCTGCAAGTGGGTTTTA---AACTGGATTTT----------------CAATTTTGCAAGACA-------------------------TTAAAGA----CATGAT---------GTTGTAGGAACTAGATGGAGTTAGTCTGGAGTCCAACATCCGCTGGGCCGACGCATT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 05:07 PM