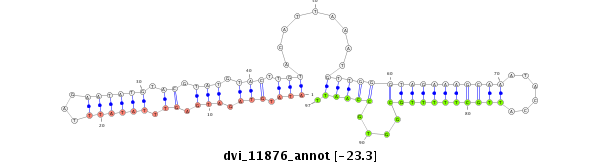
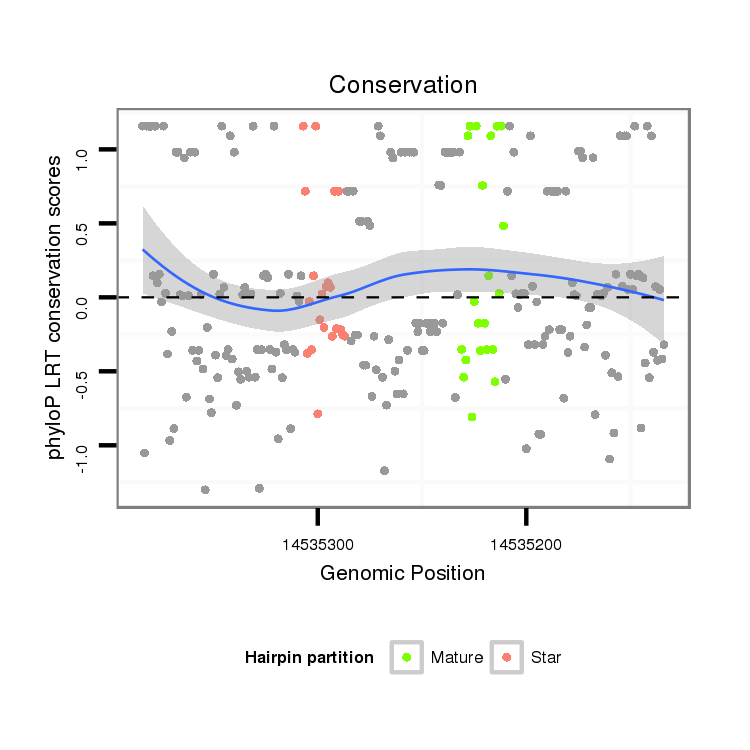
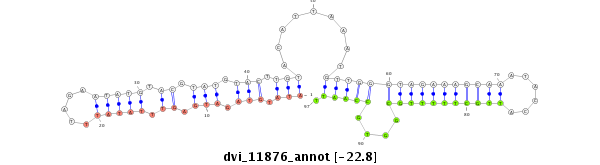
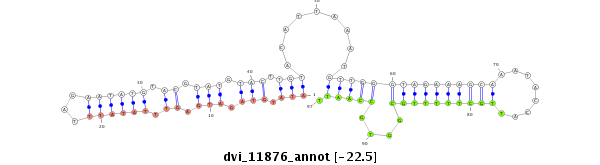
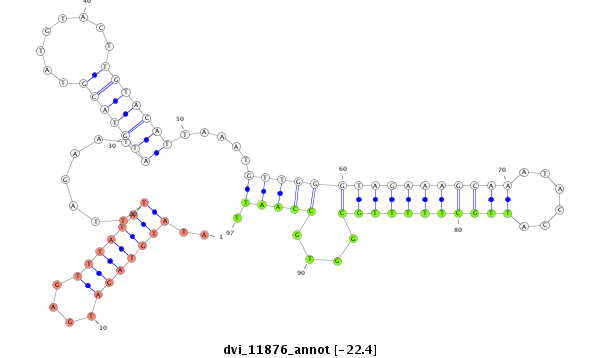
ID:dvi_11876 |
Coordinate:scaffold_12963:14535184-14535334 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -22.8 | -22.5 | -22.4 |
 |
 |
 |
exon [dvir_GLEANR_698:2]; CDS [Dvir\GJ21636-cds]; intron [Dvir\GJ21636-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AAAAATTGTCTTTGTGTTCAGTATAAATGAGCTGAGCATTATGCAGTAGCTTTTAAGCTTGATTTAACACACATCTAATATGTAGATGAGTTTATATTTAGAATATGTACGTATGTACTTGTACATTAAATGTTGGGTAGAAAGCAAATACCATTGCTTTTTGCGGTGCCAATTTTAATTTGAAACGAACATTTTCAAAAGGAACTCGTTTGTGCGCCTCGTATCATTACACCCTGTATGGCAGCGATGCG *****************************************************************************(((.(((.(((.((.((((((...)))))).)).))).))).))).........((((((((((((((((......)))))))))))....))))).***************************************************************************** |
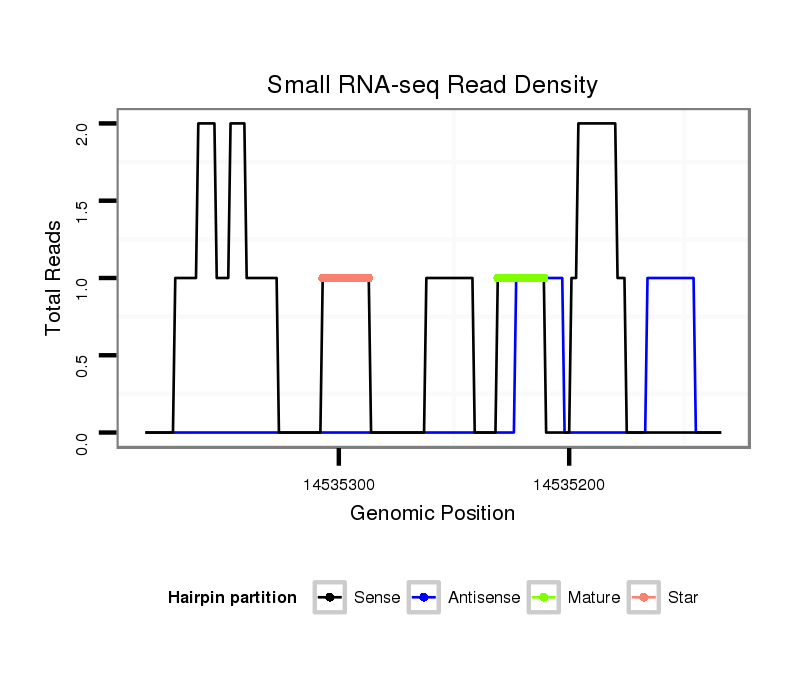
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060676 9xArg_ovaries_total |
V053 head |
M028 head |
M061 embryo |
SRR060664 9_males_carcasses_total |
SRR060667 160_females_carcasses_total |
SRR060673 9_ovaries_total |
SRR060677 Argx9_ovaries_total |
SRR060689 160x9_testes_total |
SRR1106726 embryo_14-16h |
SRR1106729 mixed whole adult body |
V047 embryo |
SRR060674 9x140_ovaries_total |
SRR1106715 embryo_4-6h |
M027 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................................................................................................................................................................CCTCGTATCATTACACCCTGTATGGCAC....... | 28 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................CGAACATTTTCAAAAGGAAC.............................................. | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............GTGTTCAGTATAAATGAG............................................................................................................................................................................................................................ | 18 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................ACTTATACATTAAATGTTGGG.................................................................................................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................ACATTTTCAAAAGGAACTCGT.......................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................................................AACGGAACTCGTTTGTGCGCC................................. | 21 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................GTGTAGAAAGCAAATACCATTGC.............................................................................................. | 23 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................ATTATGCAGTAGCTTTTAAGC................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................CATTTTCAAACGGAACTCGT.......................................... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................TTGCTTTTTGCGGTGCCAATT............................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................TCGGAAGGAACTCGTTTGTGC.................................... | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................ATATGTAGATGAGTTTATATT......................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................ACATTAAATGTTGGGTAGAAA............................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .......................TAAATGAGCTGAGCATTATGC............................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................TTTTCAAAGGGAACTCGTTTGT...................................... | 22 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................CACATCTTATATGTAGA..................................................................................................................................................................... | 17 | 1 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........................................................................................................................................................................................TGAACATTTTCAAAAGGAA............................................... | 19 | 1 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................AATGGGCTGAGCATTGTGCAG............................................................................................................................................................................................................. | 21 | 2 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................................................................................ATTAAAGATTGGGTAGAAAAC.......................................................................................................... | 21 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTTTTAACAGAAACACAAGTCATATTTACTCGACTCGTAATACGTCATCGAAAATTCGAACTAAATTGTGTGTAGATTATACATCTACTCAAATATAAATCTTATACATGCATACATGAACATGTAATTTACAACCCATCTTTCGTTTATGGTAACGAAAAACGCCACGGTTAAAATTAAACTTTGCTTGTAAAAGTTTTCCTTGAGCAAACACGCGGAGCATAGTAATGTGGGACATACCGTCGCTACGC
*****************************************************************************(((.(((.(((.((.((((((...)))))).)).))).))).))).........((((((((((((((((......)))))))))))....))))).***************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060658 140_ovaries_total |
SRR060671 9x160_males_carcasses_total |
SRR060675 140x9_ovaries_total |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................................................................ACGCGGAGCGTAGTAGTGTG................... | 20 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................................TTACAACCCATCGATCGTTTAT..................................................................................................... | 22 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 |
| ..........................................CGTCGTCGAGAATTCGAACTA............................................................................................................................................................................................ | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 |
| .................................................................................................................................................................ACGCCACGGTTAAAATTAAAC..................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................................................................AGCATAGTAATGTGGGACATA............ | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 |
| .............................................................TAGGTTGTGTGTAGATTAT........................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 1 | 0 | 0 | 0 |
| .......................................................................................................................................................................................................CCCTTGAGCGAACTCGCGGA................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:14535134-14535384 - | dvi_11876 | AAAAAT---TG-----TCTT-TGTGTT--------------CAGTATAAATGA--GCTGAGCATTATGCAGTAGCTTTTAAGCTT--GATTTAACACACATCTAATATGTAGATGA--------------------GTTTATATT---TAGAATATGTACGTA-----------------TGTAC----------------------TT------------GTA-CATTAAATGTTGGGTAGAAAGCAAATACCA-------------------------TT------GCTTTTTGCGGTGCCAAT--------------TTTAATTTGAAACGAACATTTTCAAAAGGAACTCGTTTGTGCGCCTCGT--ATCATTACACCCTGTATGGCAGCG--ATGCG |
| droMoj3 | scaffold_6500:17452092-17452410 - | ACAAAT---TAAGCAGTCTTCAATATT--------------CAGAATGATCGTCTACGTAGTTTCACGCATCTGCTCTTGGTTTTTGGGATTTATGTATGTTTAATATATATATGT--------------------ACATATACA---TAGAGC------TAC---GGTCATAAAAGTAGTGCGACTGGGCACTAAATGAAAACAAACTTGGTAACATGCTGACTTAGTGAAT--------------AAATATCAACAGGTAAATTAATTATTTTTATCACAGCTGGAACTATT----ATGTAAATTCAAGATACGTTTATATAATTTGAATTGAGCGCCATTAAAAAA-ATAT-------------ATATACGAGTTCACCCTGTATTGAAACA--GCGCA | |
| droGri2 | scaffold_15252:8232123-8232379 - | AGAAATATTTG-----TGTA-TTAT---------------------------TCTCTGTATTTTTGCGT-----TGCCTGGCTTTTGGGATACTTATAAGATGAATCGATATGCAAACGGTTGCATAAATAAAGAAGTTTGTTTTATGTATAATATACACTTCGGTGTTCGGGTCAGTAGTGCC----------------CAACA----------------------------------------------------------------AATTTGTAGCGCAGCTG---CTCGT----ATGTCATT----------------TAA----TTGCGAAGTATTTCAAAAGGCATTCGTTTGCGCGCGTCAATTGTGGATACACCCTGTATAGCAACAGAGTGTG | |
| droSec2 | scaffold_3:5339276-5339523 - | AGAAAA---TG-----CCTT-ATAGTTATAGTGCAGTTATCTTGCTTAAATAA--TTTGGTATTTTTGAA---ACAATTTAGCAC--AATTCTAAGCGCACTTTA--TGAAAGTAA--------------------GT---------------------------------------TAATGAGATTGAGTCTCAATAGTCAATAAGCT------------GAA-TATTAAATAACTTACTAGTAGTAAATAATA-------------------------TG----GAACTCTTCTCAGAGCAAAT-----------------ATTCGAAATTGCGCGCC-------------CAATTGTCATCCCTAT--TTCCACCCCCCACTTTCGATATC---GATAA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/16/2015 at 10:10 PM