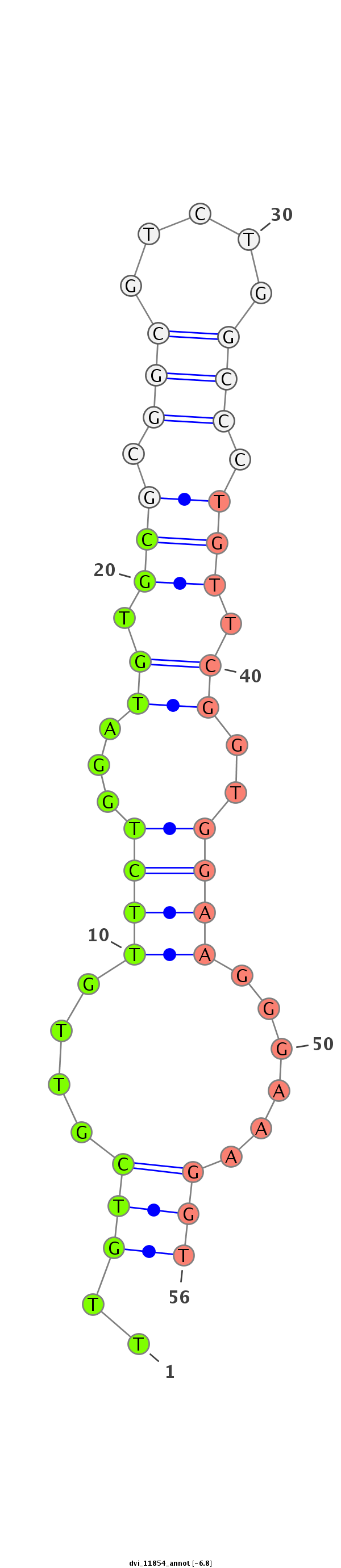
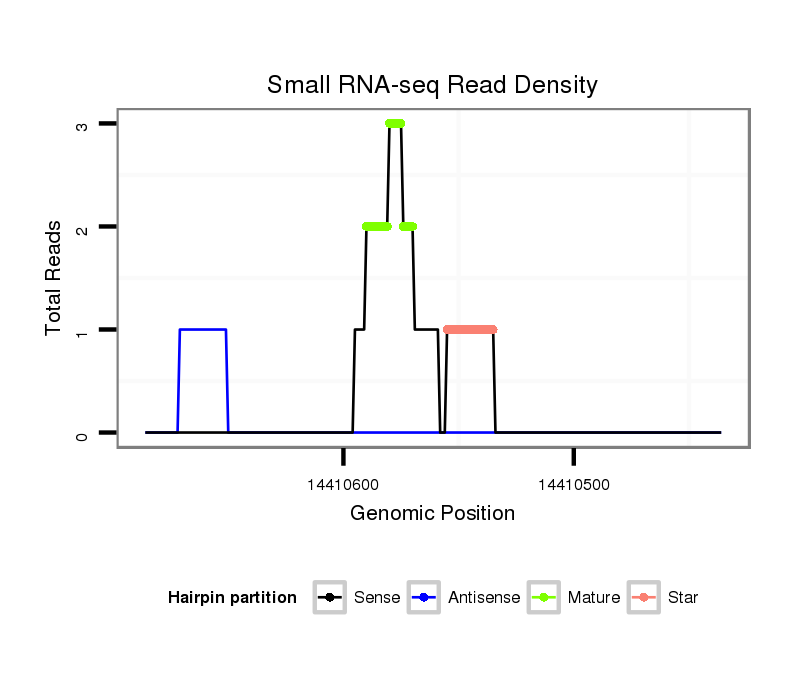

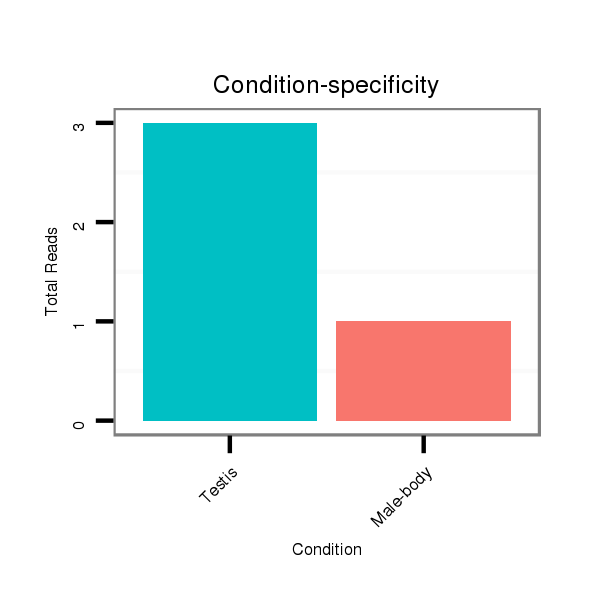
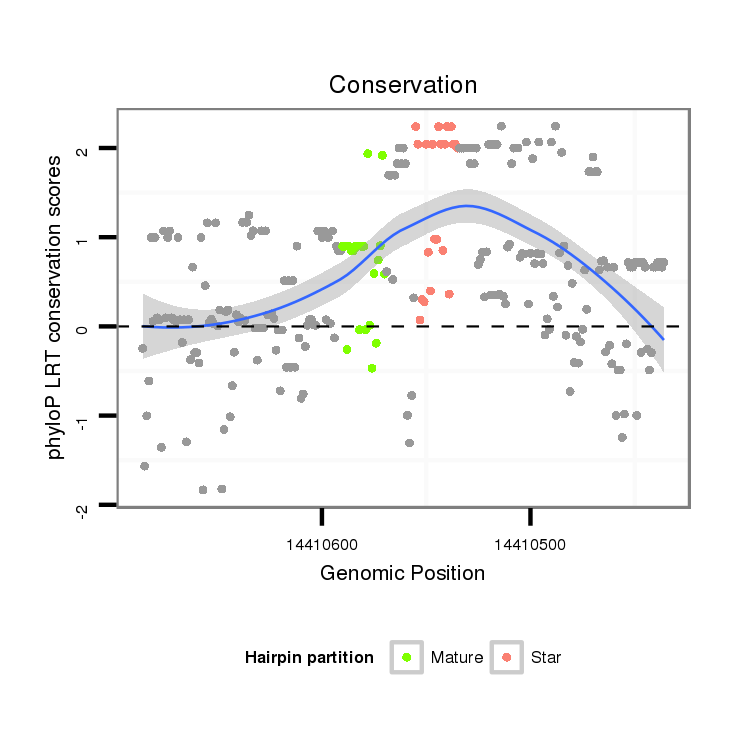
ID:dvi_11854 |
Coordinate:scaffold_12963:14410486-14410636 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -13.9 | -13.5 | -13.2 | -13.2 |
 |
 |
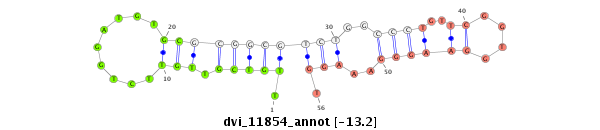 |
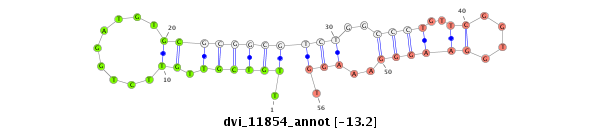 |
exon [dvir_GLEANR_712:2]; CDS [Dvir\GJ21792-cds]; intron [Dvir\GJ21792-in]
| Name | Class | Family | Strand |
| (CAGAGA)n | Simple_repeat | Simple_repeat | + |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## GTAGGGCAGCAAGATGCGCTGGCGCCCTGTCTCCGTCTCTGTCTCTGTCTCAGTCTCAGTCTCTACCCTCTATTTGATGGGGTGTTGTCCTTGTCCTTGTCGTTGTTCTGGATGTGCGCGGCGTCTGGCCCTGTTCGGTGGAAGGGAAAGGTTATTAGACTTAGCCCAGACATTTTTGTTAAAATAACCTTTCGCTTTTAGGTGGTTGGCTCGTCGACGGAAACAACCTTGCGTGGTCTGAGTGCGGTTCA ************************************************************************************************..(((....((((...((.(((.(((.....))).))).))..))))......)))*************************************************************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060659 Argentina_testes_total |
V116 male body |
V053 head |
SRR060657 140_testes_total |
SRR060689 160x9_testes_total |
M047 female body |
SRR060687 9_0-2h_embryos_total |
V047 embryo |
SRR060666 160_males_carcasses_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............................................................................................................................................GAAGGGAAGGGTTATTGGAC........................................................................................... | 20 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................TTGTCGTTGTTCTGGATGTGC...................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................TGTTCGGTGGAAGGGAAAGGT................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................AGCCTTGCGTGGTCTGAGTG....... | 20 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................TCTGGATGTGCGCGGCGTCTGG........................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................................................................TGTCCTTGTCGTTGTTCTGGA........................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................CAGCGTCTCGCCCTGTTCGGTC............................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .................................................TCAGTCTGTGTCTCTAACCTCT.................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................TCAGTCTTTGTCTCTATCCTCT.................................................................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................CTCTACTTGATGGGTTGT...................................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| .................................................TCAGTCTCTGTCTCTATCCTGT.................................................................................................................................................................................... | 22 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .................................................TCAGTCTCTGTCTCTATCCT...................................................................................................................................................................................... | 20 | 2 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................CTCTTTTTGAGGGGGTGT...................................................................................................................................................................... | 18 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .................................................TCAGTCTTCGTCTCTATCCT...................................................................................................................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................................................CCGTGGTCTGGGTGCGGTG.. | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
CATCCCGTCGTTCTACGCGACCGCGGGACAGAGGCAGAGACAGAGACAGAGTCAGAGTCAGAGATGGGAGATAAACTACCCCACAACAGGAACAGGAACAGCAACAAGACCTACACGCGCCGCAGACCGGGACAAGCCACCTTCCCTTTCCAATAATCTGAATCGGGTCTGTAAAAACAATTTTATTGGAAAGCGAAAATCCACCAACCGAGCAGCTGCCTTTGTTGGAACGCACCAGACTCACGCCAAGT
***************************************************************************************************..(((....((((...((.(((.(((.....))).))).))..))))......)))************************************************************************************************ |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106729 mixed whole adult body |
SRR060666 160_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
SRR060684 140x9_0-2h_embryos_total |
M047 female body |
SRR060665 9_females_carcasses_total |
SRR060668 160x9_males_carcasses_total |
SRR060657 140_testes_total |
SRR060669 160x9_females_carcasses_total |
SRR060682 9x140_0-2h_embryos_total |
V116 male body |
SRR060679 140x9_testes_total |
SRR060671 9x160_males_carcasses_total |
SRR060674 9x140_ovaries_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060655 9x160_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ....................................................................................................................................................................GGGACTGTAAAGCCAATTTT................................................................... | 20 | 3 | 14 | 2.79 | 39 | 0 | 10 | 8 | 3 | 1 | 3 | 3 | 0 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 0 |
| ...............CGCGACCGCGGGACAGAGGCA....................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................AGTCAGAGAGCGGTGATAAA................................................................................................................................................................................ | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................ACCGCGGGTCCGAGGGAGAG.................................................................................................................................................................................................................... | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................TTCTAATACTCTGAATCGCG.................................................................................... | 20 | 3 | 6 | 0.17 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................AGGCAGAGACAGAGACAG.......................................................................................................................................................................................................... | 18 | 0 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................CAACAGGAACTGGAACAGGT.................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .................................................................................................ACAGCTACAAGACGTACCC....................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:14410436-14410686 - | dvi_11854 | GTAGGGCAGCAAGATGCGCTGGC-GCCCTGTCTCCGTCTCTGT------------------------CTCTGTCTCAGTCTCAGTCTCTACCCTCTATTTGA-TGGGGT---GTTGTCCTTGTCCTTGTC---------------------------------------GTTGTTCTGGAT-------GTGCGCGGCGTCTGG-----------CCCTGT------TCGGTGGAAGGGAAAGGTTATTAGACTT--------------------------------AGCCCAGACA-----------------TTT--------------------TTGTTAAAATAACCTTTCGCTT-------TTAGGTGG--TTGGCTC---------GTCGACGGAAACAACCT-----TGCGTGGTCTGAGTGCGGTTCA |
| droMoj3 | scaffold_6500:15361531-15361807 - | AAGGGACAGTAAGATGTGCTGTC-ACCCCGAC----------------------------------------------------------------------TGGGGGT---GTTGTCCTTGTCCTTGTCGTTGCCGCCATCGCTGTTGTTGTTGTTATTGGT------GTTGTTCTCGAG-------GTGCGCGTCGTCTGC--T---------CCTGC------TCGTTGAAAGGGAGAGGTTATTAGACTT--------------------------------AGCCCAGACATTTGCCGCGGCTGCTCCTTTTTTTCTTTTCTATTTTCATTTTGTTATAATGACCTTTCACTT-------TTAGGTTG-GGTACGCC---------GACGACGGAAGCATCAG-----GTTGTGGTATGAGCGCGGTTCA | |
| droGri2 | scaffold_15126:1532319-1532569 + | GAACGGCAGGAAGATGCGCCGCCGACAACGAC------CTCTC------------------------CATTCCCTCA--------TTCCACTCTTTGTCTGCTGAGGGCGTCGTTGTCCTT----------------------------------------------------------------------------GTCTGC--TGGTTGTCGGTTTGTCGGTTTGGGGCGAAAGGGAAAGGTTATTAGACTT--------------------------------AGCCCAGACATTGGCTGCGGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCACTT-------TTGGGTCTTA--GCAGT-------TAGTCGACGGAAGCGTCTGCCTCAGCAATGGTTTTAGTCAGGTTCA | |
| droWil2 | scf2_1100000004511:9622681-9622864 - | GCACGGCGAAAGGACACGTCAGT-----TGCCTCCGTCTATGTGACGTGGCCACAGTAAATTCCTTGACTCGTCGCAGTTGTAATCATTTTAT------TTT-TATAAT---TTTGTTCGTTTCCTTCTC---------------------------------TTTTTAGTTATTTTCAATTTTTATTATGCC---------T--CGGGTGTGCACCTGTTGTGTTGCGGTGAGAGAGAGAGG------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ | |
| dp5 | 2:5653302-5653303 - | GT------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_13:726926-726969 + | GCT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AGAGTCCTGTCGCT------GCCTCTGCCTCT--------------------GC---------------------------CTCTGCTT----CTGCC----------------------------------------------------- | |
| droAna3 | scaffold_13258:995729-995828 + | CTG---------------------------TCTCTGTCTCTGT------------------------CTCTGTCTCAGTCTCAGTCTCTGT-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCTGTCTCTGTCTCT------GTCTCTGTCTC----------------------------TGTCTCTGTCTCTGT-------CTCTGTCT----CTGTC---------TC-----TGT---------------------------------- | |
| droBip1 | scf7180000396580:1155483-1155662 - | TAT---------------------------CCTTTTTGTGGGT------------------------G------------------------------------------------------------------------------------------------------------TGTGC-------GTGGGCGTCGTCTGC--CAC------CGTTGT------TCGGTGAAAGGGAAAGGTTATTAGACTCGTCCC--GTGCAGAGCCATA--TAGTGGTAGGGGCCCAGACACT------GACTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACTGTCGTCGTCGTCGACGGAA---------------------------------- | |
| droFic1 | scf7180000453842:958932-959089 + | TC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGAGC-------GTGGGCGTCGTCTGG--TGG------AGTTGT------TCGGCGAAAGGGAAAGGTTATTAGACTCGCCTC--GTGCAGAATTTTAAGAAGTGGTTGGGGCCCAGACATT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGCT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droEle1 | scf7180000491338:107135-107294 - | GTT---------------------------GT------------------------------------------------------------------------------------------------------------------------------------------------TGTGT-------GTGGGCGTCGTCTGC--TGGTT----TTTTGT------TCGGTGAAAGGGAAAGGTTATTAGACTCGCCCC--GTGCAGAATTTTA---AATGGTGGGGGCCCAGACATG------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droBia1 | scf7180000302261:731270-731396 - | T-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTGGGCGTCGTCTGC--TGG------CTTTGC------TCGGTGAAAGGGAAAGGTTATTAGACTCGCC-C--GTGCAGAATTTTA---AGTGGTGGGGGCCCAGACACT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TT-------------------------------------------------------------------- | |
| droEug1 | scf7180000409005:1187286-1187442 - | TC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTGT-------GTGGGCGTCGTCTGGCGTGG------ATTTGT------TCGGTGAAAGGGAAAGGTTATTAGACTCGCCCC--GTGCAGAATTTTA---AGTGGTGGGGGCCCAGACATT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droSim2 | 2l:5095009-5095163 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GTGT-------GTGGGCGTCGTCTGG--TGG------ACTTGT------TCGGTGAAAGGGAAAGGTTATTAGACTCGCCCCGCGTGCAGAATTCGA---AGTGGTGGGGGTCCAGACACT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droSec2 | scaffold_5:3365816-3365970 - | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAGT-------GTGGGCGTCGTCTGG--TGG------ACTTGT------TCGGTGAAAGGGAAAGGTTATTAGACTCGCACCGCGTGCAGAATTCGA---AGTGGTGGGGGTCCAGACACT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droYak3 | 2L:13911463-13911617 + | TC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTGT-------GTGGGCGTCGTCTGG--TGG------ACTTGT------TTGGTGAAAGGGAAAGGTTATTAGACTCGCCCC--GTGCAGAATTTTA---AGTAGTGGGGGTCCAGACACT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGTT-------TTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- | |
| droEre2 | scaffold_4929:5366905-5367066 - | TC------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TGTGT-------GAGGGCGTCGTCTGG--TGG------ACTTGC------TCGGTGAAAGGGAAAGGTTATTAGACTCGCCCC--GTGCAGAATTTGA---AGTAGTGGGGGTCCAGACACT------GGCTGCTCCTTA--------------------TTGTTATAATAACCTTTCGGCTTTCGGTTTTCTGTTT----TCACT---------GTCGACGGAA---------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/16/2015 at 09:57 PM