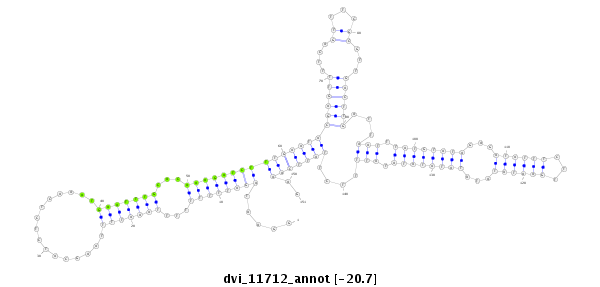
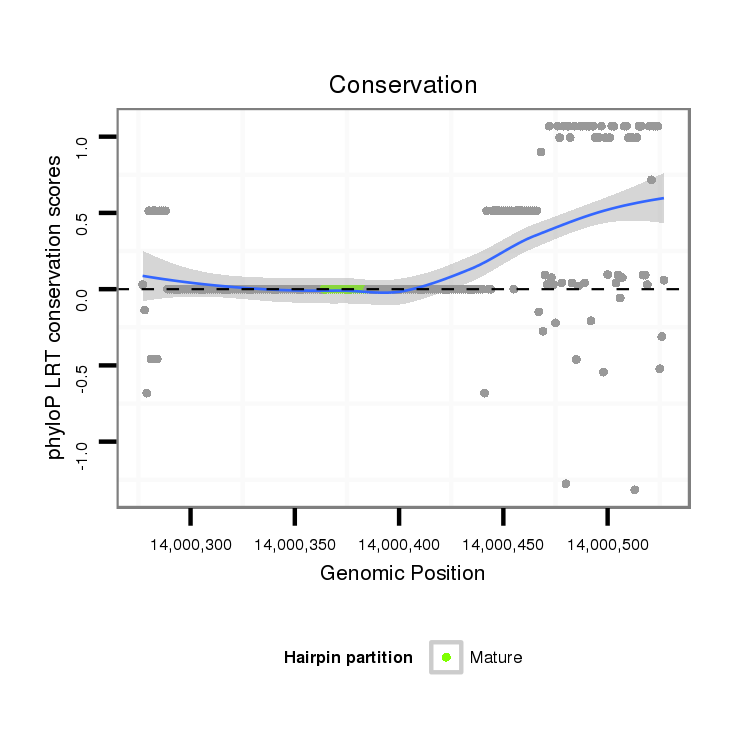
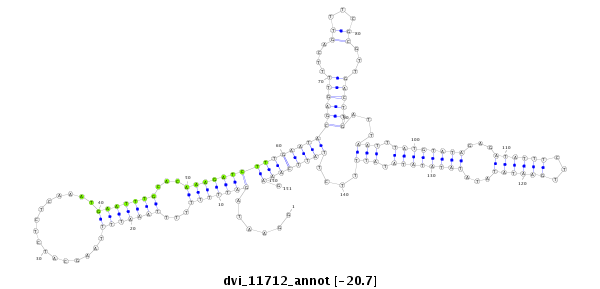
ID:dvi_11712 |
Coordinate:scaffold_12963:14000327-14000477 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
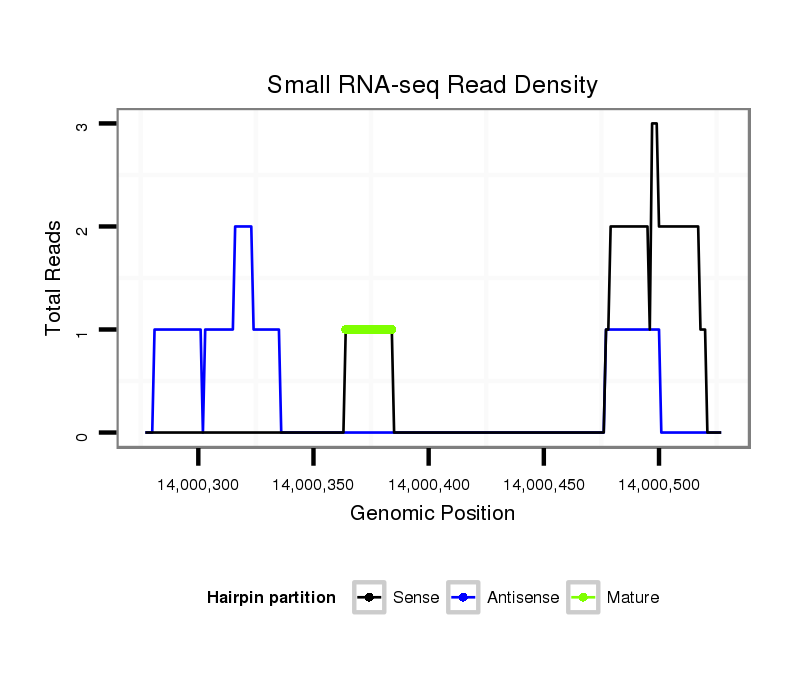
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -20.7 | -20.0 | -20.0 |
 |
 |
 |
exon [dvir_GLEANR_2103:4]; CDS [Dvir\GJ17516-cds]; intron [Dvir\GJ17516-in]
| Name | Class | Family | Strand |
| AT_rich | Low_complexity | Low_complexity | + |
| mature | star |
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## AAAACAACATTTTCCATTGGATTCTGGAAATTTCTGAGTTCCGCCAAGGAGGAATAGATTTTTTTTTAAATTTTAAGCATCTCTCAAATGAATTTGCACAAAGATCTTTGAATACGAGTTTTCAGTTTCGCGTTGACTTGATTAATTTATGTATAGAGATATTTCTTGAATATATATATATATATATTTTCTTATTCAAAGGTTTGTATCTACAAGAGCCTGCAGAAGTCGAAGCCCGAAAATTATTTAAA **************************************************.....((((((((...(((((((................)))))))...))))))))(((((((((((((....((...))...))))))...(((.((((((((...((((((...))))))...)))))))).)))....)))))))..************************************************** |
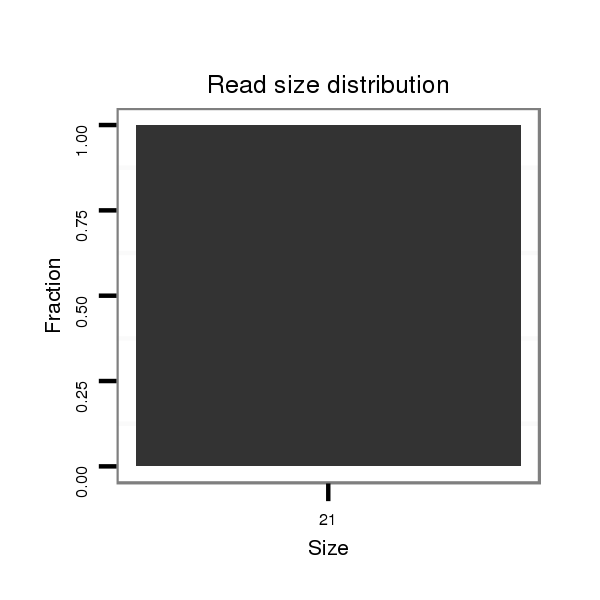
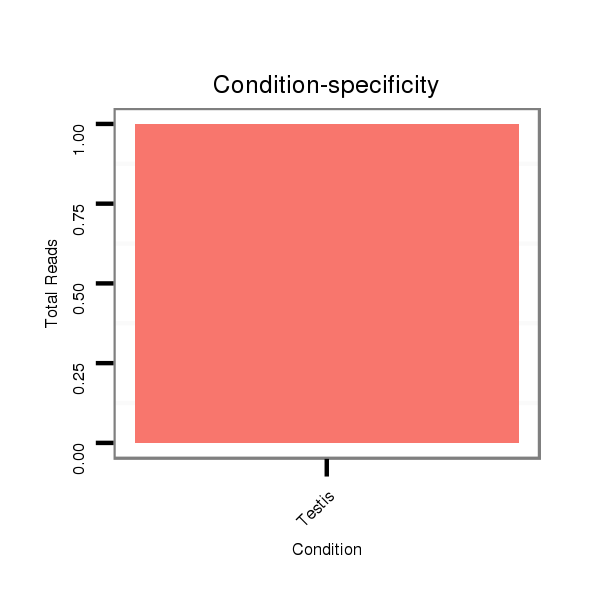
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060683 160_testes_total |
SRR060676 9xArg_ovaries_total |
SRR060663 160_0-2h_embryos_total |
SRR060664 9_males_carcasses_total |
SRR060685 9xArg_0-2h_embryos_total |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|
| .......................................................................................ATGAATTTGCACAAAGATCTT............................................................................................................................................... | 21 | 0 | 2 | 1.00 | 2 | 2 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................GGTTTGTATCTACAAGAGC................................ | 19 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ............................................................................................................................................................................................................................TGCAGAAGTCGAAGCCCGAAAATT....... | 24 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................TGCAGAAGTCGAAGCCCGAAA.......... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................................................TTTGTATCTACAAGAGCCTGC............................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................................................................................................................................GAAGTTTGTATCTACAAGAGC................................ | 21 | 2 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................................AGGAGGAATAGAGATTTTGTT........................................................................................................................................................................................ | 21 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................GTCCCGCCAACGAGGAATT................................................................................................................................................................................................... | 19 | 3 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
|
TTTTGTTGTAAAAGGTAACCTAAGACCTTTAAAGACTCAAGGCGGTTCCTCCTTATCTAAAAAAAAATTTAAAATTCGTAGAGAGTTTACTTAAACGTGTTTCTAGAAACTTATGCTCAAAAGTCAAAGCGCAACTGAACTAATTAAATACATATCTCTATAAAGAACTTATATATATATATATATAAAAGAATAAGTTTCCAAACATAGATGTTCTCGGACGTCTTCAGCTTCGGGCTTTTAATAAATTT
**************************************************.....((((((((...(((((((................)))))))...))))))))(((((((((((((....((...))...))))))...(((.((((((((...((((((...))))))...)))))))).)))....)))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060676 9xArg_ovaries_total |
SRR060689 160x9_testes_total |
SRR060655 9x160_testes_total |
SRR060671 9x160_males_carcasses_total |
SRR1106727 larvae |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ..........................CTTTAAAGACTCAAGGCGGTT............................................................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .......................................AGGCGGTTCCTCCTTATCTA................................................................................................................................................................................................ | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....GTTGTAAAAGGTAACCTAAGA.................................................................................................................................................................................................................................. | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................................................................................................................CCAAACATAGATGTTCTCGGACGT........................... | 24 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................CTAAACGTGTTTCTAGAAA.............................................................................................................................................. | 19 | 1 | 7 | 0.14 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................................................................................................................................................................................................................GTTTCCACCTTCGGGCTTTT......... | 20 | 3 | 11 | 0.09 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ........TAACAGGTAACCTAA.................................................................................................................................................................................................................................... | 15 | 1 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:14000277-14000527 + | dvi_11712 | AAA--ACAACATTTTCCATTGGATTCTGGAAATTTCTGAGTTCCGCCAAGGAGGAATAGATTTTTTTTTAAATTTTAAGCATCTCTCAAATGAATTTGCACAAAGATCTTTGAATACGAGTTTTCAGTTTCGCGTTGACTTGATTAATTTATGTATAGAGATATTTCTTGAATATATATATATATATATTTTCT------------------------TATTCAAAGGTTTGTATCTACAAGAGCCTGCAGAAGTCGAAGCCCGAAAATTATTTAAA |
| droMoj3 | scaffold_6500:29528553-29528611 + | T--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATTCAAAGGTGTGCAATTACAAGAGCCTACAGAAGTCGAAGCCAGAAAATTATTTAAG | |
| droGri2 | scaffold_15126:1842842-1842963 - | TACGTATAATATTT--------------------------------------------------------------------------------------------------------------------------------------------------------AT--AATATATATA-ATATATATTTTTTTTTTACTATAACAAATATTTTCGCAACTCGTAGATCTGCAATTATAAAAGCCTGCAGAAATCGAAGCCGGAAAACTATTTCCA | |
| dp5 | Unknown_group_17:50299-50360 - | TC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CT------------------------TTTTTATAGGTATGCATTTACAAAAGCCTTCGGAAGAGAAAGCCTGAATTTT-TTTTCA | |
| droPer2 | scaffold_16:215607-215668 - | TC----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CT------------------------TTTTTATAGGTATGCATTTACAAAAGCCTTCGGAAGAGAAAGCCTGAATTTT-TTTTCA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
Generated: 05/16/2015 at 08:46 PM