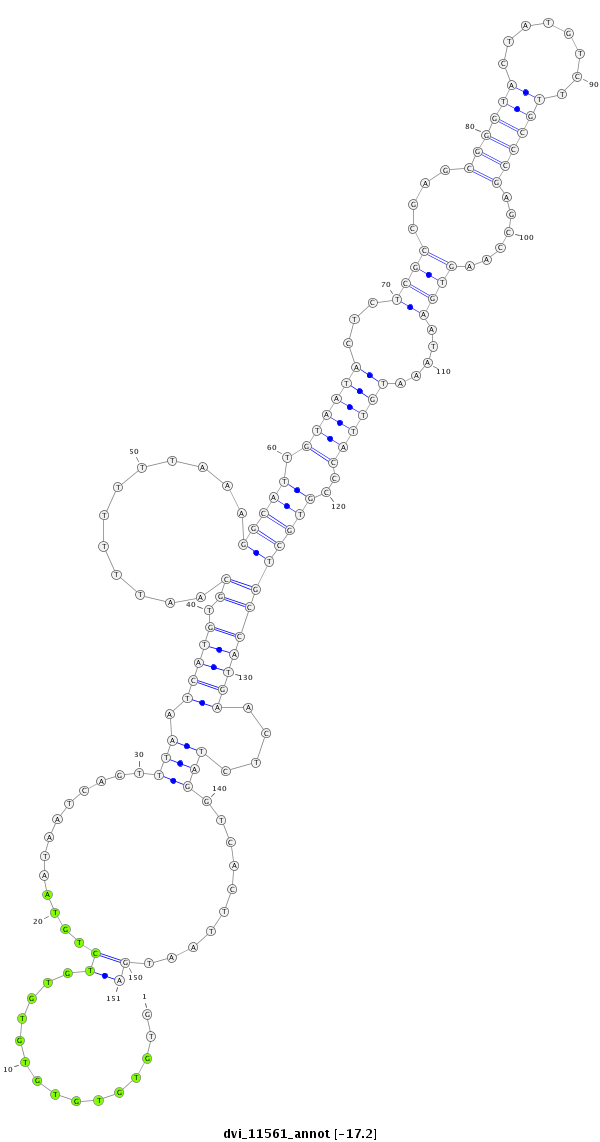
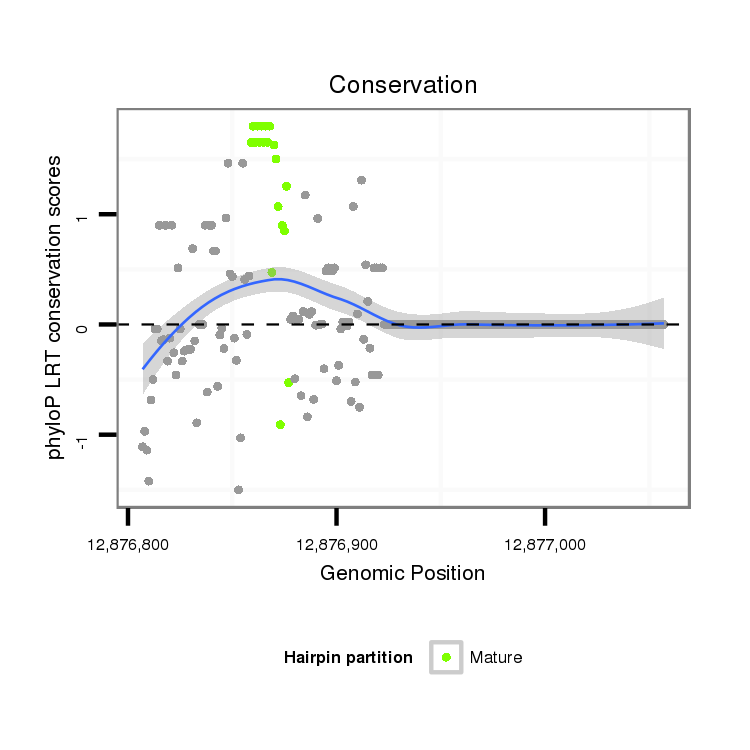
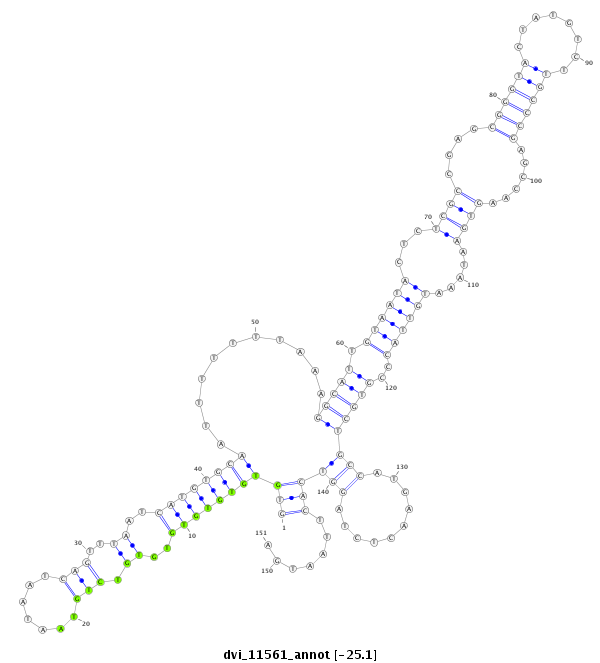
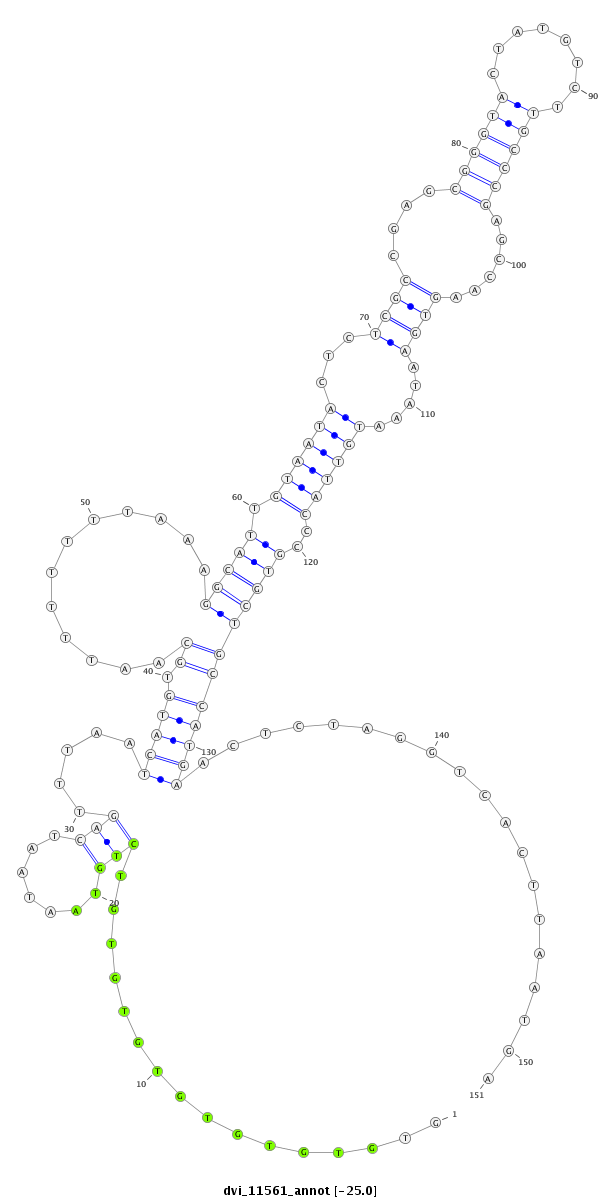
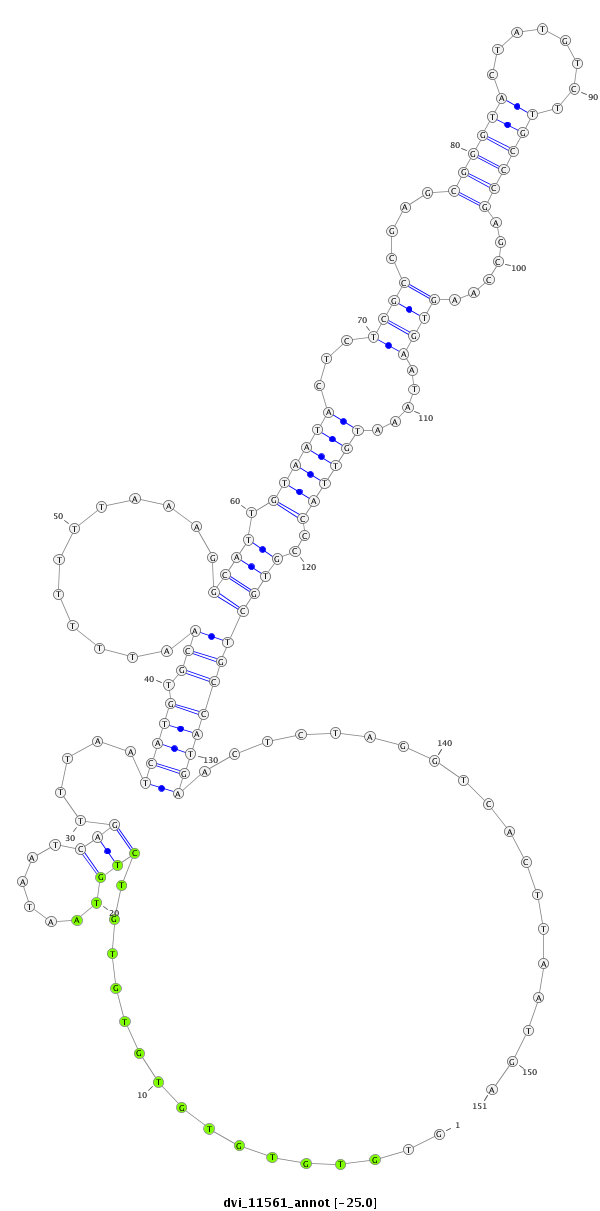
ID:dvi_11561 |
Coordinate:scaffold_12963:12876857-12877007 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
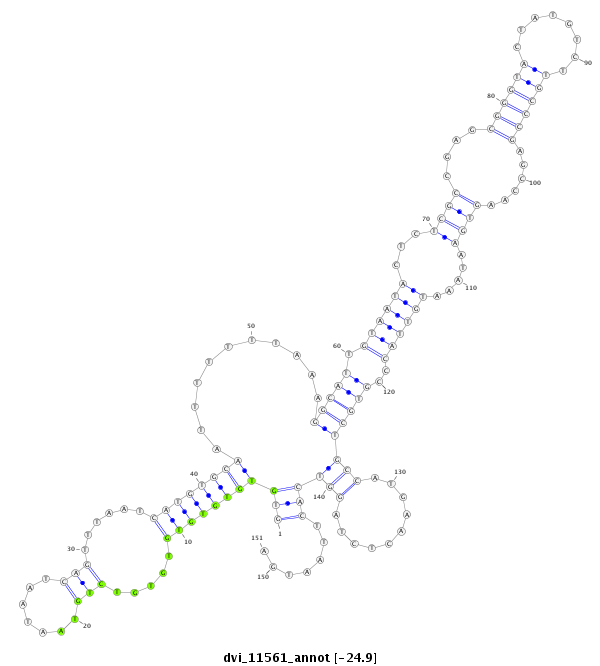
| -25.1 | -25.0 | -25.0 | -24.9 |
 |
 |
 |
 |
exon [dvir_GLEANR_2042:1]; CDS [Dvir\GJ17450-cds]; intron [Dvir\GJ17450-in]
No Repeatable elements found
| -----------------#################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- ATCGTTAAAGTTGCACCATGATGGACGGAGTCAACCAGATGCTTAACAGTGTGTGTGTGTGTGTGTCTGTAATAATCAGTTTAATCATGTGCAATTTTTTTAAAGGCATTGTAATACTCTCGCCGAGCGGGTACTATGTCTTGCCCGAGCCAAGTGAATAAATGTTACCCGTGCTGCCATGAACTCTAGGTCACTTAATGACCTCATCAGATCCTCGTTGATCTCCTCTCAAGTTGTGAACAGGGTATTTA **************************************************...............((.............(((.(((((.((............(((((.((((((...((((....((((((........))))))......)))).....))))))..))))))))))))....)))..........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
V053 head |
SRR060679 140x9_testes_total |
M027 male body |
M061 embryo |
M047 female body |
SRR060685 9xArg_0-2h_embryos_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................................GTCTTGCCCAAGCCGAGT................................................................................................ | 18 | 2 | 6 | 0.17 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................................................................TACCAGTGCCGCGATGAAC................................................................... | 19 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................CGGATCCTCGTTGATGTCCC........................ | 20 | 3 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................................CAGATCCTCGTTGATGTTCC........................ | 20 | 3 | 13 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................TTGGGTACTATGACTTGCC.......................................................................................................... | 19 | 3 | 16 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................AACAGTGTGTGTGTGTGT............................................................................................................................................................................................. | 18 | 0 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................GTGTGTGTGTGTGTCTGTA.................................................................................................................................................................................... | 19 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .....................................................TGTGTGTGTGTGTCTGTA.................................................................................................................................................................................... | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................TGTGTGTGTGTGTGACTGTAAAAA................................................................................................................................................................................ | 24 | 2 | 20 | 0.05 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................................AGGCTAAACCGTGTGTGTGT................................................................................................................................................................................................. | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TAGCAATTTCAACGTGGTACTACCTGCCTCAGTTGGTCTACGAATTGTCACACACACACACACACAGACATTATTAGTCAAATTAGTACACGTTAAAAAAATTTCCGTAACATTATGAGAGCGGCTCGCCCATGATACAGAACGGGCTCGGTTCACTTATTTACAATGGGCACGACGGTACTTGAGATCCAGTGAATTACTGGAGTAGTCTAGGAGCAACTAGAGGAGAGTTCAACACTTGTCCCATAAAT
**************************************************...............((.............(((.(((((.((............(((((.((((((...((((....((((((........))))))......)))).....))))))..))))))))))))....)))..........))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060663 160_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
V053 head |
SRR060659 Argentina_testes_total |
|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................AAAAAATTTCCGTAATACTAT....................................................................................................................................... | 21 | 2 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........................CCTCTGTTAGTCTGCGAATT............................................................................................................................................................................................................. | 20 | 3 | 7 | 0.14 | 1 | 0 | 0 | 1 | 0 | 0 | 0 |
| ....................................................................................................................................................................TATTGGCACGACGGTCCTTGA.................................................................. | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 | 0 | 0 |
| ................................................................................................................................................................................................TGAATTACTGAAGTACTC......................................... | 18 | 2 | 11 | 0.09 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| .........................................................................................................................................................................................................GGAGTAGGTTAGGAGAAAC............................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..............................................................................................................................................................ATGTACTATGGGCAGGACG.......................................................................... | 19 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................GGATCATGATGAGAGCGG............................................................................................................................... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:12876807-12877057 + | dvi_11561 | ATCGTTAAAGTTGCACCATGATGGACGGAGTCAACCAGATGCTTAACA----GTG----------TGTGTGTGTGTGTGTCTGTAATAATCAGTTT------------------------------AATCATGTGCAATTTTTTTAAAGGCATTGTAATACTCTCGCCGAGCGGGTACTATGTCTTGCCCGAGCCAAGTGAATAAATGTTACCCGTGCTGCCATGAACTCTAGGTCACTTAATGACCTCATCAGATCCTCGTTGATCTCCTCTCAAGTTGTGAACAGGGTATTTA |
| droMoj3 | scaffold_6680:3654892-3654957 - | GTGT--------------------------------------------------------------GTGTGTGTGTCTGTTTGTTTGTATTAGCTTATTGGTGTGCGTGTTGTT--------------------------TGTTGATAAGCATT--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droGri2 | scaffold_14853:1613850-1613960 - | AGGAAGAAAATTTCACTATTG----TT---TGAA-------CTTGAGT----GTGTGTTACTGTGTGTGTGTGTGTGTGT------------------------------TGTTAAAAGAAAGTTATATCAAGTGCATATCTGATATA-GTATTATAGTA--------------------------------------------------------------------------------------------------------------------------------------- | |
| droWil2 | scf2_1100000004513:182179-182245 + | GTGT--------------------------------------------------------------GTGTGTGTGTGTGTGTGTGTGAGGAGGGTCAACCAAGTGGAAAATGT-------------------------TCTTTTCAAGTGCACT--------------------------------------------------------------------------------------------------------------------------------------------- | |
| dp5 | 4_group3:8415894-8415953 - | TTCGCTGGAGGTGAAG--CGACAAACC---TGAACCTGAAGCTCGACA----GGA----------A------------------------------ATTAGTGCGC---------------------------------------------GCG--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droPer2 | scaffold_1:9864851-9864910 - | TTCGCTGGAGGTGAAG--CGACAAACC---TGAACCTGAAGCTCGACA----GGA----------A------------------------------ATTAGTGCGC---------------------------------------------GCG--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droBip1 | scf7180000396543:372543-372603 + | AATC--------------------------------GTGTGCCTGCTTGTTTGTG----------TGTGTGTGTGTGTG----TGTGTGTCAGTGT------------------------------GTTTGT-----------------GTGTA--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droRho1 | scf7180000779911:576826-576857 - | TG---------------------------------------CTTATGG----GTA----------TGTGTGTGTGTGTG------------------------TGT---------------------------------------------GTA--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droEug1 | scf7180000408958:37980-38009 - | TG---------------------------------------CTTACCT----GTG----------TGTGTGTGTGTG--------------------------TGC---------------------------------------------GTG--------------------------------------------------------------------------------------------------------------------------------------------- | |
| droSec2 | scaffold_2:6659811-6659814 - | GCCG--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
Generated: 05/19/2015 at 04:59 PM