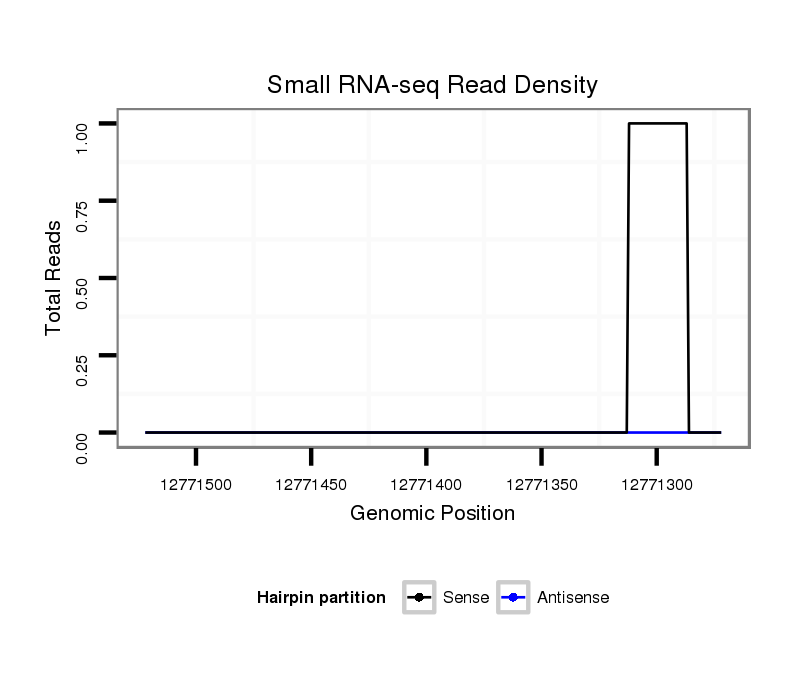
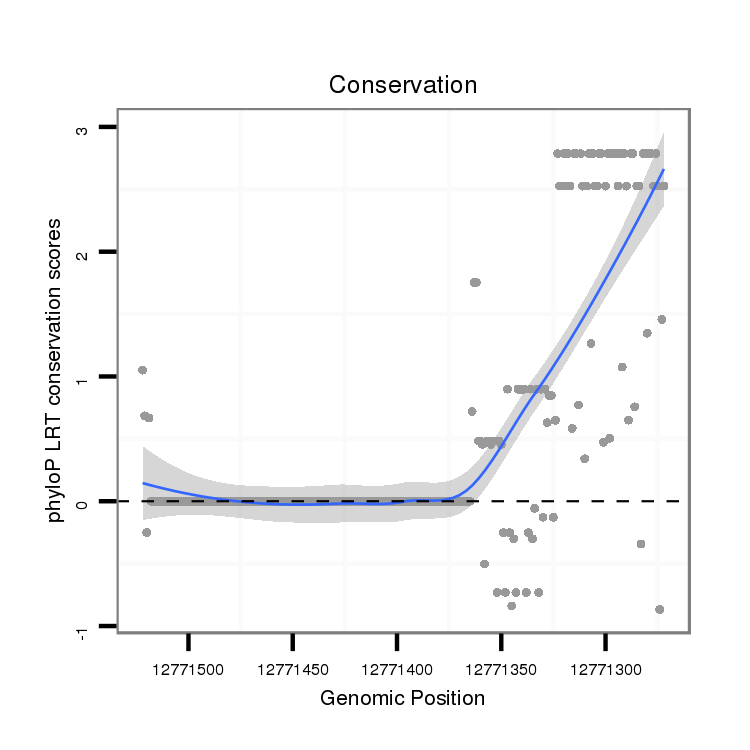
ID:dvi_11529 |
Coordinate:scaffold_12963:12771322-12771472 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_820:12]; CDS [Dvir\GJ22831-cds]; intron [Dvir\GJ22831-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TTCGTATTCAAGGGTACTTAATCTTCTGTGTGTTCAACTTTGTGACATGCTGTAGTTATGAATTCGAAAAGTTATTCCTAAATCTTTTAGTCGACACACAAACACTTAGAATAACTCTAATGCGCTTGTGGGATAAGATCTATGAGTACGATTTGACAATTTTCGTTGTTGTGATTTGTATTTTTTTTTTTTGTCAATCAGGTCACAATTTGCCACTGCAATGTCAATCAAACGTATCGCAAGTTCTCCAC **************************************************.............((((((((............))))...))))..(((((.((.(((((.....)))))))...))))).(((((((.....((((((((....(((((.......)))))...)))))))).....)))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060682 9x140_0-2h_embryos_total |
SRR060678 9x140_testes_total |
V116 male body |
SRR060662 9x160_0-2h_embryos_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR060681 Argx9_testes_total |
SRR060656 9x160_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060680 9xArg_testes_total |
M047 female body |
V047 embryo |
V053 head |
SRR060663 160_0-2h_embryos_total |
SRR060666 160_males_carcasses_total |
SRR060687 9_0-2h_embryos_total |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................................................................................GCGCTGGTGGGATACGACC............................................................................................................... | 19 | 3 | 7 | 2.00 | 14 | 6 | 0 | 3 | 2 | 1 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .........................................................................................................................GCGCTGGTGGGATAAGAC................................................................................................................ | 18 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................................................................................................................................................................................................................TGCCACTGCAATGTCAATCAAACGTA............... | 26 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................................................................................GGCGCTGGTGGGATACGA................................................................................................................. | 18 | 3 | 20 | 0.55 | 11 | 1 | 0 | 2 | 3 | 1 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .......................................................................................................................AGGCGCTGGTGGGATACGA................................................................................................................. | 19 | 3 | 16 | 0.31 | 5 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 1 | 0 | 0 |
| ........................................................................................................................TGCGCTGGTGGGATACGA................................................................................................................. | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................CCACAACCGCTTAGAATAACT....................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GTGATTGGTATTTTTTTT............................................................... | 18 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GTGATTGGTATTCTTTTTTT............................................................. | 20 | 2 | 12 | 0.08 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................CTTGCCACTGCAATGTCATA....................... | 20 | 3 | 12 | 0.08 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GTGATTTGGATTCTTTTTTT............................................................. | 20 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .....................................................................................................................TGAGGCGCTTGTGGGATAC................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..CGGATTCAAGCGGACTTAA...................................................................................................................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................................................................................................................................GTGATTGGTATTTTCTTTT.............................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................................................................................................................GTGATTGGTATTTTATTTT.............................................................. | 19 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
|
AAGCATAAGTTCCCATGAATTAGAAGACACACAAGTTGAAACACTGTACGACATCAATACTTAAGCTTTTCAATAAGGATTTAGAAAATCAGCTGTGTGTTTGTGAATCTTATTGAGATTACGCGAACACCCTATTCTAGATACTCATGCTAAACTGTTAAAAGCAACAACACTAAACATAAAAAAAAAAAACAGTTAGTCCAGTGTTAAACGGTGACGTTACAGTTAGTTTGCATAGCGTTCAAGAGGTG
**************************************************.............((((((((............))))...))))..(((((.((.(((((.....)))))))...))))).(((((((.....((((((((....(((((.......)))))...)))))))).....)))))))......************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V047 embryo |
SRR060681 Argx9_testes_total |
V053 head |
SRR060682 9x140_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060665 9_females_carcasses_total |
SRR060679 140x9_testes_total |
M027 male body |
GSM1528803 follicle cells |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ........................................................................ATTAGGATTTAGAAGATCAGC.............................................................................................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................GTTGGTATGCATCGCGTTCAA...... | 21 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................AACATAAAAAAAAAAAACAGTA...................................................... | 22 | 1 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................AGGGATTTAAAAAATCCGCTGT........................................................................................................................................................... | 22 | 3 | 4 | 0.25 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................................................................TAGGATTTAGAAGATCAG............................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..........TCCCATGGATTAGTAGAAAC............................................................................................................................................................................................................................. | 20 | 3 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................AAAAAAAAACAGGTCGTCCA................................................ | 20 | 2 | 7 | 0.14 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| .....................................................................TCAATAAGGGTTTAGTAAACC................................................................................................................................................................. | 21 | 3 | 9 | 0.11 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ...............................................................AGATTTTCAATAAGGAGT.......................................................................................................................................................................... | 18 | 2 | 11 | 0.09 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ..............................ACAAGTGGAAACTCTGGACGAC....................................................................................................................................................................................................... | 22 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ..............................ACAAGTTGAACCTCTGGACGAC....................................................................................................................................................................................................... | 22 | 3 | 19 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..............................ACAAGTTGAACCTCTGGACG......................................................................................................................................................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ..........................................................CGTTAAGCTTTTCAATAA............................................................................................................................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................................................................TAGTGCAGTCTTAAACGG..................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| .............................................................TAAGCTTTTCAATAATGCTTA......................................................................................................................................................................... | 21 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:12771272-12771522 - | dvi_11529 | TTCGTATTCAAGGGTACTTAATCTTCTGTGTGTTCAACTTTGTGACATGCTGTAGTTATGAATTCGAAAAGTTATTCCTAAATCTTTTAGTCGACACACAAACACTTAGAATAACTCTAATGCGCTTGTGGGATAAGATCTATGAGTACGATTTGACAATTTTCGTTGTTGTGAT----------------TTGTATTTTTTTTT----------TTTGTCAATCAGGTCACAATTTGCCACTGCAATGTCAATCAAACGTATCGCAAGTTCTCCAC |
| droMoj3 | scaffold_6500:7292807-7292909 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATTTTCATTGTTTTGGCTGTTT-----TCTCT-TCTCGTTTTCCTCG----------TCTGTCAACTAGGTCACAATATGTCACTGCAACGTGAATCAAACGTACCGTAAGTTCTCGAC | |
| droGri2 | scaffold_14978:897103-897159 + | ----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAACCAGGTCACTATTTGTCATTGCAACGTGAATCAGACATACCGCAAGTTCTCGAC | |
| droWil2 | scf2_1100000004521:11807344-11807451 - | TTTG-------------------------------------------------------------------------------------------------------------------------------------------------------------------------GAAAATTTCGTTTCTCTTTCATTTTTTACTTGTGCGTGGGTGTATTTGAAACAGGTCACCATCTGTCACTGCAAGGTCAATCAGACGTATCGAAAATTCTCCGC | |
| dp5 | 4_group3:8590367-8590420 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------T-----------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCCAC | |
| droPer2 | scaffold_1:10044351-10044404 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------T-----------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCCAC | |
| droAna3 | scaffold_12916:15730868-15730920 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGAAAATTCTCGAC | |
| droBip1 | scf7180000396769:20702-20754 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droKik1 | scf7180000302408:51455-51522 + | T-------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTT-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droFic1 | scf7180000453949:281804-281856 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTCACCATTTGCCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droEle1 | scf7180000491273:1488503-1488569 - | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droRho1 | scf7180000770100:728-794 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCCCTC-------------C---------------GTTGTAGGTCACCATTTGCCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droBia1 | scf7180000302408:46927-46993 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCAAC | |
| droTak1 | scf7180000415779:295075-295141 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| droEug1 | scf7180000407253:54525-54591 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTT-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCGAC | |
| dm3 | chr2L:1856413-1856479 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACTTATCGAAAATTCTCCAC | |
| droSim2 | 2l:1772408-1772474 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCCAC | |
| droSec2 | scaffold_14:1803171-1803237 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------ATT------------------------TCTCTC-------------C---------------GTTGCAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCCAC | |
| droYak3 | 2L:1830411-1830463 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCAAC | |
| droEre2 | scaffold_4929:1903291-1903343 + | --------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CAGGTCACCATTTGTCACTGCAAGGTCAATCAGACGTATCGGAAATTCTCAAC |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dm3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSim2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droSec2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droYak3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEre2 |
|
Generated: 05/19/2015 at 04:41 PM