
ID:dvi_11451 |
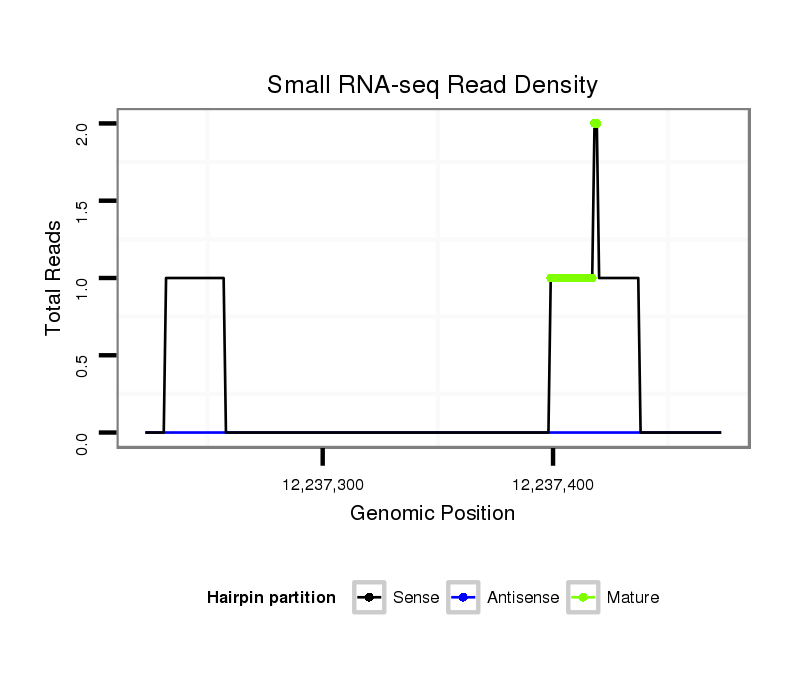
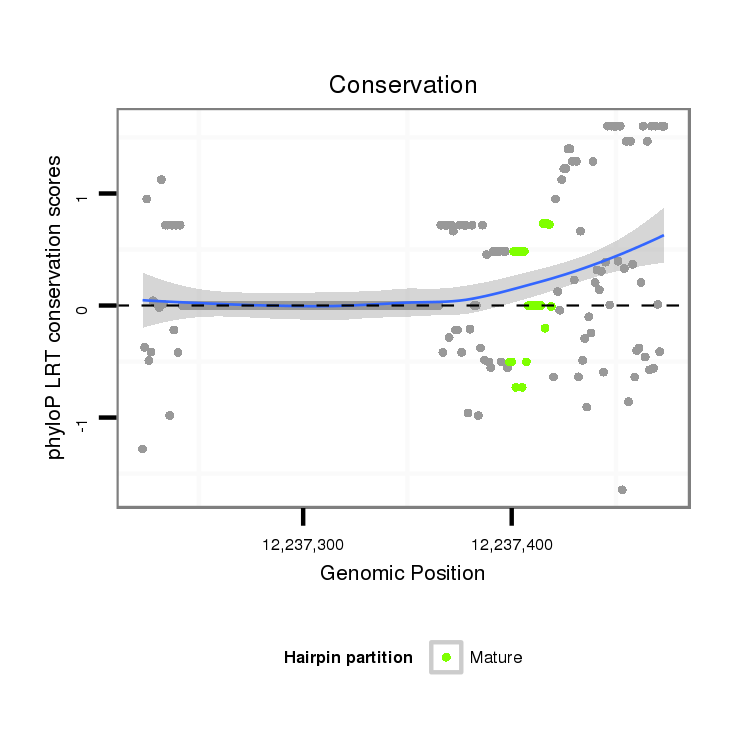
Coordinate:scaffold_12963:12237273-12237423 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -25.9 | -25.9 | -25.9 |
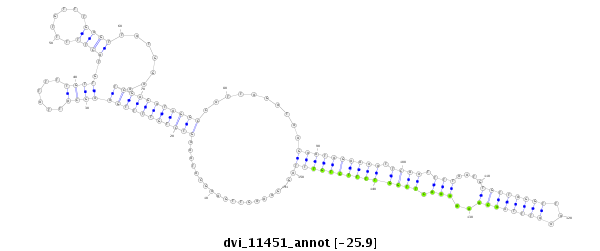 |
 |
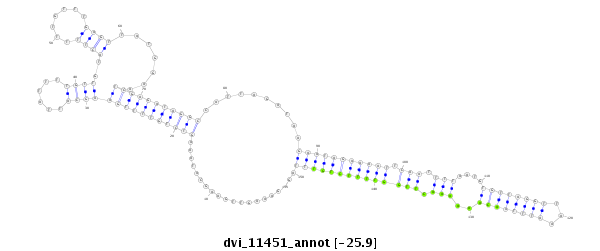 |
exon [dvir_GLEANR_2004:2]; CDS [Dvir\GJ17408-cds]; intron [Dvir\GJ17408-in]
No Repeatable elements found
| ---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------################################################## TCAACATGTTCAACATAAAGCTGTCTGTGGTAAGTTGTCATAAATCTTATGAAAGTTGAAGCATAAAGTCTGTTTTGAAGCATTATTTTGTTGTACTTTTTGTTTGAGTTATGGAATCGAGATAGGCCATTAGATAAGAATAGAAAATTCAATTTTAATGTGTTAGGTTAAATTTAACAAAAAACTTGGTTTTCTGTTTAGAAAATGGCAGGCAACGATGTTCTGATTATTTCACCAACGAACGTCAAGTT **************************************************.................(((((((((((..(((.((((.................)))).)))...)))))))))))..........(((((((((((.(((.(((....((((((((...))))))))..))).))))))))))))))..************************************************** |
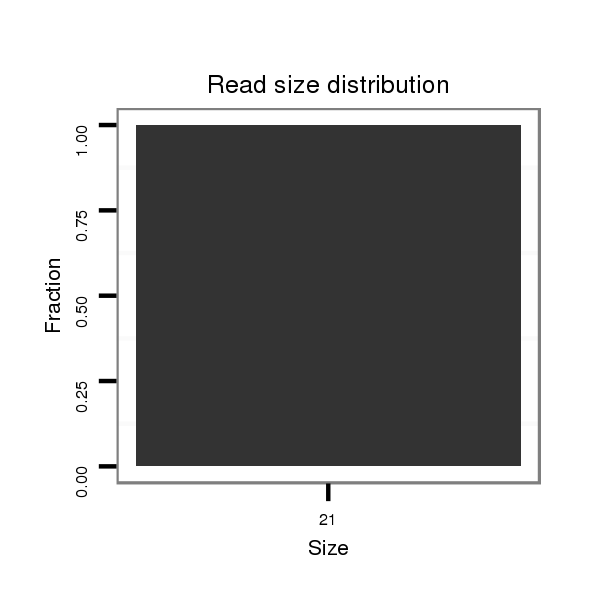
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
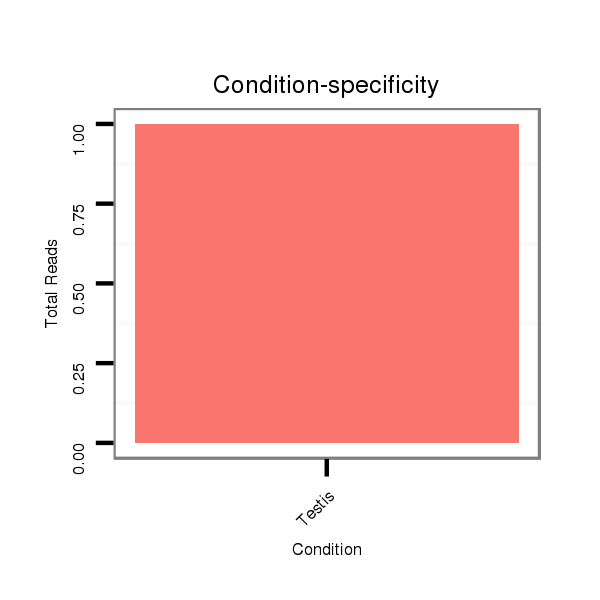
SRR060681 Argx9_testes_total |
SRR060654 160x9_ovaries_total |
SRR060659 Argentina_testes_total |
SRR060672 9x160_females_carcasses_total |
V047 embryo |
M047 female body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................................................................................................................................................................ACAAAAAACTTGGTTTTCTGT...................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................TATGGAATCGAGATAGACC........................................................................................................................... | 19 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .........TCAACATAAAGCTGTCTGTGGTAAGT........................................................................................................................................................................................................................ | 26 | 0 | 1 | 1.00 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ........................................................................................................TGAGTTAGGGAATCGAGAC................................................................................................................................ | 19 | 2 | 1 | 1.00 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ............................................................................................................................................................................................................................TTCTGATTATTTCACCAGCG........... | 20 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................................................................GATTGTTTCACCAACGAACGA...... | 21 | 2 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................................TGGCAGGCAACGACGTTCGGAT........................ | 22 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................................................................................................................................................................................GTTTAGAAAATGGCAGGCAA.................................... | 20 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ........................................................TGAAGCATCTAGTCTGTTTTG.............................................................................................................................................................................. | 21 | 2 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................TGAAGCATATAGTCTGTATTGC............................................................................................................................................................................. | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................TAAAGCATATAGGCTGTTTTG.............................................................................................................................................................................. | 21 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ........................................................TGGAGCATAGAGGCTGTTTT............................................................................................................................................................................... | 20 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................TCTGTGGTAAATTTTCATAAAA.............................................................................................................................................................................................................. | 22 | 3 | 8 | 0.13 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
AGTTGTACAAGTTGTATTTCGACAGACACCATTCAACAGTATTTAGAATACTTTCAACTTCGTATTTCAGACAAAACTTCGTAATAAAACAACATGAAAAACAAACTCAATACCTTAGCTCTATCCGGTAATCTATTCTTATCTTTTAAGTTAAAATTACACAATCCAATTTAAATTGTTTTTTGAACCAAAAGACAAATCTTTTACCGTCCGTTGCTACAAGACTAATAAAGTGGTTGCTTGCAGTTCAA
**************************************************.................(((((((((((..(((.((((.................)))).)))...)))))))))))..........(((((((((((.(((.(((....((((((((...))))))))..))).))))))))))))))..************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | V053 head |
SRR060670 9_testes_total |
SRR060688 160_ovaries_total |
SRR060681 Argx9_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| ..........GTTGTGTTTCGACCGACACTAT........................................................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| ..........GTGGTGTTTCGACAGACACTAT........................................................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 0 | 1 | 0 | 0 |
| ..........GAGGTATTTCGACAGACACTAT........................................................................................................................................................................................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 |
| ..........GATGATTTTCGACAGACACCAT........................................................................................................................................................................................................................... | 22 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 |
| ..........GTTGAGTTTCGACAGACACTAT........................................................................................................................................................................................................................... | 22 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 |
| ...........TTGTCTATCGACAGACACAAT........................................................................................................................................................................................................................... | 21 | 3 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 |
| ...........AGGTATTTCGACAGACACTAT........................................................................................................................................................................................................................... | 21 | 3 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| ...............................................................ATTTCAGACAACACTTTGT......................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 |
| ............TGTAGTTCGACCGACACTAT........................................................................................................................................................................................................................... | 20 | 3 | 10 | 0.10 | 1 | 0 | 0 | 0 | 1 |
| ............TGTCTTTCGACCGACACTAT........................................................................................................................................................................................................................... | 20 | 3 | 13 | 0.08 | 1 | 1 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:12237223-12237473 + | dvi_11451 | TCAACATGTTCAACATAA----------------AGCTGTCTGTGGTAAGTTGTCATAAATCTTATGAAAGTTGAAGCATAAAGTCTGTTTTGAAGCATTATTTTGTTGTACTTTTTGTTTGAGTTATGGAATCGAGATAGGCCATTAGATAAGAATAGAAAATTCAATTTTAATGTGTTAGGTTAAATTTAAC---AAAAAACTTGGTTTTCTGTTTAGAAAATGGCAGGCAACGATGTTCTGATTATTTCACCAACGAACGTCAAGTT |
| droMoj3 | scaffold_6500:20956783-20956899 + | AAATTCAATTTAATATAT----------------A----------------------------------------------------------------------------------------------------------------------------ATAACTCAATATTTAT--ATTCGAATAAACTTTGTAATACAACAT-----------TGTAGAAAATGGCAGGCCACGACGTCCTGATTGTGTCGCCGTTGAACGTAAAGTT | |
| droGri2 | scaffold_15126:5753904-5754007 + | GT---CAAGTTAAAAAATTCGACAAATCAGTAAAA----------------------------------------------------------------------------------------------------------------------------ATAACTCTTTATTGGT--TGTC-------------------------------------AGAAAATGGCTGCAAAAGATGTACTGATTGTATCGCAAACAAACATGAAGTT | |
| droKik1 | scf7180000302271:374014-374059 - | AA-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ATGTCT---GGCGATCAGTTAATTGTTTCTCCCTTAAGCCTGAGCTT | |
| droEle1 | scf7180000491028:452069-452135 - | AAATTCAATTT--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------TTTGATTTCAAAATGTCT---AACGTTCAGCTAATTGTAACACCTTTAATCCTGAGTTT | |
| droRho1 | scf7180000778052:17285-17332 - | GA---------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------AAATGTCA---GGAGTTCAGCTAATTGTTACACCTTTAACCCTGAGTTT | |
| droBia1 | scf7180000302408:1742722-1742750 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTCATTGTAACGCCCACAACCGTGAGTTT | |
| droTak1 | scf7180000415399:434056-434084 - | -------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------CTAATTGTTACACCCACAACCCTAAACTT |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
Generated: 05/17/2015 at 04:26 AM