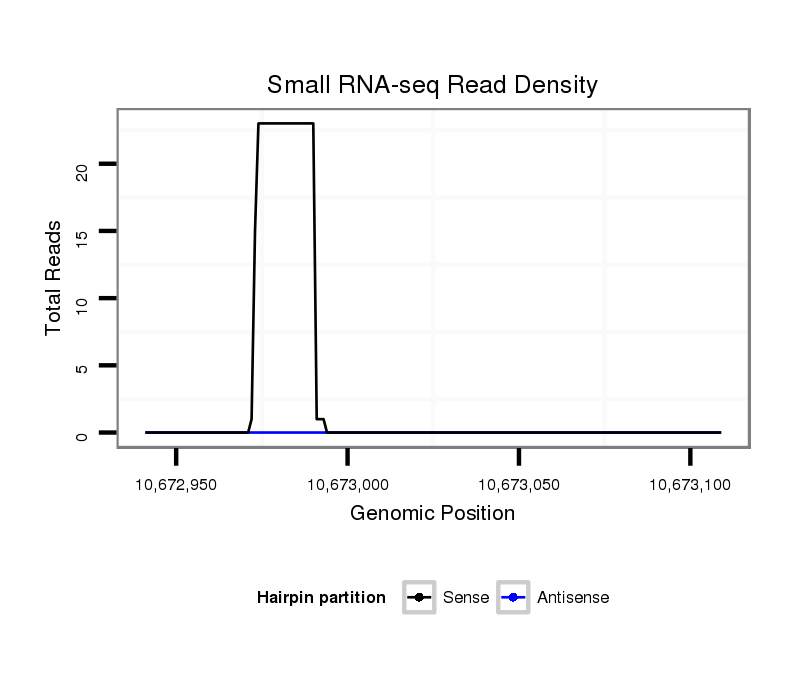
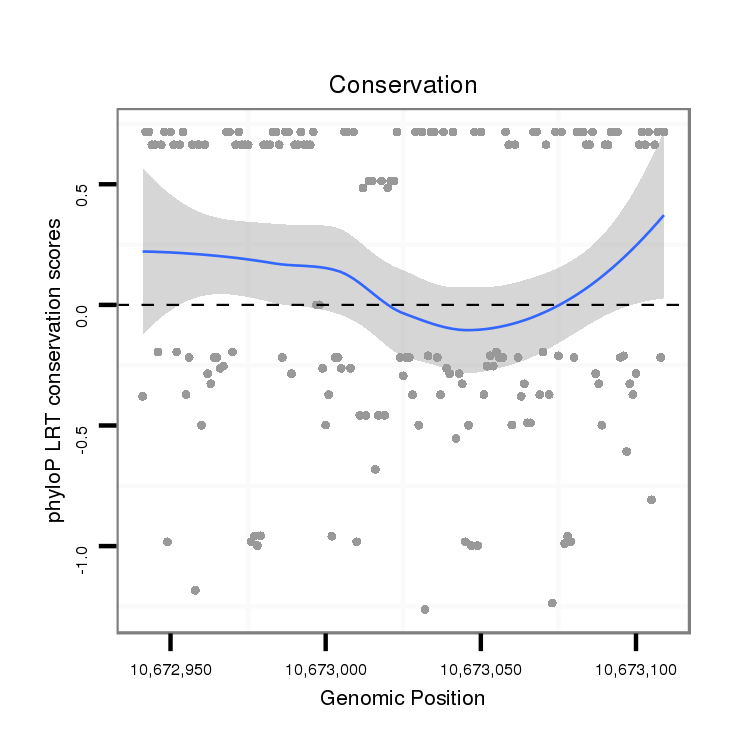
ID:dvi_11284 |
Coordinate:scaffold_12963:10672991-10673059 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_1937:7]; CDS [Dvir\GJ17340-cds]; CDS [Dvir\GJ17340-cds]; exon [dvir_GLEANR_1937:6]; intron [Dvir\GJ17340-in]
No Repeatable elements found
| ##################################################---------------------------------------------------------------------################################################## AATCCGGTCACCCTGACACAGACCGAATACCTGCCCAGACGCATGGAATGGTGGGTGTTTGTCGTTAATCGCCAACTAGGATATCAACAATGTATACAATTTACCACACTCTTTCGCAGTGCTGCAAAGCGCGTTAAAGCTTTGGAAGAGGTAACTGGCTCAGTGCTGA **************************************************(((((((.((((((((.....((.....))........))))...))))..)))).)))..........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR1106718 embryo_6-8h |
SRR1106723 embryo_12-14h |
SRR1106724 embryo_12-14h |
SRR1106730 embryo_16-30h |
SRR1106715 embryo_4-6h |
SRR1106720 embryo_8-10h |
SRR1106721 embryo_10-12h |
M061 embryo |
SRR060678 9x140_testes_total |
SRR1106716 embryo_4-6h |
SRR1106722 embryo_10-12h |
SRR1106725 embryo_14-16h |
SRR1106726 embryo_14-16h |
V053 head |
SRR060687 9_0-2h_embryos_total |
M047 female body |
SRR060689 160x9_testes_total |
SRR1106729 mixed whole adult body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ................................GCCCAGACGCATGGAATG....................................................................................................................... | 18 | 0 | 1 | 14.00 | 14 | 3 | 3 | 2 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 1 | 0 | 0 | 0 | 0 | 0 |
| .................................CCCAGACGCATGGAATG....................................................................................................................... | 17 | 0 | 1 | 8.00 | 8 | 0 | 0 | 1 | 2 | 1 | 1 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................TGCCCAGACGCATGGAATGGTG.................................................................................................................... | 22 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...........................................TGGAATGGTGGGTGGTTGTATT........................................................................................................ | 22 | 3 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................GGCCAGACGCATGGAATG....................................................................................................................... | 18 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................................................AGGTAGCGGGCTTAGTGCTG. | 20 | 3 | 3 | 0.33 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .........................................................................................................................................AGGCTTGGAAGAGGTACCT............. | 19 | 3 | 14 | 0.07 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
| ...................................................................ATAGCCAACTAGGATGT..................................................................................... | 17 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ................................................TGTTGGGTGTTTGTGGTTA...................................................................................................... | 19 | 2 | 16 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
|
TTAGGCCAGTGGGACTGTGTCTGGCTTATGGACGGGTCTGCGTACCTTACCACCCACAAACAGCAATTAGCGGTTGATCCTATAGTTGTTACATATGTTAAATGGTGTGAGAAAGCGTCACGACGTTTCGCGCAATTTCGAAACCTTCTCCATTGACCGAGTCACGACT
**************************************************(((((((.((((((((.....((.....))........))))...))))..)))).)))..........************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M028 head |
SRR060664 9_males_carcasses_total |
SRR060672 9x160_females_carcasses_total |
V047 embryo |
|---|---|---|---|---|---|---|---|---|---|
| ......................................................................CGGTTGATTCCATAGTTG................................................................................. | 18 | 2 | 3 | 0.67 | 2 | 2 | 0 | 0 | 0 |
| ...........................................................................................................................................GAAGTCATCTCCATTGACC........... | 19 | 3 | 10 | 0.10 | 1 | 0 | 1 | 0 | 0 |
| ............................................................................................................................................AAGTCATCTCCATTGACC........... | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 1 | 0 |
| ......CAGTGGGACTGTTGCAGG................................................................................................................................................. | 18 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:10672941-10673109 + | dvi_11284 | AATCCGGTCACCCTGACACAGACCGAATACCTGCCCAGACGCATGGAATGGTGGGTGTTTGTCGTTAATCGCCAACTA-GGATATCAACAATGTATACAATTTACCACA-CTCTTTCGCAGTGCTGCAAAGCGCGTTAAAGCTTTGGAAGAGGTAACTGGCTCAGTGCTGA |
| droMoj3 | scaffold_6500:19387715-19387869 + | AATCCGGTTACCCTAACCCCGGATATGTACCTGCCTTTGCGCATGAAACGGTGGGT--AGAATAATATTT-------------ATCAATACTCTATATATCTAGATCAA-ATTCTCCATAGGGTTTCCAAACGTCTTATTATTTTGGAGTCGGTAATTCTTCCAGTCCTAA | |
| droGri2 | scaffold_15252:8704766-8704933 + | CATCCAGTAACTCTATCGCAGGCCGAATATCTGCCAGATCGCATGGAACGGTGGGT--TTACCGTTAATAACTAAACACAGATAAATTTAATTCATTTAACT-GCAATATTTTTCTTGCAGTGCGGGCAAATGTATCACGTCTTTGGAGGAGGTAACCGGTCCAGTCCTGA |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/16/2015 at 10:12 PM