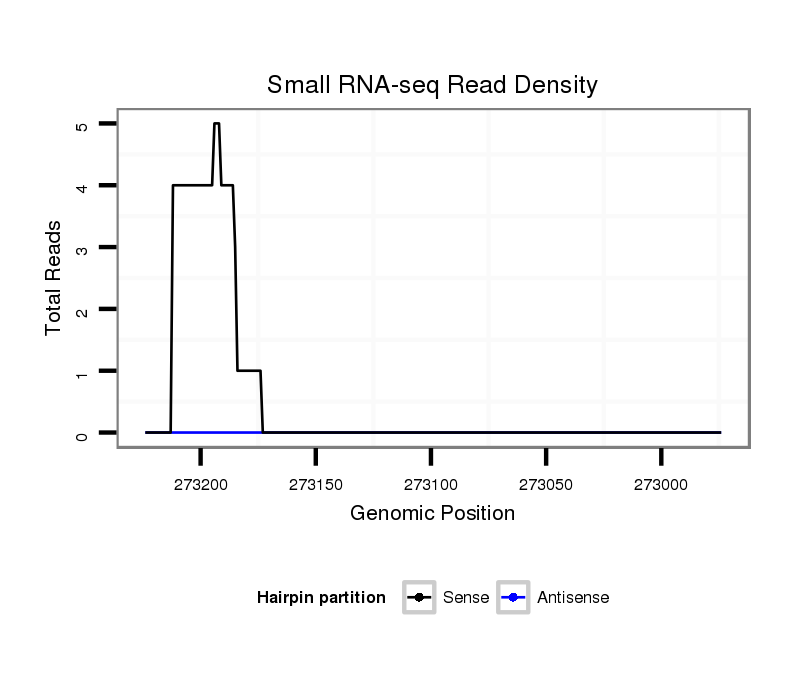
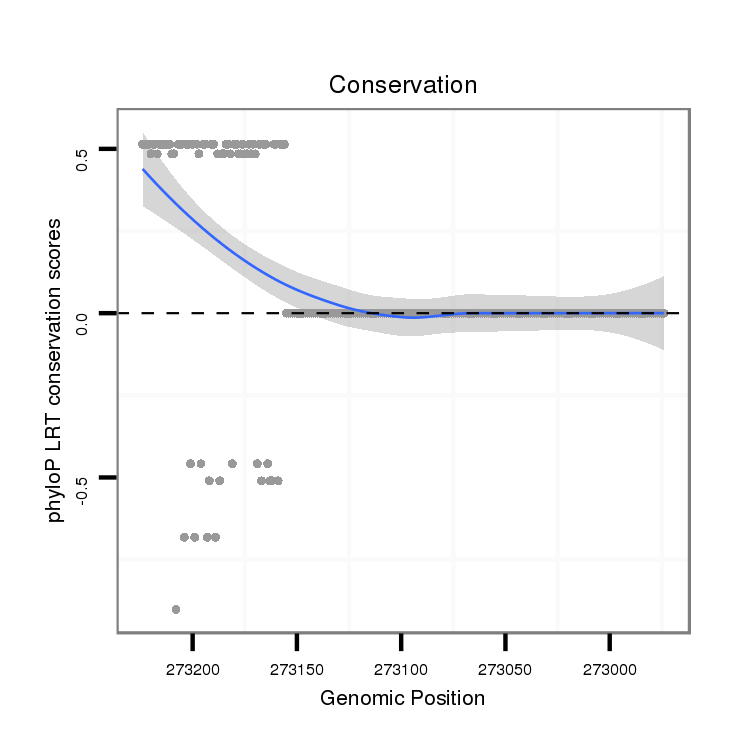
ID:dvi_1125 |
Coordinate:scaffold_12723:273024-273174 - |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
exon [dvir_GLEANR_2667:1]; CDS [Dvir\GJ18108-cds]; intron [Dvir\GJ18108-in]
No Repeatable elements found
| ##################################################--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- AATTCTTCATATTTGGGATACTTATATCTTTCATTTGTGCATCGAAGCAGGTGAGTTATAGATTATATAGCTTGGCAACATGTTTCTGTAATTTCTTTTTCACTGACTGCGCATACTATGATCTCAGCGTAATCACAGCTTTGGTCAATCGAAATGTTTTATGATAATTTTAAGTAATACCAACTTCGGGAACCCACGAGCATCAACGGCTATTACATATGTAAAGTCAAGCAATTTGCTTGTGTTTGACC **************************************************(((((....((((((((.(((.((....)).)))..))))))))....)))))(((.(((((..............))))).)))..(((((((....(((((..((.((((...........)))).))....)))))....))).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | M047 female body |
SRR060678 9x140_testes_total |
SRR060683 160_testes_total |
SRR060674 9x140_ovaries_total |
SRR060675 140x9_ovaries_total |
SRR060676 9xArg_ovaries_total |
SRR060681 Argx9_testes_total |
GSM1528803 follicle cells |
V053 head |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ............TTGGGATACTTATATCTTTCATTTGTGC................................................................................................................................................................................................................... | 28 | 0 | 1 | 2.00 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 |
| ..............................TCATTTGTGCATCGAAGCAGGATTG.................................................................................................................................................................................................... | 25 | 3 | 1 | 2.00 | 2 | 0 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................CATTTGTGCATCGAAGCAGGATT..................................................................................................................................................................................................... | 23 | 3 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............TTGGGATACTTATATCTTTCA.......................................................................................................................................................................................................................... | 21 | 0 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| ..............................TCATTTGTGCATCGAAGCAGG........................................................................................................................................................................................................ | 21 | 0 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..................TACTTATATCTTTCATTTGCGCATCGAA............................................................................................................................................................................................................. | 28 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............TTGGGATACTTATATCTTTCATTTGTG.................................................................................................................................................................................................................... | 27 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ............TTGGGTTACTTATATCTTTCATTTGTG.................................................................................................................................................................................................................... | 27 | 1 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..........................TCTTTCATTTGTGCATCGAAGCAGGA....................................................................................................................................................................................................... | 26 | 1 | 1 | 1.00 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ............................................................................................................................................................................................GGAGCCCCCGAGCACCAACG........................................... | 20 | 3 | 3 | 0.33 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................TGTGCATCGAAGCAGGATTGT................................................................................................................................................................................................... | 21 | 3 | 3 | 0.33 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...................................TGTGCATCGAAGCAGGAT...................................................................................................................................................................................................... | 18 | 2 | 4 | 0.25 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| .....................................................................................................AGAGACTGCGCATACGATG................................................................................................................................... | 19 | 3 | 18 | 0.06 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
|
TTAAGAAGTATAAACCCTATGAATATAGAAAGTAAACACGTAGCTTCGTCCACTCAATATCTAATATATCGAACCGTTGTACAAAGACATTAAAGAAAAAGTGACTGACGCGTATGATACTAGAGTCGCATTAGTGTCGAAACCAGTTAGCTTTACAAAATACTATTAAAATTCATTATGGTTGAAGCCCTTGGGTGCTCGTAGTTGCCGATAATGTATACATTTCAGTTCGTTAAACGAACACAAACTGG
**************************************************(((((....((((((((.(((.((....)).)))..))))))))....)))))(((.(((((..............))))).)))..(((((((....(((((..((.((((...........)))).))....)))))....))).))))************************************************** |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060684 140x9_0-2h_embryos_total |
SRR060687 9_0-2h_embryos_total |
SRR060661 160x9_0-2h_embryos_total |
SRR060657 140_testes_total |
SRR060686 Argx9_0-2h_embryos_total |
SRR1106713 embryo_0-2h |
SRR1106719 embryo_8-10h |
SRR1106726 embryo_14-16h |
SRR1106727 larvae |
SRR1106728 larvae |
SRR1106729 mixed whole adult body |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ...............................................................................................................................................................................TTGTGGTTGAAGCCTTTGGTTG...................................................... | 22 | 3 | 2 | 0.50 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .......................................................................................................................................................................................GGTGCCCTTGGGTGC..................................................... | 15 | 2 | 20 | 0.30 | 6 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 0 |
| .............................................................................................................................................................................................CTTGGGTCTTCGTAGTTG............................................ | 18 | 2 | 9 | 0.11 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| .............................................................................................................................................................................................CTTGGGTGGTCGTAGTAG............................................ | 18 | 2 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ..............................................................................GTACAAGGACATCGAAGAAA......................................................................................................................................................... | 20 | 3 | 11 | 0.09 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................TCGAACCGTTGTATGAAG..................................................................................................................................................................... | 18 | 2 | 14 | 0.07 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...............................................................................................................................................................................................AGGGTGCTCGTAGTTTCAG......................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| ................................................................................................................................................................................TGTGGTTGAAGCCTTTGGT........................................................ | 19 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ..................................................................................................AAGTGACTGACGCACATAA...................................................................................................................................... | 19 | 3 | 20 | 0.05 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12723:272974-273224 - | dvi_1125 | AATTCTTCATATTTGGGATACTTATATCTTTCATTTGTGCATCGAAGCAGGTGAGTTATAGATTATATAGCTTGGCAACATGTTTCTGTAATTTCTTTTTCACTGACTGCGCATACTATGATCTCAGCGTAATCACAGCTTTGGTCAATCGAAATGTTTTATGATAATTTTAAGTAATACCAACTTCGGGAACCCACGAGCATCAACGGCTATTACATATGTAAAGTCAAGCAATTTGCTTGTGTTTGACC |
| droGri2 | scaffold_15126:1574581-1574649 - | AATTCTTCATATTTGGCATAATTGTCTCCTTATTTGGAGCATCAAAGCAGGTGAGCTTTAATATAAATA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
Generated: 05/19/2015 at 11:06 AM