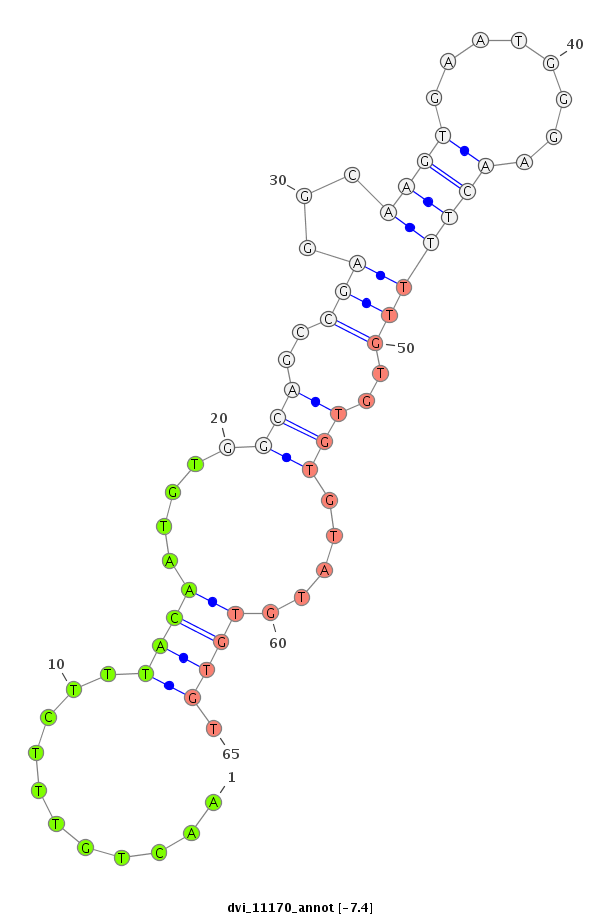
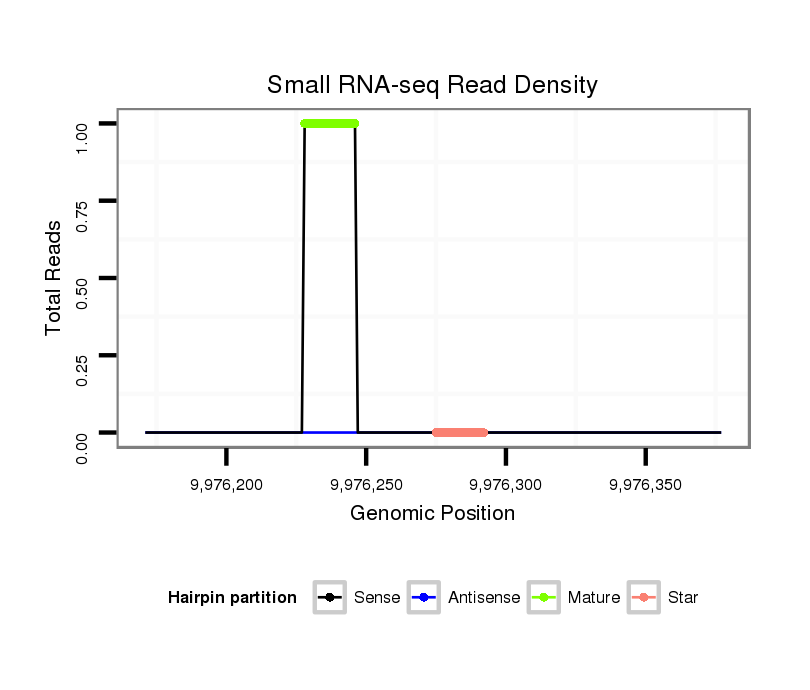
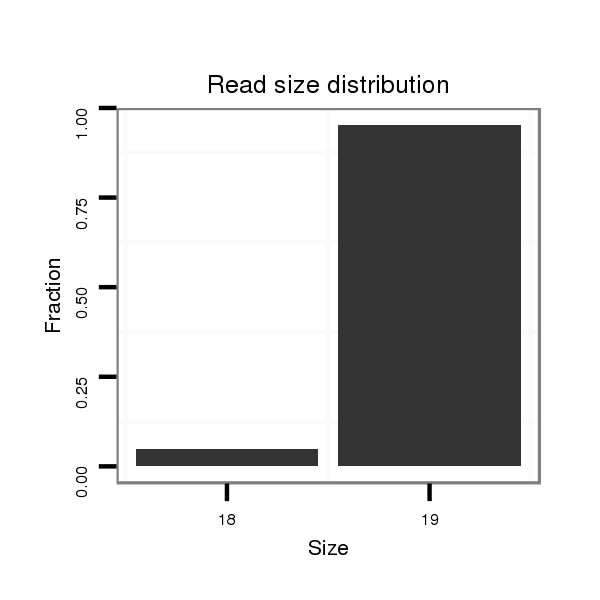
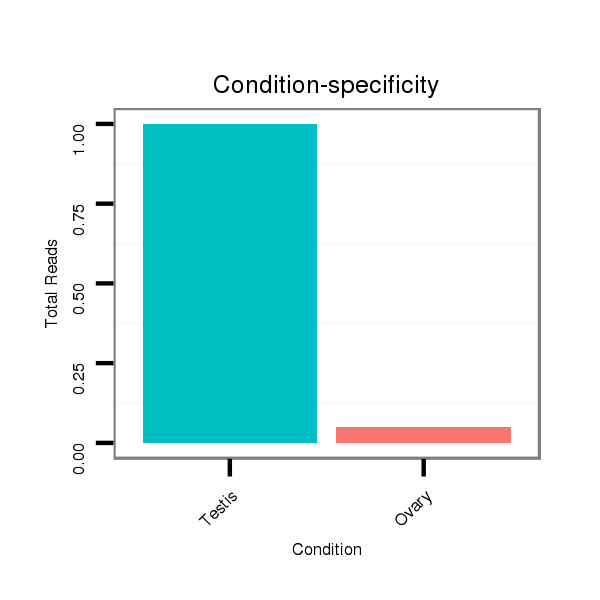
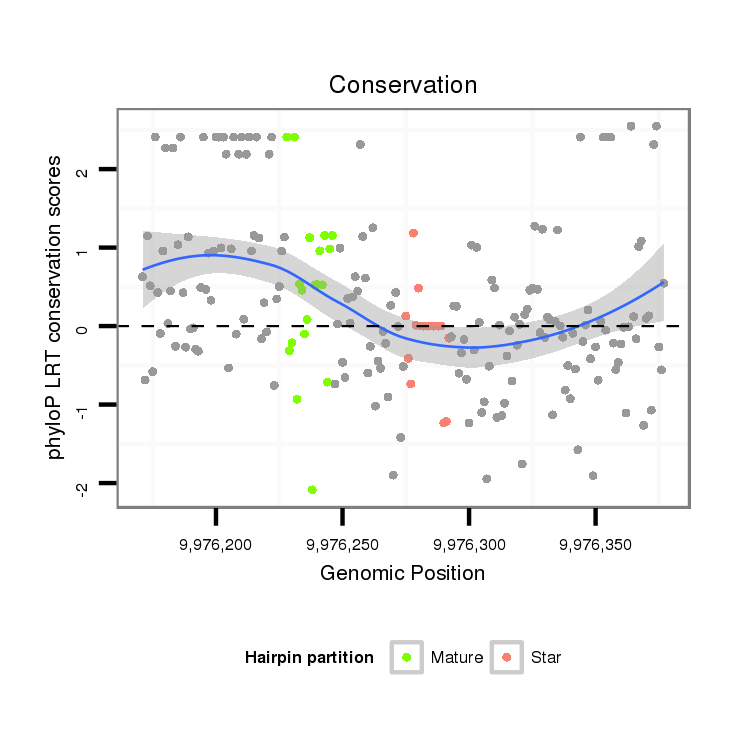
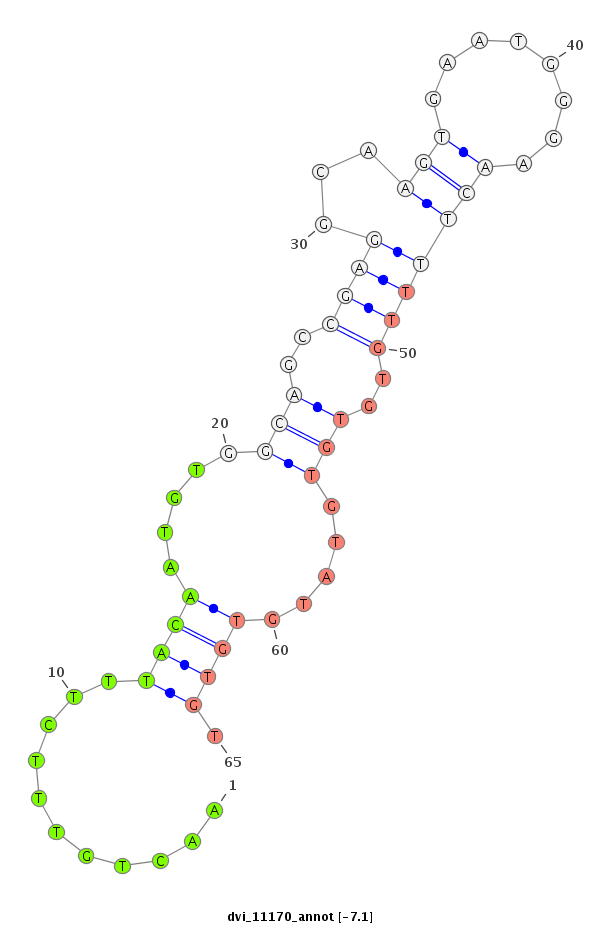
ID:dvi_11170 |
Coordinate:scaffold_12963:9976221-9976327 + |
Confidence:Candidate |
Type:Unknown |
[View on UCSC Genome Browser {Cornell Mirror}] |
| Legend: | mature | star | mismatch in alignment | mismatch in read |
 |
 |
 |
 |
 |
| -7.1 | -6.5 |
 |
 |
CDS [Dvir\GJ17296-cds]; exon [dvir_GLEANR_1897:1]; exon [dvir_GLEANR_1897:2]; CDS [Dvir\GJ17296-cds]; intron [Dvir\GJ17296-in]
No Repeatable elements found
| ##################################################-----------------------------------------------------------------------------------------------------------################################################## AGAATTATTACTTCAAGAAGAGCGTTGCCTACACGGAGGACCACATCCTGGTGAAGAAACTGTTTCTTTACAATGTGGCAGCCGAGGCAAGTGAATGGGAACTTTTGTGTGTGTATGTGTGTGTGGCTTGTCGTTATTTGAGCTGTTCTCTTTTCAGCTGTTGCAGCAGGAGGTGCAAACATATTTCGAGGGCTTTGGCCGTGTGCT *********************************************************...........((((.....(((..(((...((((........)))))))..))).....)))).************************************************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060681 Argx9_testes_total |
SRR060679 140x9_testes_total |
SRR060687 9_0-2h_embryos_total |
SRR060682 9x140_0-2h_embryos_total |
SRR060673 9_ovaries_total |
SRR060684 140x9_0-2h_embryos_total |
V116 male body |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| .........................................................AACTGTTTCTTTACAATGT................................................................................................................................... | 19 | 0 | 1 | 1.00 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 |
| ......................................................................................GCAAGTGAAGGGGAACTTTT..................................................................................................... | 20 | 1 | 1 | 1.00 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ......................CGATCCCTACAGGGAGGACCA.................................................................................................................................................................... | 21 | 3 | 2 | 0.50 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ................................................................................................................GTATGTGTGTGTTGCTTGTC........................................................................... | 20 | 1 | 4 | 0.25 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ...ATTATTACTACAAGATGAG......................................................................................................................................................................................... | 19 | 2 | 5 | 0.20 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 0 |
| ...........................................CAACCTGCTGAAGAAACT.................................................................................................................................................. | 18 | 2 | 8 | 0.13 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 |
| .............................................................................................................TGTGTATGTGTGTGTTGCTTG............................................................................. | 21 | 1 | 15 | 0.07 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| ....................................................................................................................GTTTGTGTGGATTGTCGT......................................................................... | 18 | 2 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 |
| ....................................................................................................................GTTTGTGTGGATTGTCGTGA....................................................................... | 20 | 3 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ........................................................................................................TTGTGTGTGTATGTGTGT..................................................................................... | 18 | 0 | 20 | 0.05 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 |
|
TCTTAATAATGAAGTTCTTCTCGCAACGGATGTGCCTCCTGGTGTAGGACCACTTCTTTGACAAAGAAATGTTACACCGTCGGCTCCGTTCACTTACCCTTGAAAACACACACATACACACACACCGAACAGCAATAAACTCGACAAGAGAAAAGTCGACAACGTCGTCCTCCACGTTTGTATAAAGCTCCCGAAACCGGCACACGA
*************************************************************************************...........((((.....(((..(((...((((........)))))))..))).....)))).********************************************************* |
Read size | # Mismatch | Hit Count | Total Norm | Total | SRR060686 Argx9_0-2h_embryos_total |
SRR060685 9xArg_0-2h_embryos_total |
SRR060667 160_females_carcasses_total |
SRR060680 9xArg_testes_total |
|---|---|---|---|---|---|---|---|---|---|
| ..........................................TGTAGGACAACTTGTTTG................................................................................................................................................... | 18 | 2 | 5 | 0.20 | 1 | 1 | 0 | 0 | 0 |
| .........................ACGTATGTGCGTCCTGGT.................................................................................................................................................................... | 18 | 2 | 6 | 0.17 | 1 | 0 | 1 | 0 | 0 |
| ................................................................................................................................ACGGCAATAATCTCCACAAG........................................................... | 20 | 3 | 9 | 0.11 | 1 | 0 | 0 | 1 | 0 |
| ..............................................................................................................CACATACACACACAACGAACA............................................................................ | 21 | 1 | 9 | 0.11 | 1 | 0 | 0 | 0 | 1 |
| Species | Coordinate | ID | Alignment |
|---|---|---|---|
| droVir3 | scaffold_12963:9976171-9976377 + | dvi_11170 | AGAATTATTACTTCAAGAAGAGCGTTGCCTACACGGAGGACCACATCCTGGTGAAGAAACTGTTTCTTTACAATGTGGCAGCCGAGGCAAG-TGAATGGGA------ACTT--TT-GTGTGTGTATGTGTGT------G----TGG---C----TTGTCGTTATTT-----GA---GC----TGTT-CT-C---TTTTCA-GCTGTTGCAGCAGGAGGTGCAAACATATTTCGAGGGCTTTGGCCGTGTGCT--- |
| droMoj3 | scaffold_6500:1316937-1317140 - | AGAACTACTACTTCAAGAGGAGCATCGCCTACACCGAGGATCACATTGTGGTGAGGAAGCTGTTCCTCTACAATGTGGCAGCCGAGGCAAG-TGGGTGTGGCG--------------TTT---------GCTTATGCTTAATATAGTTT----GTG------ACTTGAGCCGT--------TCG-TTCT-C---CTTGCA-GCTGCTGCAGCGGGAGCTGCTGGCGTACTTCGAGGGCTTTGGCCGCGTGCT--- | |
| droGri2 | scaffold_15252:11881051-11881243 - | A--------------AGTAGAACATTGCCTACACGGAGGATCACATTGTGGTAAAGAAATTGTTTCTGCACAATGTGGCAAACGAGGCAGG-TAAATGTGAGGCACAATAT------------------TGGCCAGGCC----CAACTT----GTACTTATTTGGC-----GC--------TGTATTCC-T---TTTGCA-GTTGTTGCCGCAGCAGTTGCAAACATATTTCGAGACTTTCGGCTGTGTTCA--- | |
| droWil2 | scf2_1100000004954:4512415-4512626 + | AAAGTTGTGACTTTAAAAATGACTTGTGCTACACAAAGGACCACATTGTAGTCCAGGACTTATATCTGTACGACGTACCAACAGAGGTAAG-TGCATGACCCAGGAAACGA--TC-CTT----------TTTCAATCCT----TATTTT----GGTTTTAAATGCC-----GA--------TTCGAATG-T---TTTGCA-GCTGAAAGAGGAAGACATGCTTGAGCATTTCAATAGATTTGGAAAAGTCGG--- | |
| dp5 | 3:19267896-19268099 + | AGAACTATTACTTCAAGAATGGTATTGCCTACACAGAAGACCACATTCCAGTGCTGAAACTGTATTTATACAAGATCCCACCGGATGTAAG-TGTCCGCGTCC-------C--CC-TTT----------TGTCAAGCTT----TACTCT----GAAACAATACACT-----GT--------CCT-GTCT-C---ATCGCA-GCTAAATCAATCTACGCTGCATTCGCACTTCTCAAACTATGGACGCGTTTT--- | |
| droPer2 | scaffold_37:270822-271025 + | AGAACTATTACTTCAAGAATGGTATTGCCTACACAGAAGACCACATTCCAGTGCTGAAACTGTATTTATACAAGATCCCACCGGATGTAAG-TGTCCGCGTTC-------C--CC-TTT----------TGTCAAGCTT----TACTCT----GAAACAATACACT-----GT--------CCT-GTCT-C---ATCGCA-GCTAAATCAATCTACGCTGCATTCGCACTTCTCAAACTATGGGCGCGTTTT--- | |
| droAna3 | scaffold_13266:10715779-10715970 + | AAAATTACTATTTCAAGGAATCGATTGCCTACACCGAAGACCATATTCCCGTGAAAAAGATCTATCTGTACAAGGTGCCGCCGGAGGTGCC-AAGACCAC-------------TCG-------------CTC-----------------CCTAGAAATCATCAACT-----GA---TC----ACTCCTCTT---TTTGCA-GTTGAATCAAAATGTCTTGGAGGAGCACTTTAATGTGTACGGTCGTGTGCT--- | |
| droBip1 | scf7180000396428:1144769-1144959 + | AAAATTACTATTTCAAGGAATCGATTGCATACACCGAAGACCACATTCCCGTCCAGAAGCTTTACCTGTATAAAGTGCCAGCGGAGGTGCC-AAGACCAC-------------TTG-------------TTT-----------------CCTAGAAATCATGAATT-----GA---TA----ATTCCTC-T---CTTGCA-GTTAAGTCAAAATGTCTTGGAGCTGCATTTCTGTGGGTACGGCCGAGTAGT--- | |
| droKik1 | scf7180000302476:2865297-2865386 - | TTTTGT------------------------------------------------------------------------------GTGTATG-TGTGTGTGTTT----------------------------------------------TTTG---TTCATTTTTT-----TT--------CGTTT-TT-C---GTTTCAGGCGGCAGCAAAAATT---CCA----------------TTGCGCCGTGTGTTTAT | |
| droFic1 | scf7180000453858:524594-524805 - | AAAACTACTACTTTGAAGACTCTATTGCCTACACAGAAGACCACATTAAAGTGCAGAAACTAAGTTGGTGCGATGCCCCAAGGGAGGTAGCCTTTTTAAGATTG----TCT--G---------------CA----GTGTTATAAAATTC----GTT------TCCTTATCCACTCGTTAAATGATT----TTCTGCAGAG-TTTAAATAAGAGCTCGATACAAGCATACTTTGAAAGCTTTGGGCGCGTGTT--- | |
| droEle1 | scf7180000491211:527152-527357 - | ACAACTGCTACCTCAAGAACTCGATTGTCTACACCGAGGACCACATTCCCGTGCAGAAGCTCTACTTCAGCAAGGTGCCTCCGGAGGTTGCGTCTTTCTGGCC----ACGT--T---------------GTTTGTCCTCTGTGTGC---CTGG---CTAACCCACT-----GT--------CGA-GCCC-T---TTTGCA-GCTGAACCAAAGCATTCTTGAGCATCACTTCCGGATCTATGTGCGCGTGCT--- | |
| droRho1 | scf7180000777716:13622-13816 + | GAAACTACTATTTCAAGGACTCGATTGCCTACACCGAGGATCACATTCCCGTGCAGAAGGTCCACATTTACAATGTTTTGCCGAAGGTAGCGTTTTTCTGGG---------------------------------CCTTTGGGTAC---CTCG---TTAACCCATT-----GT--------TGATTCTC-T---CCAGAA-GTTGAACAAAAAAATGCTTGAGCATTACTTTGGGATCTATGGACGCGTGCT--- | |
| droBia1 | scf7180000301506:2432856-2433056 + | AGAACTACTACTTTAAGGACTCCATTGCCTACACCGAGGACCACATTCCCGTGCAGAAGCTCTACTTTTACAGGGTTCCTCCAGGGGTAGG-TCCTCCTGCTCCTC--------------------------AGCCCTTTACATAC---GCCA---CTAAACCCCT-----GC--------TAATCATC-T---CCTGCA-GCTCAGACAGGCCGATTTAAGGAAGCACTTCGGGATCTACGGGACTGTGCT--- | |
| droTak1 | scf7180000415346:220120-220322 - | AGAATTACTACTTTAAGGACTCGATTGCCTACACCGAGGACCACATTCCCGTCCAGAAGCTCTACTTCTACAAGGTTCCGCCGGAGGTAGCGTGTTTCTGGCCA---ACGC------------------CT----CTTTCGTTTAC---CCCA---CTAATCCCCT-----GC--------CGATCCTC-T---CCTGCA-GCTCAGCCAGGCCATTTTGGAGCAGCACTTTGGGATCTACGGGCACGTGCT--- | |
| droEug1 | scf7180000409663:88449-88644 + | AAAACTATTATATAAAGAACATGATTGCCTATACCGAAGACCACGTTCCCGTACAGAAGCTGTACTTCTACAAGGTTTCGCCGCAGGTACA-TGTACAA----TATAGCACCTTTG-------------TGG-----------------TCCA---CTAATTTTCT-----GT--------TGACCTTC-T---CATGCA-TCTGAATCAGAGCATTCTGGAGCACCACTTTGAGGCCTATGGGCGCGTGCT--- |
| Species | Read alignment | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| droVir3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droMoj3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droGri2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droWil2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| dp5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droPer2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droAna3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBip1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droKik1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droFic1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEle1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droRho1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droBia1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droTak1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| droEug1 |
|
Generated: 05/16/2015 at 08:44 PM